Практикум по алгоритмам и программам множественного выравнивания
Сравнение выравниваний одних и тех же последовательностей разными программами множественного выравнивания
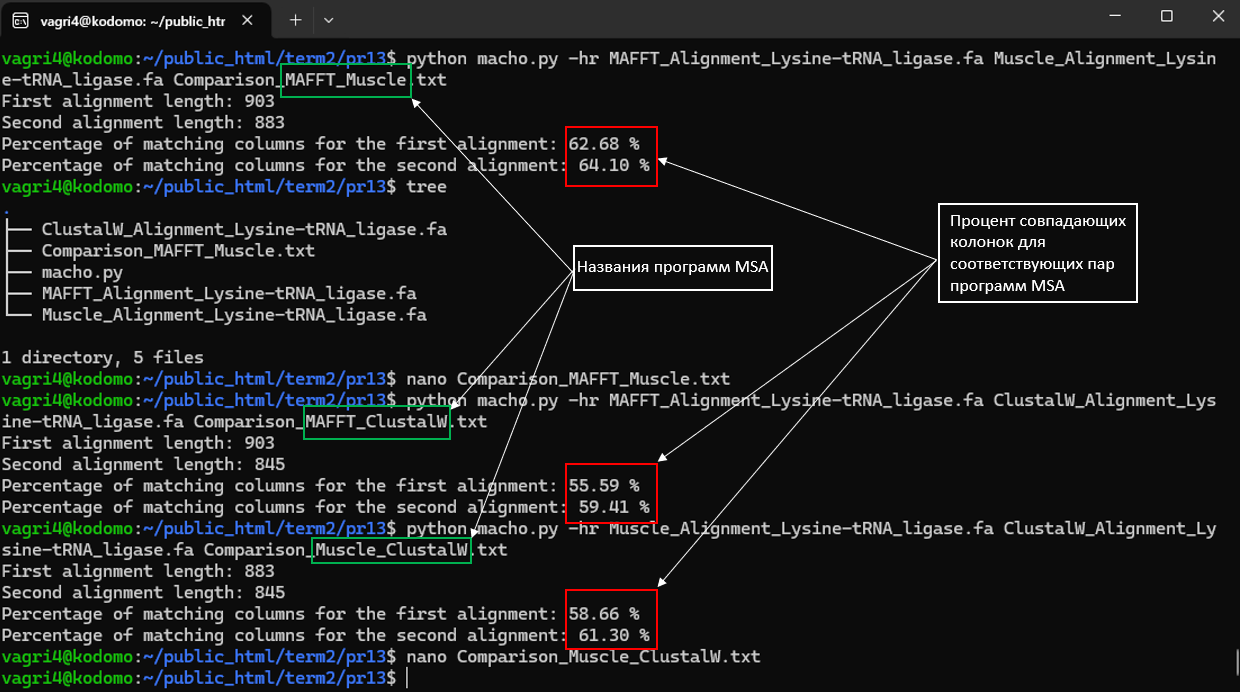
В данном задании необходимо было сравнить характеристики разных программ множественного выравнивания и выяснить, в выравнивании с помощью каких пар программ достигается наибольшее процентное совпадение
выравненных колонок, в каких — наименьшее. Для выполнения задания было проведено три множестенных выравнивания 46 последовательностей белков, содержащих доменную архитектуру PF01336 - PF00152 - PF09391
(репрезентативный белок — Lysine-tRNA ligase, AC=A0A0G0BKH8). Доменная архитектура была взята из базы данных семейств белков доменов Pfam (запрос — PF00152).
Ниже приведены результаты выдачи скрипта macho.py для трёх пар сравниваемых программ множественного выравнивания (см. Рис. 1.), а также координаты совпадающих блоков в выравнивании
(см. Таблицы 1, 2, 3.)
| Блок | MAFFT | Muscle |
|---|---|---|
| 1 | 1-7 | 1-7 |
| 2 | 42-47 | 36-41 |
| 3 | 53-58 | 46-51 |
| 4 | 86-92 | 71-77 |
| 5 | 104-125 | 100-121 |
| 6 | 127-136 | 123-132 |
| 7 | 138-146 | 134-142 |
| 8 | 153-158 | 149-154 |
| 9 | 162-166 | 158-162 |
| 10 | 169-207 | 165-203 |
| 11 | 214-293 | 210-289 |
| 12 | 296-360 | 292-356 |
| 13 | 367-367 | 360-360 |
| 14 | 371-404 | 368-401 |
| 15 | 451-458 | 439-446 |
| 16 | 465-484 | 453-472 |
| 17 | 489-562 | 477-550 |
| 18 | 576-585 | 564-573 |
| 19 | 602-603 | 588-589 |
| 20 | 607-608 | 593-594 |
| 21 | 642-644 | 638-640 |
| 22 | 685-715 | 668-698 |
| 23 | 717-719 | 700-702 |
| 24 | 730-749 | 709-728 |
| 25 | 754-754 | 733-733 |
| Блок | Muscle | ClustalW |
|---|---|---|
| 1 | 1-11 | 1-11 |
| 2 | 34-49 | 34-49 |
| 3 | 99-121 | 89-111 |
| 4 | 123-132 | 113-122 |
| 5 | 134-142 | 124-132 |
| 6 | 165-203 | 154-192 |
| 7 | 211-247 | 200-236 |
| 8 | 253-357 | 242-346 |
| 9 | 368-383 | 355-370 |
| 10 | 386-403 | 373-390 |
| 11 | 453-472 | 438-457 |
| 12 | 482-549 | 467-534 |
| 13 | 563-575 | 548-560 |
| 14 | 668-699 | 632-663 |
| 15 | 714-728 | 677-691 |
| 16 | 732-732 | 695-695 |
| 17 | 751-751 | 713-713 |
| 18 | 757-766 | 719-728 |
| 19 | 796-817 | 758-779 |
| 20 | 825-851 | 787-813 |
| 21 | 855-875 | 817-837 |
| 22 | 880-883 | 842-845 |
| Блок | MAFFT | ClustalW |
|---|---|---|
| 1 | 1-7 | 1-7 |
| 2 | 42-47 | 36-41 |
| 3 | 53-56 | 46-49 |
| 4 | 104-146 | 90-132 |
| 5 | 169-208 | 154-193 |
| 6 | 212-212 | 197-197 |
| 7 | 215-251 | 200-236 |
| 8 | 257-293 | 242-278 |
| 9 | 296-360 | 281-345 |
| 10 | 368-386 | 352-370 |
| 11 | 389-404 | 373-388 |
| 12 | 465-484 | 438-457 |
| 13 | 494-561 | 467-534 |
| 14 | 576-585 | 549-558 |
| 15 | 682-715 | 629-662 |
| 16 | 735-749 | 677-691 |
| 17 | 771-771 | 713-713 |
| 18 | 777-786 | 719-728 |
| 19 | 813-835 | 758-780 |
| 20 | 842-868 | 787-813 |
| 21 | 872-889 | 817-834 |
| 22 | 903-903 | 845-845 |
Ссылка на проект Jalview. В проекте три файла, в каждом файле — выравнивание с помощью соответствующей программы. Колонки окрашены с помощью Clustal.
На основании данных, полученных в результате проведения множественного выравнивания тремя различными программами, можно сделать вывод о том, что программы MAFFT и Muscle выравнивают последовательности более схожим образом (что и неудивительно, ведь алгоритм их работы основан на итеративном рафинировании), по сравнению с менее совершенной программой ClustalW (алгоритм которой основан на прогрессивном выравнивании без итеративного рафинирования). Это нетрудно заметить по проценту совпадающих колонок выравниваний (см. Рис. 1.), где у выравниваний с помощью MAFFT и Muscle наблюдается наибольшее совпадение. Также это хорошо видно по числу совпадающих блоков выравниваний (см. Таблицы 1, 2, 3), где у выравниваний с помощью MAFFT и Muscle наблюдается наибольшее количество таких блоков.
Построение выравнивания по совмещению структур и его сравнение с выравниванием MSA
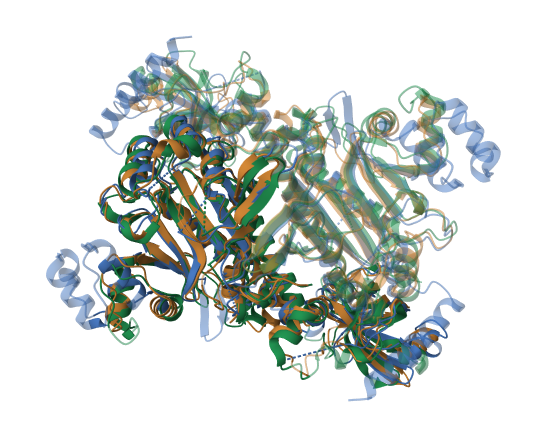
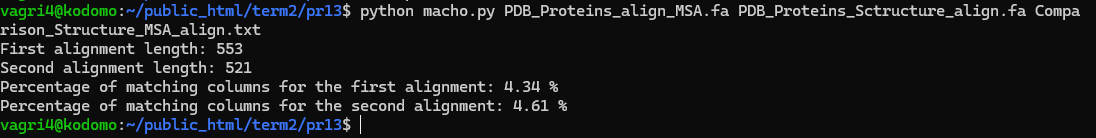
Выравнивание по совмещению структур проводилось с помощью соответствующего ресурса на сайте PDB. Выравнивание MSA проводилось с помощью программы множественного выравнивания последовательностей MAFFT. Ниже приведены рисунок совмещения пространственных структур заданных белков (см. Рис. 2.), выдача скрипта macho.py для двух данных выравниваний (см. Рис. 3.), координаты совпадающих блоков в выравниваниях (см. Таблицу 4) и ссылка на проект Jalview с двумя выравниваниями (пространственное и с помощью программы MSA).
| Блок | Структурное выравнивание | MAFFT |
|---|---|---|
| 1 | 23-34 | 13-24 |
| 2 | 49-50 | 39-40 |
| 3 | 53-54 | 43-44 |
| 4 | 56-59 | 46-49 |
| 5 | 316-316 | 289-289 |
| 6 | 372-372 | 370-370 |
| 7 | 374-375 | 372-373 |
Проанализировав полученные данные, нетрудно заметить, что процент совпадающих колонок для выравнивания с помощью совмещеня пространственных структур белков и с помощью программы множественного выравнивания MAFFT очень небольшой (см. Рис.3.) — всего 4.34 и 4.61 процента для соответствующих выравниваний. Такое сильно несовпадение выравниваний объясняется тем, что в ходе эволюции белки могут накапливать большое количество мутаций, что можно обнаружить по множественному выравниванию с помощью какой-нибудь программы MSA, однако, сохраняя при этом высокую консервативность структур, что хорошо подтверждается рассматриваемым примером (см. Рис. 2.).
Ссылка на проект Jalview с выравниванием, полученным совмещением пространственных структур, и выравниванием, полученным программой MAFFT.
Описание программы множественного выравнивания MAFFT
MAFFT (multiple alignment using fast Fourier transform) — это программа, используемая для создания множественных выравниваний аминокислотных или нуклеотидных последовательностей [1].
Опубликованная в 2002 году, первая версия использовала алгоритм, основанный на прогрессивном выравнивании, в котором последовательности группировались с помощью быстрого преобразования Фурье [2].
Последующие версии MAFFT добавили другие алгоритмы и режимы работы, включая опции для более быстрого выравнивания большого числа последовательностей, выравнивания с более высокой точностью,
выравнивания некодирующих последовательностей РНК [3] и добавления новых последовательностей к существующим выравниваниям [4].
Принцип работы программы основывается на 5 следующих шагах [5]:
1. Парное выравнивание. На данном шаге производится попарное выравнивание всех заданных последовательностей. Этот шаг нужен для того, чтобы определить, какие участки последовательностей имеют наибольшее сходство.
2. Расчёт матрицы расстояний последовательностей. На этом этапе производится построение матрицы расстояний, отражающей степень сходства между разными парами заданных последовательностей.
3. Построение направляющего дерева. Направляющее дерево строится на основе матрицы расстояний, посчитанной на предыдущем шаге. В листьях такого дерева оказываются пары последовательностей, наиболее
схожих между собой.
4. Прогрессивное выравнивание. На этом шаге производится собственно множественное выравнивание последовательностей от "листьев" направляющего дерева к "корню" — то есть от наиболее схожих (родственных)
между собой последовательностей к наименее схожим.
5. Итеративное рафинирование. На конечном этапе происходит итеративное разделение и перевыравнивание каждой из двух подгрупп исходной группы последовательностей для "очищения" (рафинирования) результата прогрессивного выравнивания.
Литература
1. https://en.wikipedia.org/wiki/MAFFT?ysclid=max2ptu562799609601
2. Katoh, Kazutaka; Misawa, Kazuharu; Kuma, Kei-ichi; Miyata, Takashi (2002). "MAFFT: a novel method for rapid multiple sequence alignment based on fast Fourier transform". Nucleic Acids Research. 30 (14): 3059–66.
3. Katoh, Kazutaka; Toh, Hiroyuki (2008). "Improved accuracy of multiple ncRNA alignment by incorporating structural information into a MAFFT-based framework". BMC Bioinformatics. 9: 212. doi:10.1186/1471-2105-9-212. PMC 2387179. PMID 18439255
4. Katoh, K.; Standley, D. M. (April 2013). "MAFFT Multiple Sequence Alignment Software Version 7: Improvements in Performance and Usability". Molecular Biology and Evolution. 30 (4): 772–780. doi:10.1093/molbev/mst010. PMC 3603318. PMID 23329690
5. The base MAFFT software is released under one of the BSD licenses, while versions for Microsoft Windows are released under a GNU General Public License. Some distributions of MAFFT contain software licensed under other licenses https://mafft.cbrc.jp/alignment/software/