Практикум по работе в базах данных последовательностей белков UniProtKB
1. Выбор белка
Для выбора подходящего для выполнения заданий белка был использован продвинутый поиск (Advanced search) с указанием видового названия организма (Natronomonas pharaonis (strain DSM 2160 Gabara)), а также названия класса ферментов, к которому относится данный белок (Ligase). Всего таких запросов было найдено 59 (в базе данных TrEMBL), однако были отобраны только записи, содержащиеся также и в Swiss-Prot (28 записей). Первый же встретившийся белок показался весьма интересным с точки зрения своей функции (CTP sythase), а также хорошо аннотированным, поэтому и был выбран для дальнейшего анализа.
2. Информация о белке
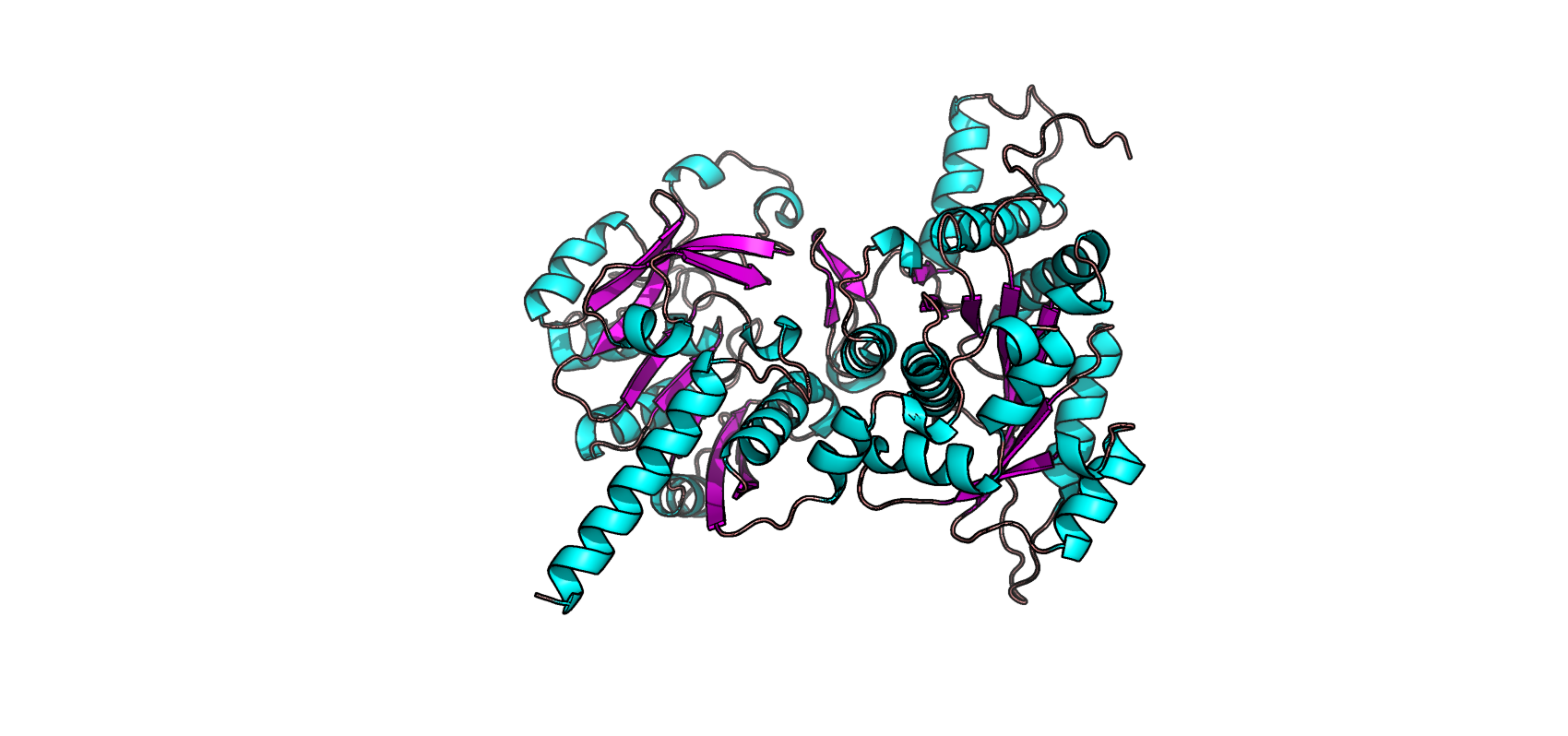
Как уже было упомянуто ранее, для анализа был выбран белок ЦТФ-синтаза. По названию данного белка очевидно, что его функция заключается в синтезе ЦТФ (Цитидинтрифосфат) с затратой энергии АТФ (ибо фермент относится к классу лигаз, EC=6.3.4.2). Если говорить более точно, данный фермент осуществляет реакцию АТФ-зависимого аминирования УТФ L-глутамином или аммонием с образованием ЦТФ. Из интересных данных, встретившихся в аннотации последовательности, можно отметить, что этот фермент имеет сайты связывания иона магния (что типично для лигаз).
3. Кластеры похожих белков
Для данного фермента известно, что размер кластеров UniRef100 и UniRef90 состоит всего лишь из одного белка (собственно самой ЦТФ-синтазы), в то время как в состав кластера UniRef50 входит целых 1010 белков. Это говорит о том, что данный белок является довольно нераспространённым (в каком-то смысле уникальным), а также неконсервативным, так как количество белков, последовательности которых совпадают на 50 процентов (UniRef50) значительно превышает количество белков, совпадающих по последовательности с исходным ферментом на 90 и 100 процентов (UniRef90 и UniRef100, соответственно).
4. Поисковые запросы
Первый запрос был составлен с помощью продвинутого поиска с заполнением полей Protein Name [DE] (CTP syntase) и Taxonomy [OC] (Natronomonas). Идея заключалась в том, чтобы оценить распространёность рассматриваемого белка в пределах одного рода архей. По итогам поиска выяснилось, что только у Natronomonas pharaonis имеется аннотированная ЦТФ-синтаза в базе Swiss-Prot, а для остальных 4-х видов на данный момент записи существуют только в TrEMBL. Аналогично, для всех архей (Taxonomy [OC]: Archaea) было выявлено, что этот фермент представлен лишь 39 аннотированными в базе Swiss-Prot записями, что говорит о весьма низкой распространнёности белка как в пределах рода, так и в пределах целого домена. Результаты поиска по роду Natronomonas и по домену Archaea.
Второй же запрос был составлен путём заполнения полей Enzyme Classification [EC] (6.3.4.2), Protein Existence [PE] (Evidence at protein level) и Taxonomy [OC] (Archaea). По результатам запроса была найдена лишь одна запись (в организме Saccharolobus solfataricus). Запрос составлялся с целью выяснить, с какой точностью можно судить о достоверности существования изучаемого фермента среди архей. Как выяснилось, подавляющее большинство записей белка имеют PE 3 (inferred from homology). Cсылка на соответствующий запрос. Поэтому резонно предположить, что рассматриваемый белок, как и большинство белков в Swiss-Prot, предсказанных на основе гомологии, имеет несколько спорную достоверность существования, несмотря на то, что все записи, содержащиейся в Swiss-Prot, проверяются и подтверждаются кураторами.
Третий запрос составлялся с целью выявить, какие лигазы среди класса архей Halobacteria встречаются чаще — те, которые связывают катион магния (как и семейство ЦТФ-синтаз), или же те, которые связывают какой-то другой катион. Сначала были заполнены поля Taxonomy [OC] (Halobacteria), Keyword [KW] (Ligase); затем было добавлено ещё одно поле Keyword [KW] (Magnesium) с целью оценить количество записей магний-связывающих лигаз относительно их общего числа в пределах рассматриваемого класса. По первому запросу было найдено 180 записей, по второму же — только 65 (рассматриваются только записи в Swiss-Prot как наиболее достоверные). Из полученных результатов можно сделать вывод о том, что семейство магний-связывающих лигаз, к которым относится и рассматриваемый белок, представлено довольно скудно (65 записей из 180) относительно общего количества лигаз в пределах рассматриваемого класса архей. Следовательно, и само семейство ЦТФ-синтаз весьма бедно представлено среди этого класса.
5. Поиск источника аннотации
ECO (Evidence and Conclusion Ontology) — это онтология стандартизованных типов доказательств, которые используются в аннотациях белковых последовательностей. Эти коды позволяют понять, на каком основании был сделан тот или иной вывод о различных особенностях белка. В изучаемом же белке во всех разделах аннотации, требующих подтверждения данных, написан один и тот же ECO код, а именно ECO:0000255 (так как код везде одинаковый, то принципиально неважно, какие факты о белке выбирать, формально выберем [SUBUNIT: Homotetramer], [FUNCTION: Catalyzes the ATP-dependent amination of UTP to CTP with either L-glutamine or ammonia as the source of nitrogen. Regulates intracellular CTP levels through interactions with the four ribonucleotide triphosphates] и [SIMILARITY: Belongs to the CTP synthase family]). Данный код означает, что доказательства были сгенерированы системой автоматической аннотации на основе соответствия последовательностной модели (Sequence model evidence). Также указывается база данных и идентификатор в ней (в данном случае HAMAP-Rule:MF_01227), модель которой была использована данной системой. Однако, как указано на самой странице белка в разделе Help, эти доказательства, хоть и были сгенерированы автоматически, в дальнейшем были проверены и подтверждены кураторами, следовательно, таким ECO кодам можно доверять.