Практикум по работе с UniProt Proteomes и EMBOSS
Поиск протеома, соответствующего геномной сборке
В данном практикуме будут рассмотрены основные методы анализа протеомов при помощи базы данных UniProt Proteomes и пакета биоинформатических программ EMBOSS (The European Molecular Biology Open Software Suite)
Ниже перечислены индентификаторы геномной сборки и протеома Natronomonas pharaonis, статус протеома, а также ссылки на соответствующие страницы из баз данных.
1. Идентификатор сборки RefSeq: GCF_000026045.1;
2. Ссылка на страницу из базы NCBI Datasets Genome, которая соответствует этой сборке;
3. Идентификатор последней версии сборки INSDC: GCA_000026045.1;
4. Поисковый запрос по UniProt Proteomes, который выдал протеом: (genome_assembly:GCA_000026045.1). Cсылка на результат запроса;
5. Идентификатор протеома: UP000002698;
6. Статус протеома: референсный (Status: Reference proteome).
Поиск и скачивание референсного протеома
Так как протеом, соответствующий геномной сборке рассматриваемой археи, является референсным, то нет необходимости продолжать поиск протеомов из того же таксона или таксонов
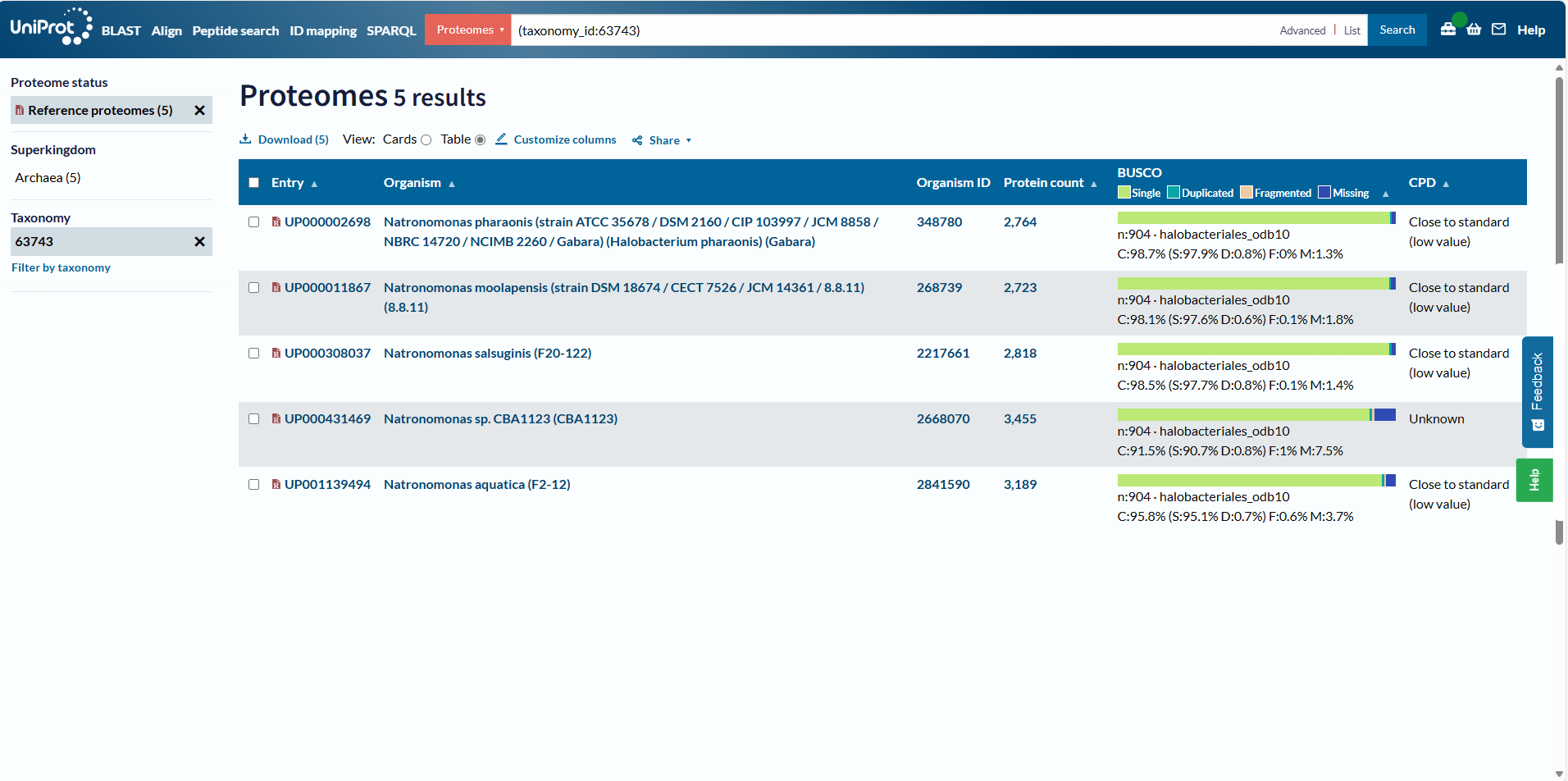
более высокого ранга. Однако с целью удостовериться в этом был произвёден поиск референсных протеомов (Рис.1.) в пределах рода Natronomonas (taxonomy_id:63743). В результате поиска
было найдено 5 референсных протеомов из видов того же рода, включая и сам вид Natronomonas pharaonis (strain DSM 2160 Gabara). Идентификатор протеома: UP000002698. Полная команда в bash,
использовавшаяся для скачивания протеома: wget 'https://rest.uniprot.org/uniprotkb/stream?compressed=true&format=txt&query=proteome%3AUP000002698' -O UP000002698.swiss.gz
Оценка количества ферментов в протеоме
Для оценки количества ферментов в протеоме рассматриваемой археи Natronomonas pharaonis (strain DSM2160 Gabara) был использован следующий поисковый запрос: (organism_id:348780) AND (proteome:UP000002698) AND (ec:*). В результате был выявлен 801 белок, который обладает ферментативной активностью, ибо EC-коды указываются только лишь в аннотациях подобных белков. Ссылка на результат запроса.
Далее был написан конвейер на bash, который аналогично позволил выявить белки, обладающие ферментативной активностью, по наличию у них EC-кода: zgrep '^DE' term2/pr8/UP000002698.swiss.gz | zgrep -c 'EC='. Эта команда выявляет 829 ферментов, что немного больше того, что удалось оценить с помощью UniProtKB. Несовпадение результатов оценки, вероятно, объясняется тем, что некоторые ферменты из данного протеома могут обладать несколькими ферментативными активностями (например, Probable bifunctional tRNA threonylcarbamoyladenosine biosynthesis protein, ID: KAE1B_NATPD), и для каждого домена со своей активностью будет и свой EC-код. Конвейер, написанный на bash, не учитывает этого, а потому может один фермент "посчитать" дважды, в отличие от поиска в UniProtKB.
Анализ протеома консольными средствами
Для анализа протеома с помощью командной строки bash был написан скрипт, вычисляющий частоту встречаемости каждой протеиногенной аминокислоты в протеоме археи Natronomonas pharaonis. В скрипт входят 3 команды:
1. seqret -filter 'sw:*_NATPD' 'Proteomes.fasta' — с помощью этой команды все белки, входящие в протеом исследуемой археи и имеющие унифицированный ID, записываются в файл с форматом fasta.
2. seqret -filter 'Proteomes.fasta' 'plain::Proteomes.plain' — эта команда меняет формат файла с fasta на plain (не содержит краткой аннотации в начале последовательностей).
3. wordcount 'Proteomes.plain' -wordsize 1 — данная команда позволяет рассчитать, сколько раз каждая аминокислота встретилась в протеоме.
На выходе мы получаем файл proteomes.wordcount, в котором напротив каждой аминокислоты указано её количество в протеоме. Ссылка на файл. По результатам анализа нетрудно заметить, что одними из наиболее часто встречающихся аминокислот являются отрицательно заряженные глутамат (E, 7885) и аспартат (D, 7469). Как было ранее упомянуто в мини-обзоре, такое высокое содержание отрицательно заряженных аминокислот не случайно, ибо изучаемая архея является экстремофилом, а точнее, галоалкалифилом, то есть живёт в среде с повышенным содержанием солей и очень высоким значением pH (до 11). Так что повышенное содержание глутамата и аспартата является адаптацией к условиям окружающей среды и позволяет этой архее выживать в столь суровых и экстремальных биотопах.