Нуклеотидный BLAST
В этом практикуме предлагалось попробовать различные алгоритмы поиска BLAST через веб-интерфейс на сайте и локально через командную строку.Поиск в геноме эукариота гена, кодирующего δ-субъединицу АТФ-синтазы
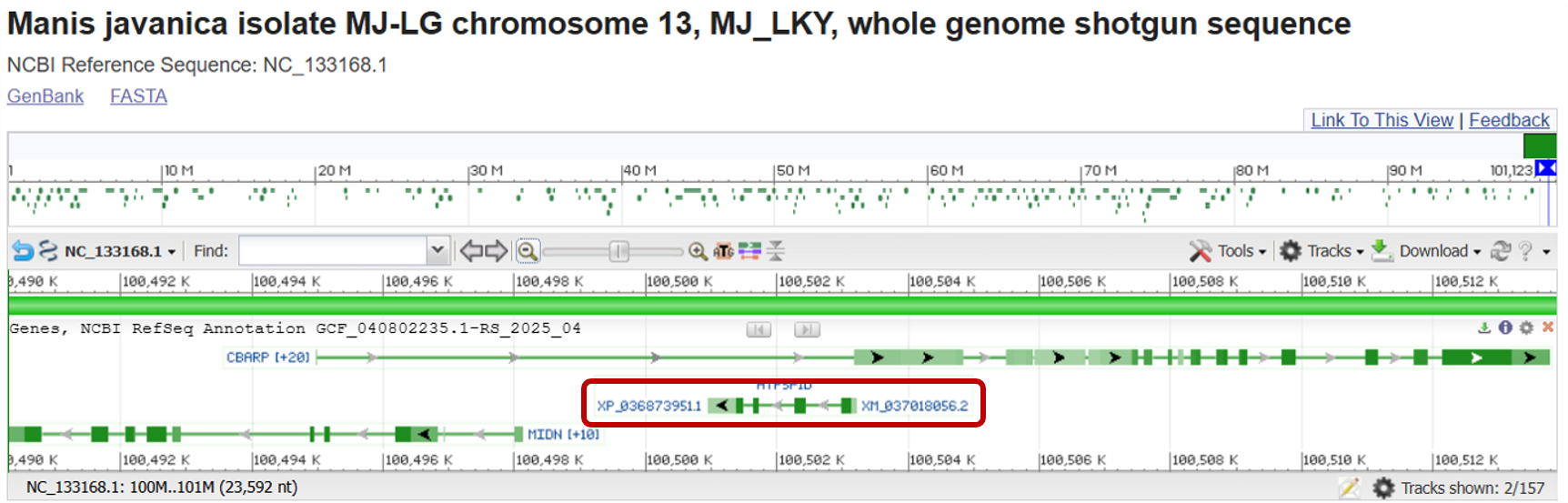
Изначально необходимо было найти последовательность белка дельта-субъединицы АТФ-синтазы выбранного в прошлом практикуме эукариота (Manis javanica) и сохранить в отдельный FASTA-файл. Затем по идентификатору белка проводился поиск идентификатора нуклеотидной записи, к которой относится ген, кодирующий данный белок. Далее с помощью NCBI Nucleotide была получена последовательность ДНК, содержащая белок-кодирующую часть гена исследуемого белка с небольшой окрестностью вокруг. Ниже приведено изображение окрестности этого участка из геномного браузера (см. Рис. 1.). Все необходимые файлы и данные также приведены ниже.
1. Ссылка на последовательность белка дельта-субъединицы АТФ-синтазы эукариота;
2. Идентификатор нуклеотидной записи: NC_133168.1;
3. Ссылка на последовательность белок-кодирующего участка ДНК
Применение различных алгоритмов BLAST для фрагмента ДНК
Дальнеший поиск BLAST проводился в семействе Apoidea (пчёлы) — достаточно удалённом таксоне от исходного организма.

Поиск в геноме Manis javanica генов основных рибосомальных РНК по далёкому гомологу
Сначала было необходимо проиндексировать последовательность генома своего эукариотического организма для работы с локальным BLAST. Индексация последовательности
проводилась с помощью следующей команды:
Затем проводился локальный поиск BLAST с помощью алгоритма blastn, предназначенного для поиска гомологичных
нуклеотидных последовательностей, для каждой из последовательностей рРНК E.Coli по полученной базе данных. В результате
был получен файл с результатами поиска по каждой (16S и 23S) рРНК бактерии. Команды, которые использовались для поиска и выдачи результатов в формате
"Tabular with comment lines":
blastn -task blastn -query 16S_rRNA_ecoli.txt -db ncbi_dataset_MJ_LKY\ncbi_dataset\data\GCF_040802235.1\GCF_040802235.1_MJ_LKY_genomic.fna -out 16S_rRNA_BLASTN.fna -outfmt 7
Ниже указаны ссылки на файлы с результатами поиска по каждой из двух рРНК:
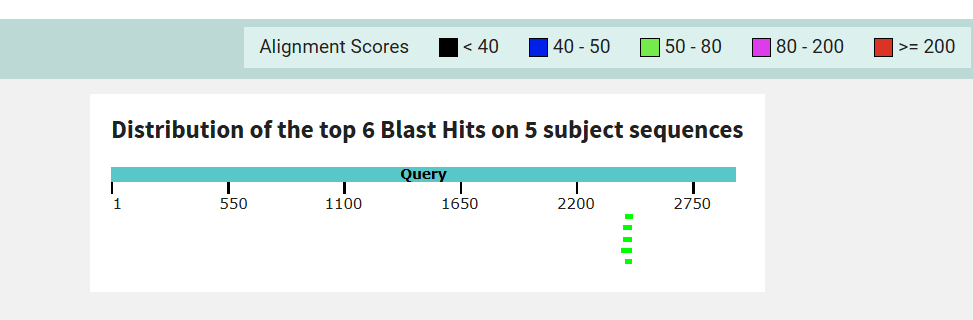
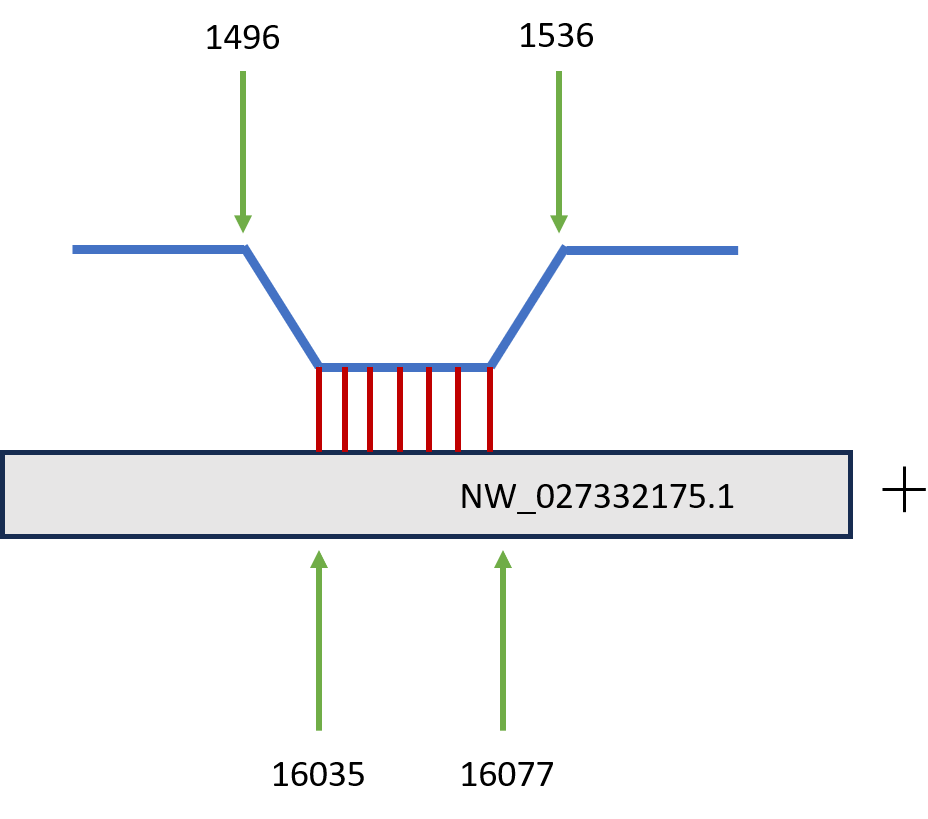
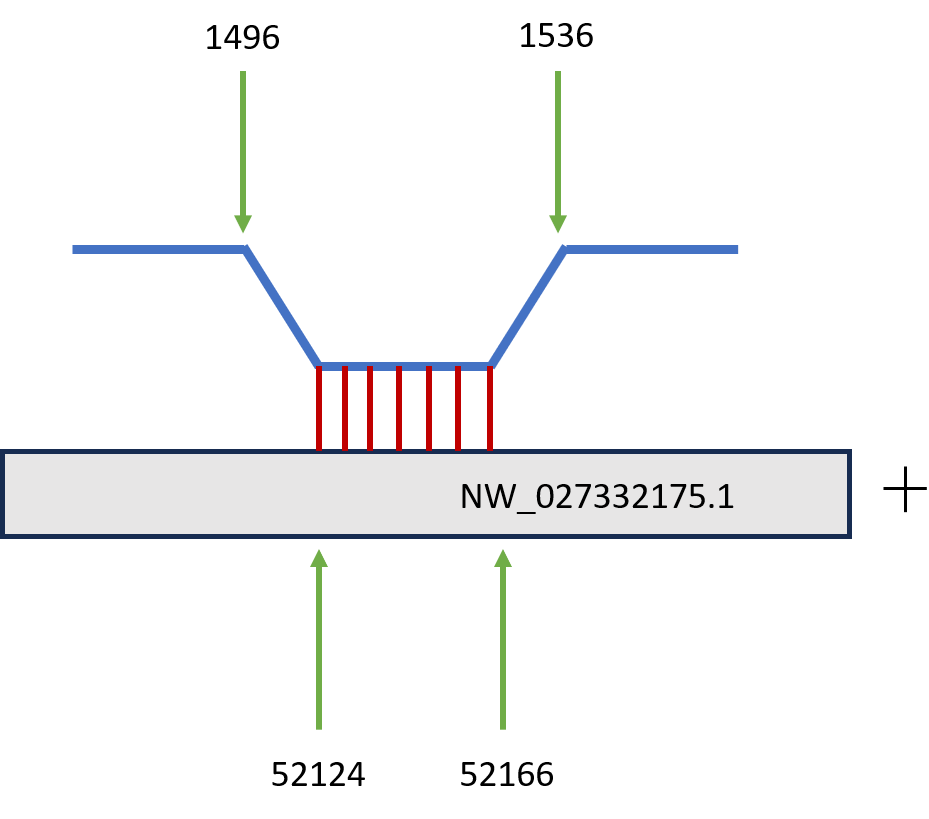
В результате локального поиска BLAST с помощью алгоритма blastn для 16S рРНК было найдено 22 хита, из которых только 6 имели значение E-value меньше 0.05; из них было выделено 6 гомологов, соответствующих участку 1496-1536, исходя из координат выровненных участков: на скэффолде NW_027332175.1 было найдено 4 гомолога, на скэффолдах NC_026781.1 и NC_133159.1 было обнаружено по одному гомологу. Для 23S рРНК было найдено 47 хитов, из которых лишь 28 содержает значение E-value меньше 0.05. Из них можно выделить 12 гомологов (на скэффолде NW_027332175.1 4 гомолога, на скэффолдах NC_133166.1, NC_133164.1, NC_133161.1, NC_026781.1, NC_133174.1, NC_133160.1, NC_133159.1, NC_133157.1 по одному гомологу). Ниже приведены схематичные результаты выравнивания для всех шести гомологов, найденных на 16S рРНК.
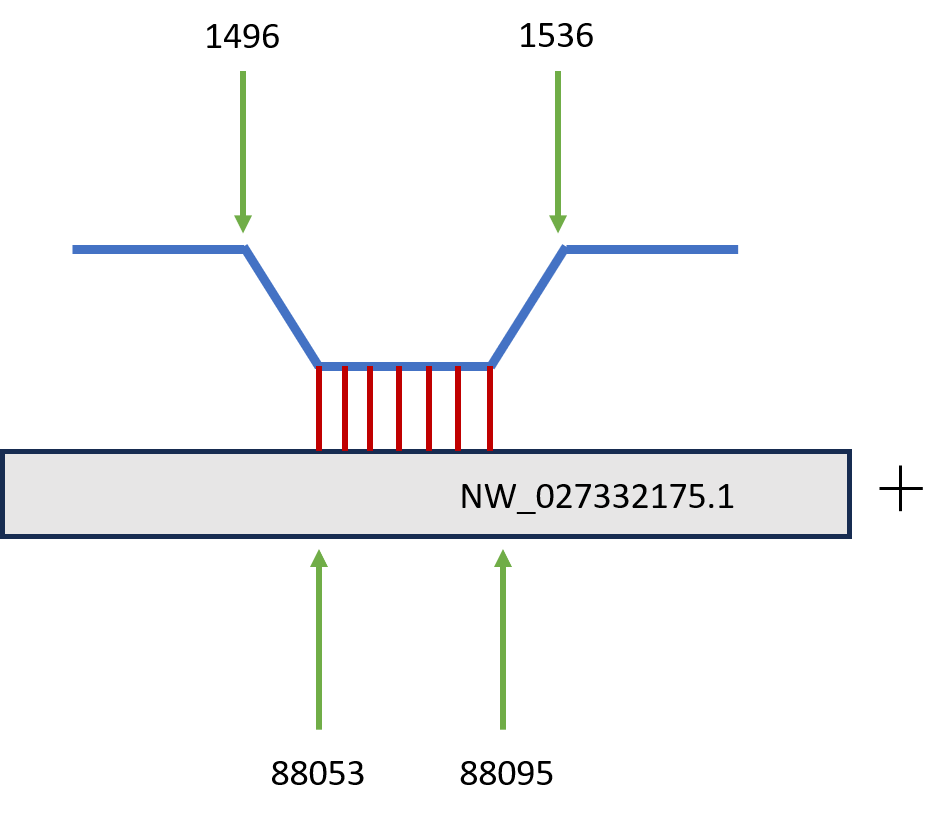
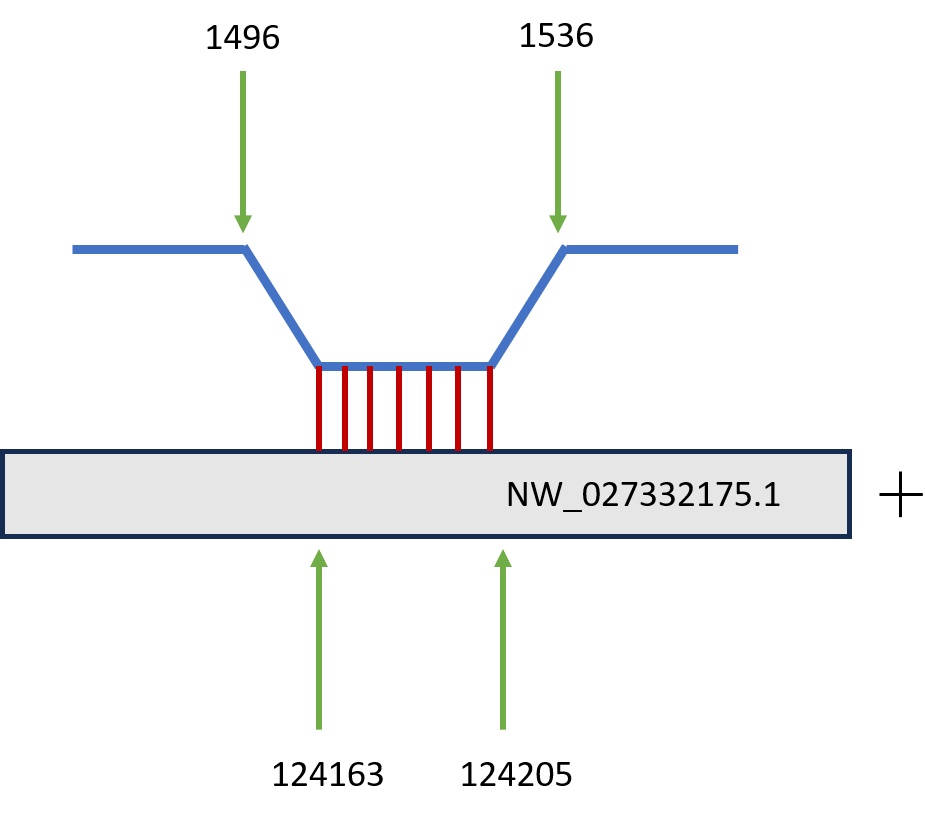
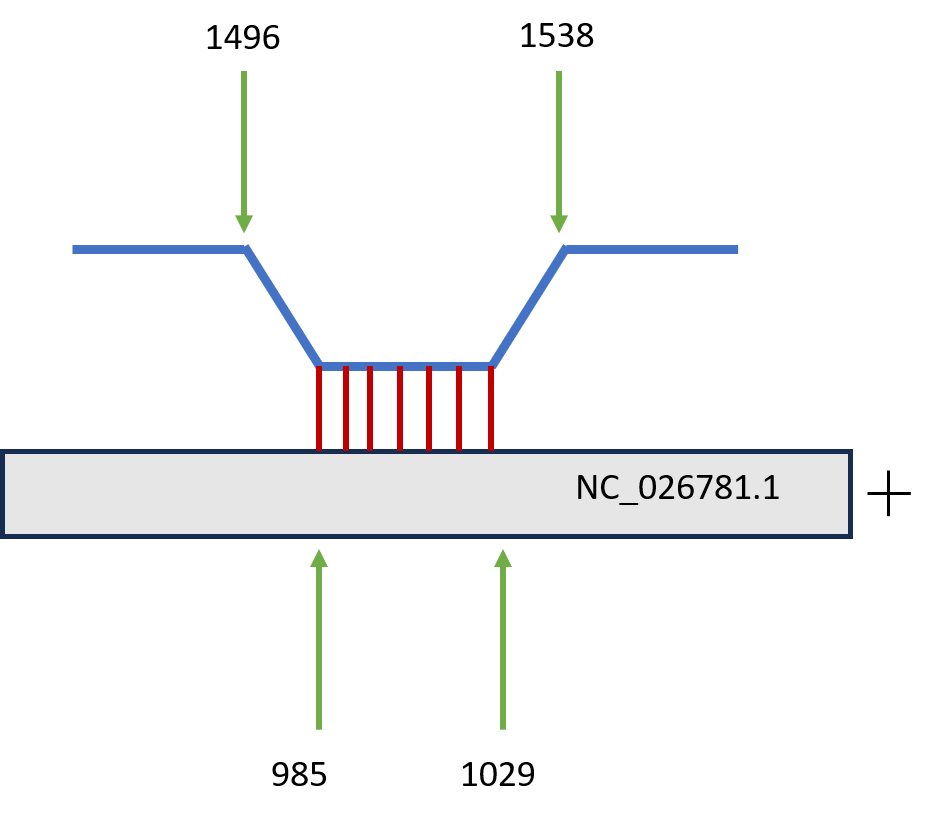
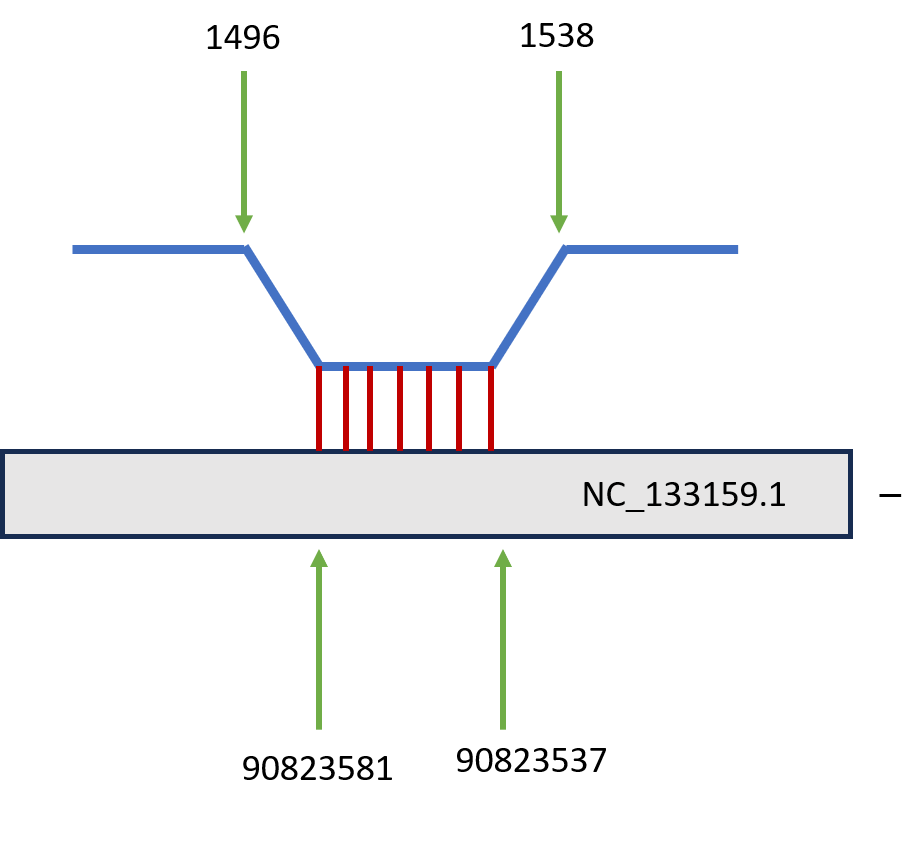
Построение карты локального сходства участков двух геномов
Для построения карты локального сходства были подобраны две геномные последовательности, для которых дотплоты, построенные с помощью алгоритмов megablast, blastn и tblastx заметно различаются в связи с разными принципами работы этих алгоритмов. Были выбраны хромосомы двух штаммов бактерии Paraclostridium sordellii: ATCC 9714 (AC — NZ_LN679998.1) и C125 (AC — NZ_CP124287.1). Данные последовательности были найдены при помощи поиска в NCBI Genome. Организмы подбирались таким образом, чтобы оба имели хромосомный и выше уровень сборки генома, имели различные элементы генома (хромосомы и плазмиды), а также являлись бы представителями разных штаммов одного и того же вида бактерий, для более достоверной интерпретации результатов. Ниже приведены карты для хромосомы двух разных штаммов бактерии Paraclostridium sordellii (см. Рис. 10, 11.), построенные с помощью megablast и blastn, так как алгоритм tblastx не укладывается во временные рамки вычислений из-за размера участка поиска (размер хромосомы — 3.4 Mb), а также отдельно карты, построенные для плазмид тех же штаммов с помощью всех трёх алгоритмов (см. Рис. 12, 13, 14).
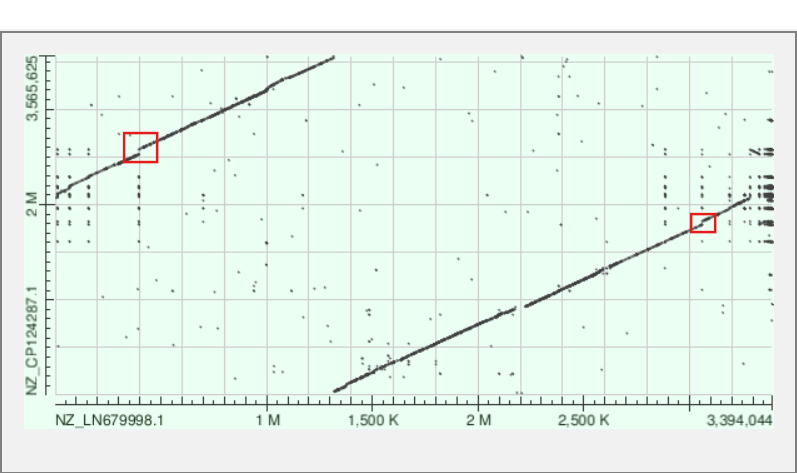
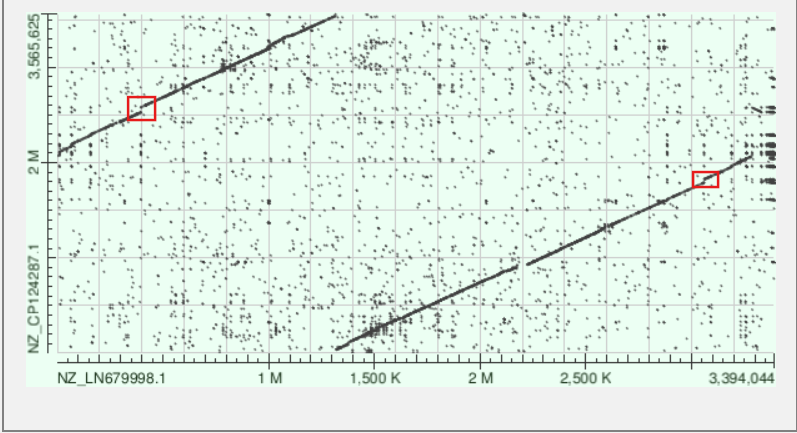
Такая картина на картах объясняется разным выбором точки начала кольцевой нуклеотидной последовательности. Также нетрудно заметить, что алгоритм blastn более точно выявляет повторы в нуклеотидных последовательностях (точки на карте). В красные рамки обведены небольшие индели (инсерции/делеции).
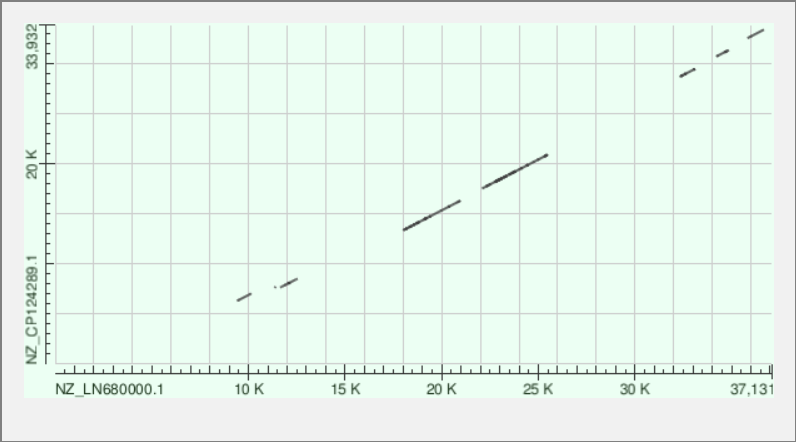
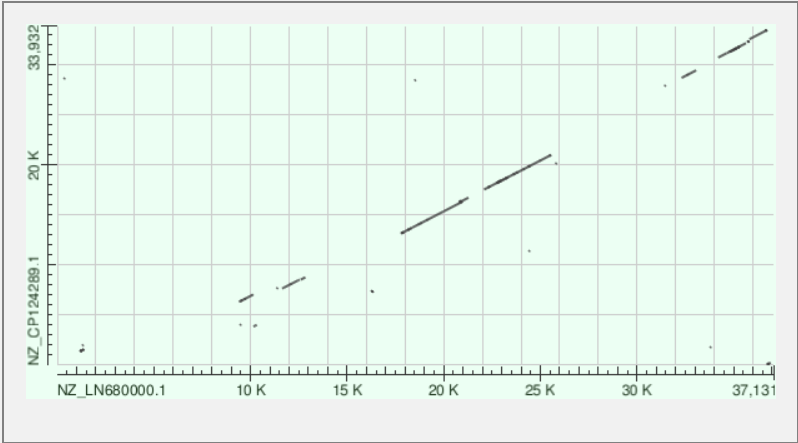
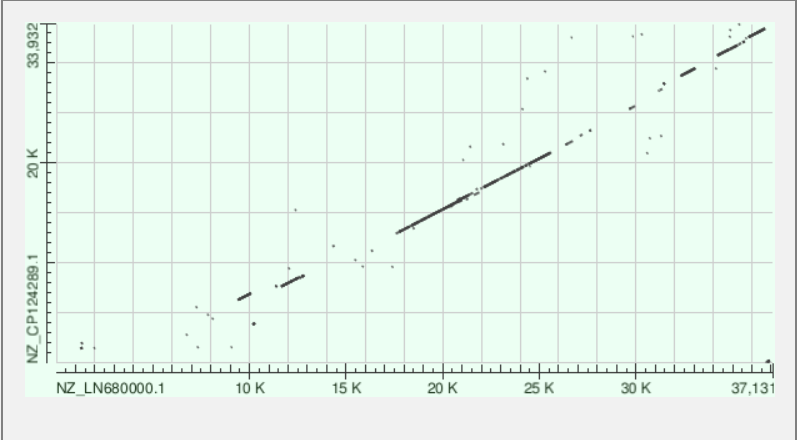
На картах локального сходства плазмиды мы видим, что поиск с помощью алгоритмов blastn и tblastx позволил определить более протяжённые участки гомологии нуклеотидных последовательностей, а также большее число коротких повторов в геноме.
Глобальных перестроек между последовательностями плазмиды не наблюдается.