BLAST
1. Поиск гипотетических гомологов изучаемого белка в разных банках
Результаты поиска представлены в таблице 1.
Таблица 1. Результаты поиска гипотетических гомологов белка PDXK_BACSU
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
1. Лучшая находка (с последовательностью исходного белка) |
|||
| Accession | P39610 | 25IB | NP_391681 |
| E-value | 0.0 | 0.0 | 0.0 |
| Вес (в битах) | 556 | 556 | 556 |
| Процент идентичности | 100 | 100 | 100 |
2. Число находок с E-value < 10–10 |
82 | 17 | 6260 |
3. "Худшая из удовлетворительных" находка (последняя в выдаче с E-value < 1) |
|||
| Номер находки в списке описаний | 101 | 18 | 6819 |
| Accession | A7GZF6 | 3PZS | ZP_11181844 |
| E-value | 0.53 | 0.14 | 0.99 |
| Вес (в битах) | 35.0 | 33.5 | 39.3 |
| % идентичности | 32 | 28 | 39 |
| % сходства | 48 | 50 | 58 |
| Длина выравнивания | 105 | 72 | 70 |
| Координаты выравнивания (от-до, в запросе и в находке) | Query 135-239; Sbjct 186-283 |
Query 106-177; Sbjct 113-183 |
Query 114-182; Sbjct 157-225 |
| Число гэпов | 7 | 1 | 2 |
Исходный белок удалось найти во всех базах данных.
Минимальное число гомологов было найдено в базе PDB. Это связано с тем, что по некоторым причинам белковых последовательноей известно больше, чем 3D структур. По числу гомологов лидирует rn (Non-redundant protein sequences), так он включает в себя все белковые последовательности из всевозможных источников.
В случае SwissProt и rn при стандартных настройках ограничителем было число находок. В PDB количество находок было ограничено предельным E-value (10).
2. Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам
Поиск гомологов проводился в SwissProt по следующим таксонам:
- Eukaryota
- Actinobacteria
- Clostridia
- Lactobacillales
- Listeriaceae
- Geobacillus
- Bacillus anthracis
Результат поиска можно увидеть в таблице 2.
Таблица 2. Результаты поиска гомологов белка PDXK_BACSU с фильтром по таксонам
| BLOSUM62 | BLOSUM45 | |
| Accession | O48881 | O48881 |
| E-value | 8e-48 | 2e-51 |
| Вес (в битах) | 171 | 177 |
| % идентичности | 38 | 38 |
| % сходства | 57 | 59 |
| Длина выравнивания | 246 | 246 |
| Координаты выравнивания (от-до, в запросе и в находке) | Query 5-250; Sbjct 34-277 | Query 5-250; Sbjct 34-277 |
| Число гэпов | 2 | 2 |
3. BLAST двух последовательностей
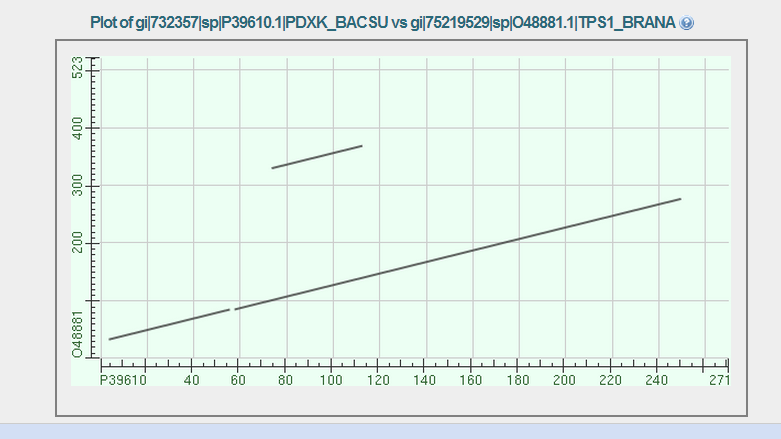
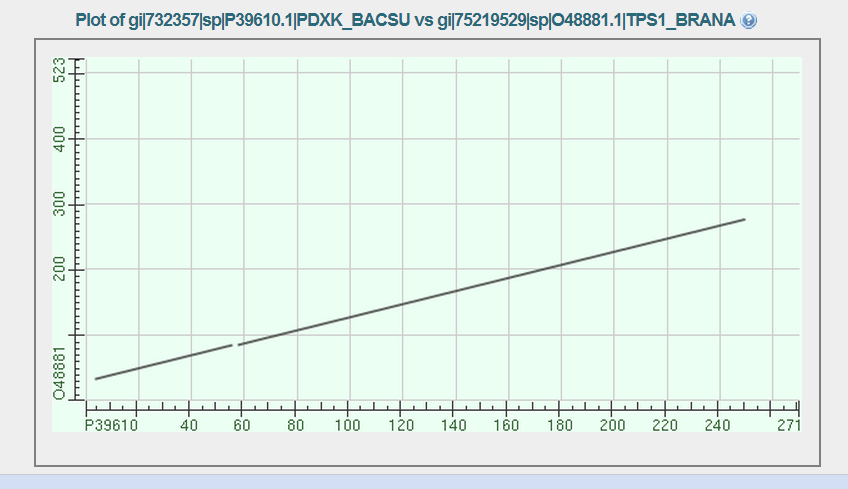
С помощью опции “Align two or more sequences” выполнено парное выравнивание белка P39610 и его гомолога O48881. На рисунке 1 представлена карта локального сходства со стандартным порогом E-value=10, на рисунке 2 – с порогом E-value=0.01. На втором рисунке отсутствует часть графика. Видимо, процент идентичности этого выравнивания выравнивания меньше, а следовательно E-value больше указанного порога.
4. Сравнение результатов поиска с различными матрицами BLOSUM
Был найден гомолог белка, но вместо матрицы BLOSUM62 была использована матрица BLOSUM45. Результат поиска можно увидеть в таблице 2. Из таблицы видно, что изменились значения E-value, вес выравнивания и процент сходства. Так как порог кластеризации в BLOSUM45 меньше, чем в BLOSUM62, вес замен некоторых аминокислот в BLOSUM45 будет больше. Этим можно объяснить изменение данных параметров.
5. Сравнение различных интерфейсов программы BLAST
В задании требовалось сравнить интерфейсы программы BLAST на серверах NCBI, EMBL-EBI и UniProt. Интерфейс программы на сервере UniProt очень лаконичный: можно ввести последовательность или идентификатор, задать порог E-value, максимальное количество результатов, матрицу весов замен аминокислотных остатков, наличие гэпов и фильтр. На сервере EMBL-EBI при поиске можно задать чуть больше параметров, лист запроса разбит на неколько шагов: выбор базы данных, введение последовательности, настройка параметров. Самым удобным, на мой взгляд, является интерфейс программы на NCBI.