Комплексы ДНК-белок
Задание 1
Упражнение 1
С помощью команды define в Jmol были определены множества атомов ДНК из файла 1LLR.pdb: set1 - множество атомов кислорода 2'-дезоксирибозы , set2 - множество атомов кислорода в остатке фосфорной кислоты, set3 - множество атомов азота в азотистых основаниях. Создан скрипт-файл, вызов которого дает последовательное изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов set1, set2 и set3. Результат программы можно увидеть на рисунках 1-5.
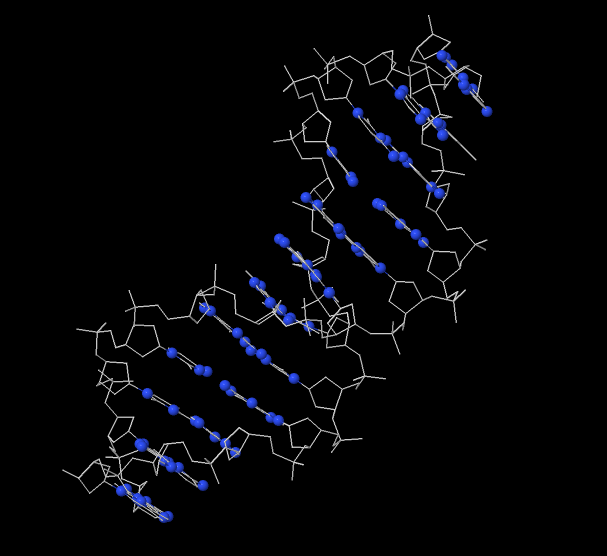
Упражнение 2
Для того, чтобы найти контакты ДНК с белком был написан скрипт. Полярными считали атомы кислорода и азота, а неполярными ? атомы углерода, фосфора и серы. Полярным контактом считали ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5A. Аналогично, неполярным контактом считали пару неполярных атомов на расстоянии меньше 4.5A. Результат можно увидеть в таблице ниже.
| Контакты белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 3 | 21 | 24 |
| остатками фосфорной кислоты | 14 | 16 | 30 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 6 | 8 |
| остатками азотистых оснований со стороны малой бороздки | 1 | 0 | 1 |
Неполярных контактов больше, чем полярных. Наибольшее число контактов у белка с остатком фосфорной кислоты и остатками 2'-дезоксирибозы, что связано с их пространственным расположением (они лучше доступны для контактов с белком). Наименьшое число контактов с атомами азотистых оснований со стороны малой бороздкисвязано с их пространственной недоступностью.
Упражнение 3 и 4
С помощью программы nucplot была получена схема контактов белка с ДНК.
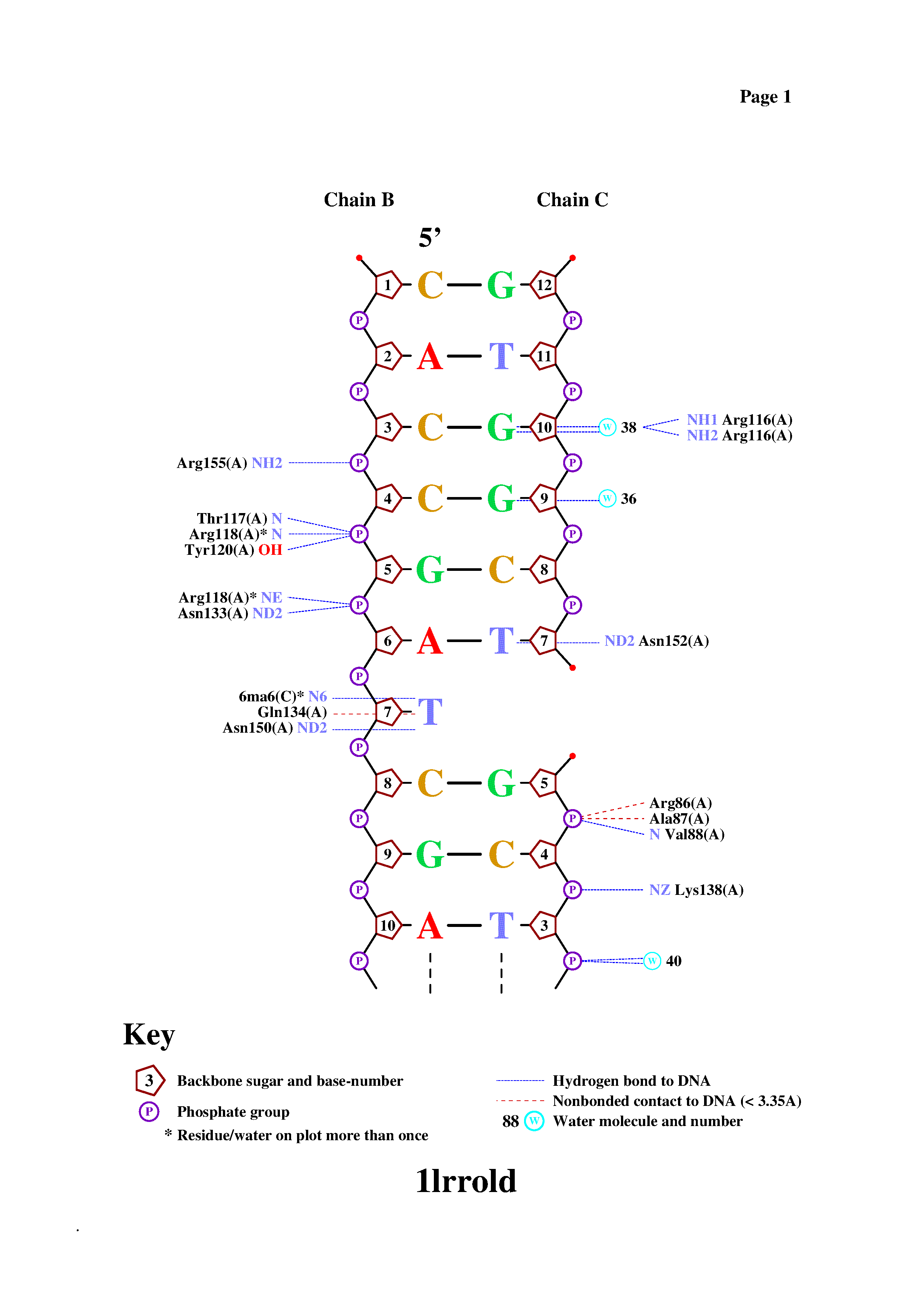
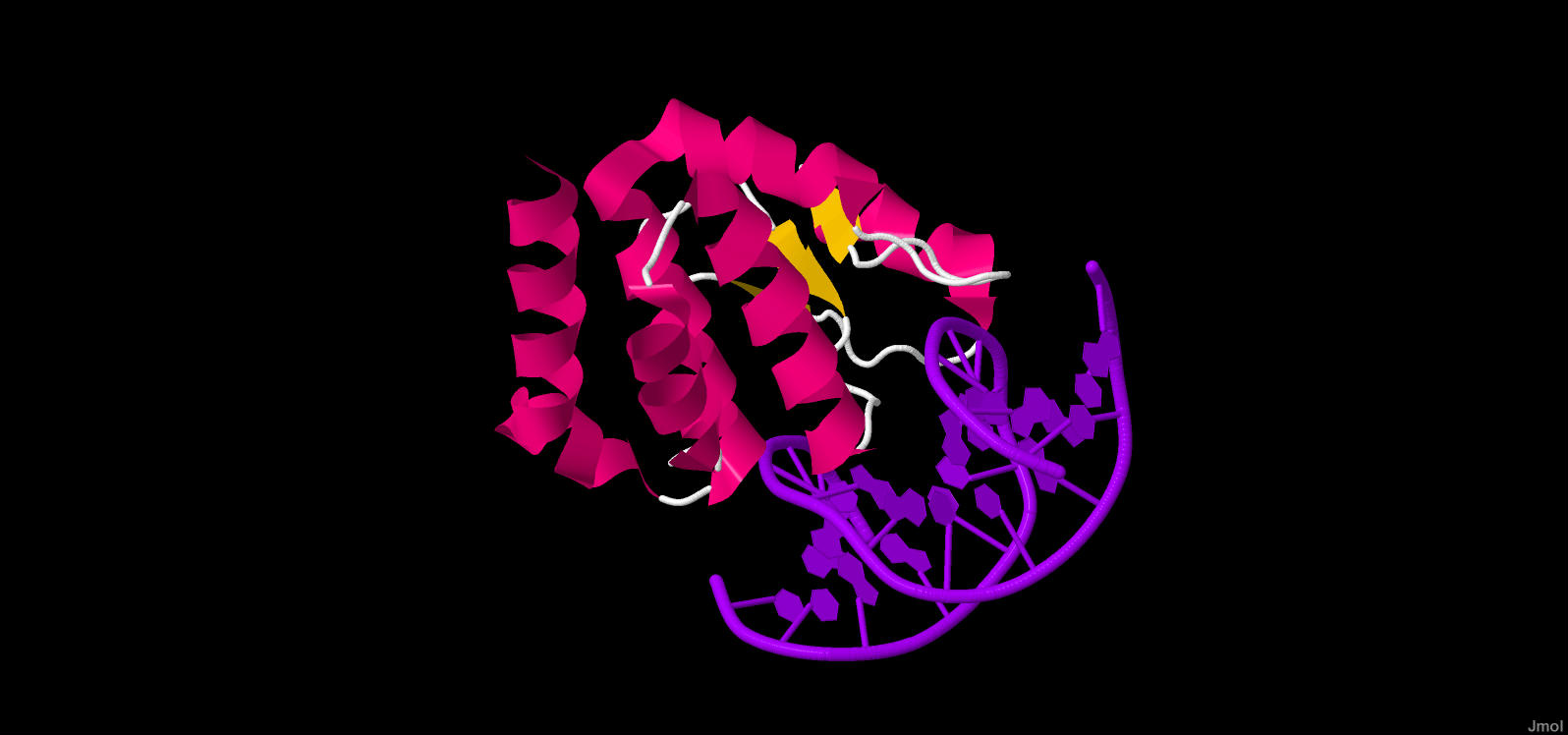
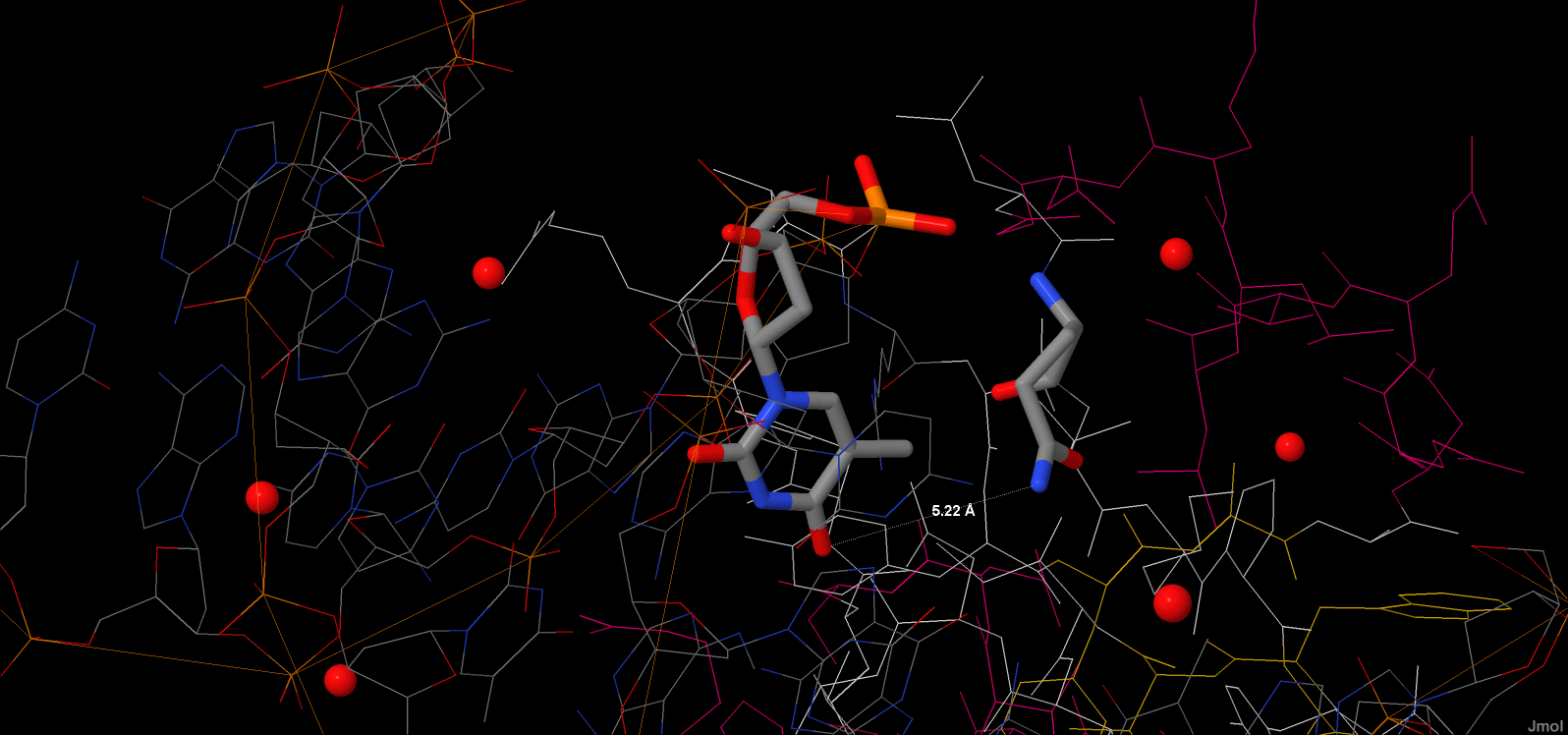
Максимальное количество контактов было ДНК имеет c Arg118 (2 контакта). Контакты этого аминокислотного остатка с ДНК показаны на следующем изображении, полученным с помощью программы JMol:
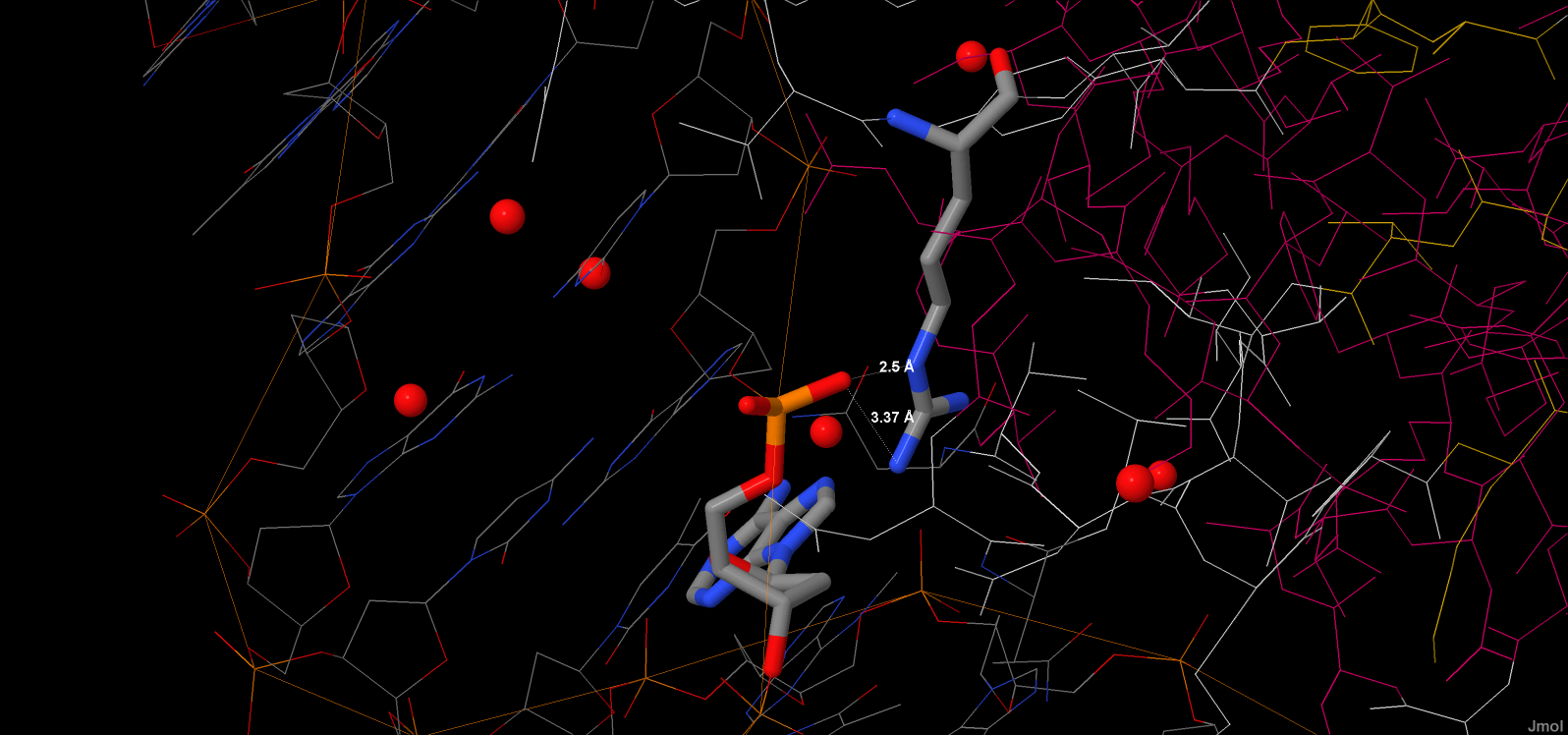
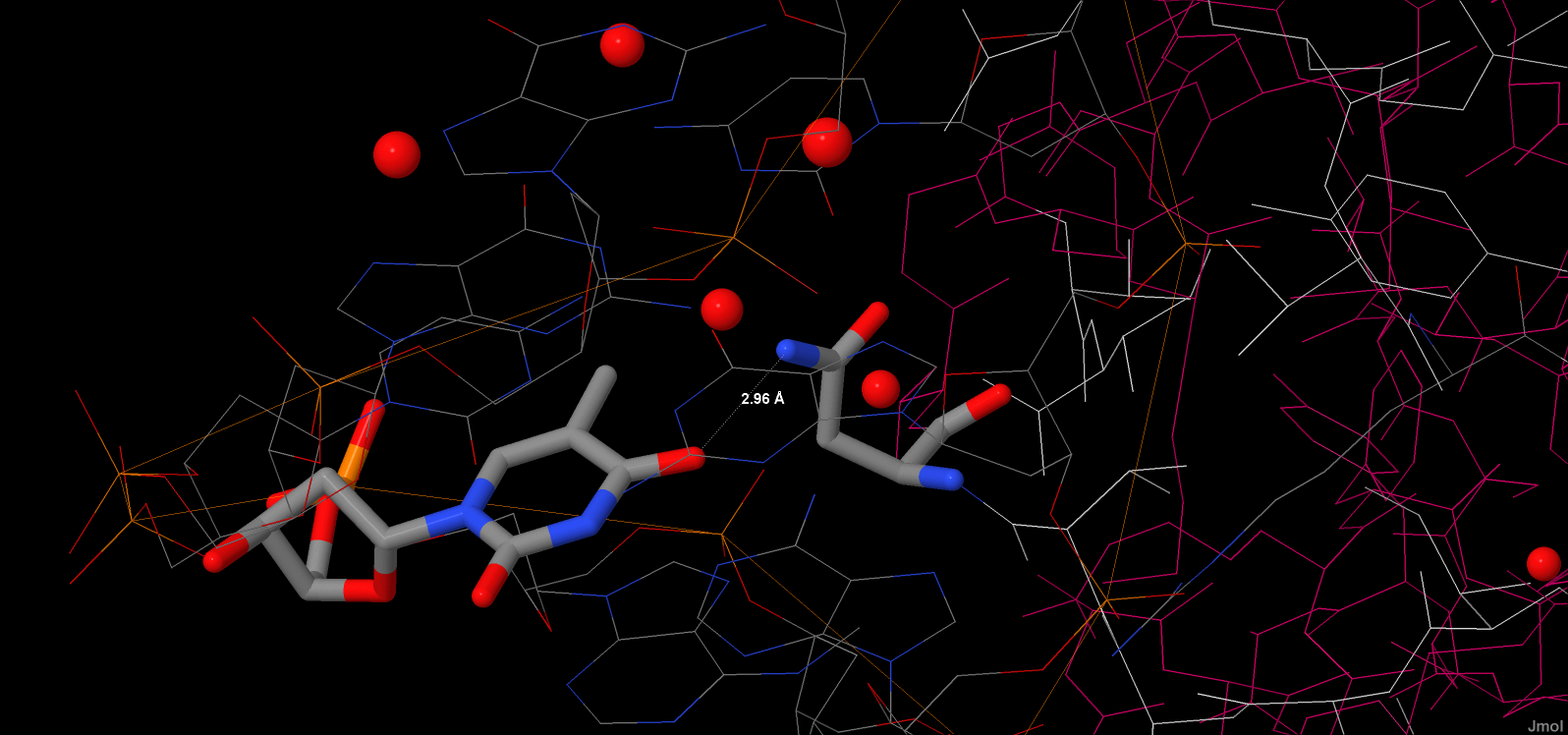
Для участия в распознавании последовательности ДНК аминокислотный остаток должен связываться с азотистым основанием. Такими аминокислотными остатками являются Gln134, Asn152. Контакты аминокислотных остатков с ДНК показаны на рисунках 8 и 9.
Задание 2
Упражнение 1
В задании работали с тРНК 1QTQ. С помощью программы einverted была получена информация об инвертированных участках тРНК. Инвертированные участки не были найдены при настройках по умолчанию. При параметрах
gap penalty 20 minimum score threshold 25 match score 10 mismatch score -20был найден только акцепторный стебель.
Параметры неоднократно менялись, наиболее удачными, на мой взгляд оказались:
gap penalty 10 minimum score threshold 25 match score 5 mismatch score -2
При таких настройках нашелся акцепторный стебель, большая часть D-стебля, пара T-стебля и 3 пары антикодонового стебля(с большими пропусками). Есть сомнения насчет того, стоит ли верить этому выравниванию.
Упражнение 2
С помощью программы mfold были получено предсказание вторичной структуры тРНК по алгоритму Зукера. Наиболее удачное предсказание оказалось при параметре P=0.
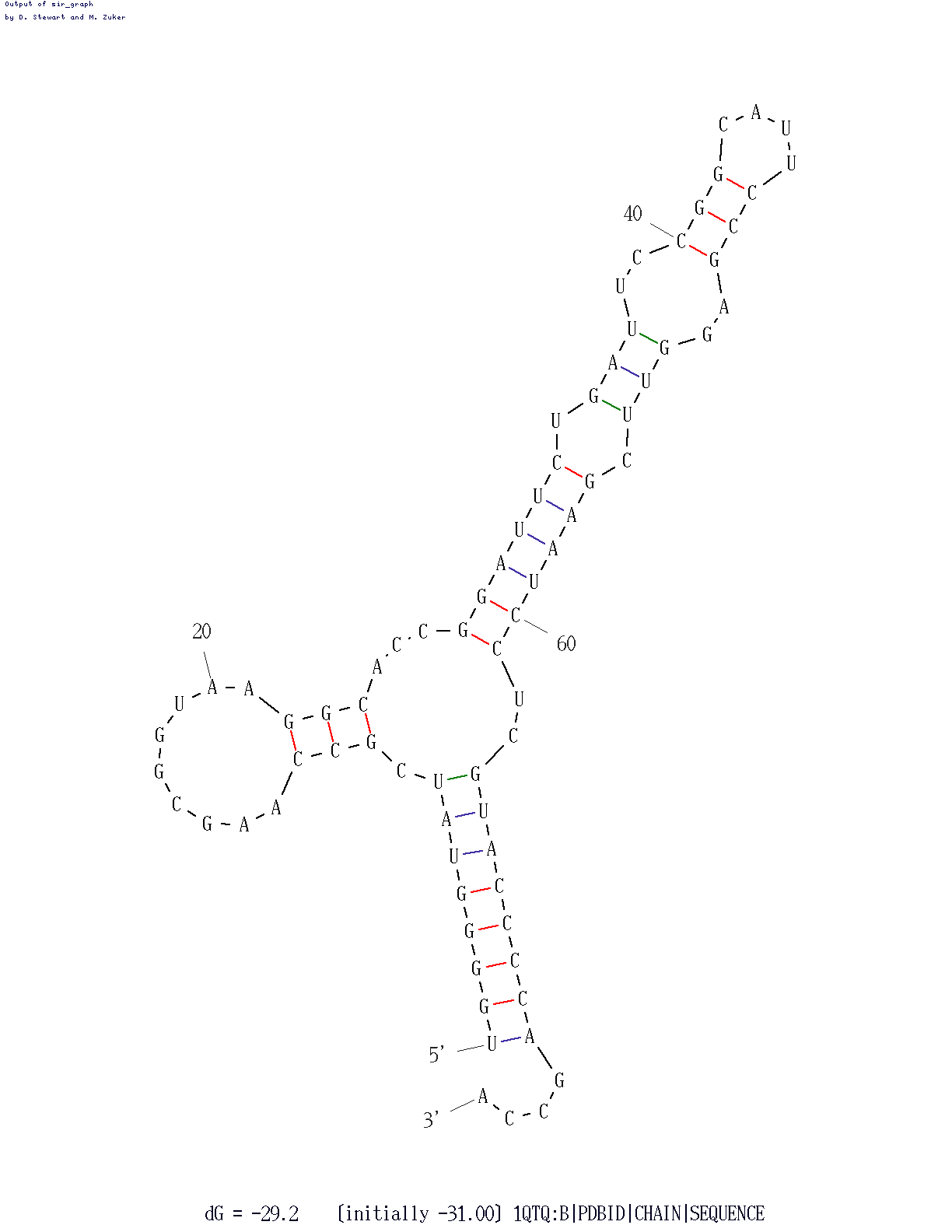
Сравнение результатов программ, с помощью которых в этом задании мы пытались прдсказать вторичную структуру тРНК 1QTQ можно посмотреть в таблице:
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-902-907-3' 5'-966-970-3' всего 7 пар |
7 из 7 | 7 из 7(одна лишняя пара) |
| D-стебель | 5'-910-912-3' 5'-923-925-3' всего 3 пары |
2 из 3 | 3 из 3 |
| T-стебель | 5'-949-953-3' 5'-926-933-3' всего 8 пар |
1 из 3 | 6 из 8 |
| Антикодоновый стебель | 5'-937-944-3' 5'-961-965-3' всего 5 пар |
3 из 5 | 3 из 5 |
| Общее число канонических пар нуклеотидов | 23 | 13 | 18 |