Занятие 3
Напомним структуру правильного дерева.
Задание 1. Укоренение в среднюю точку
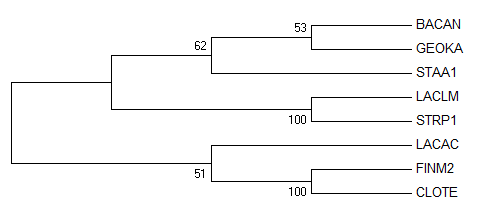
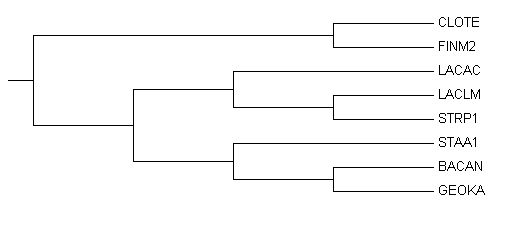
Дерево, полученное в предыдущем практикуме, с помощью программы JalView методом Neighbour Joining Using % Identity не являлось ультраметрическим, и корень в нем не соответствовал корню правильного дерева. Для того, чтобы укоренить это дерево в среднюю точку, использовалась программа retree пакета Phylip. Укоренение в среднюю точку означает, что расстояние от корня до всех ветвей станет одинаковым, то есть принимается гипотеза молекулярных часов. На рисунках 1 и 1' изображены деревья из JalView изначальное и переукорененное соответственно
Из рисунка 1' можно увидеть, что теперь корень стоит правильно - он разделяет Bacilli и Clostridia.
Задание 2. Использование внешней группы
Метод максимальной экономии ("Maximum parsimony") предполагает работу с выравниванием последовательностей и не возвращает длины ветвей, поэтому дерево построенное таким методом нельзя укоренить в среднюю точку. Но можно воспользоваться укоренением с помощью внешней группы. В качестве внешней группы выбрали белок EFG_ECOLI, принадлежащий E.coli.
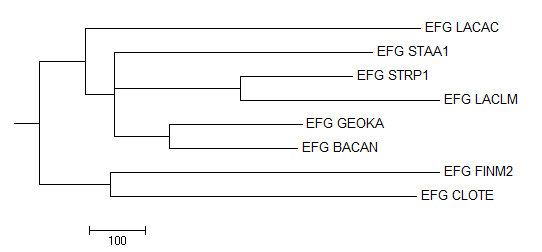
На рисунке 2 изображено дерево, полученное в предыдущем практикуме в MEGA методом "Maximum parsimony", переукорененное с помощью внешней группы.
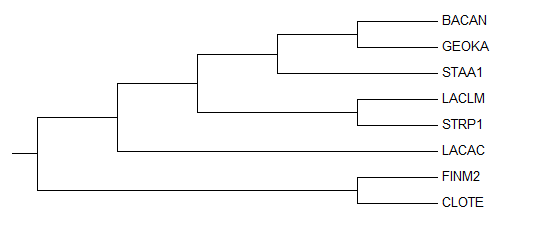
Укоренение произошло в ветвь {CLOTE, FINM2} vs {LACAC, LACLM, STRP1, STAA1, BACAN, GEOKA}, как и в предыдущем задании. Это правильное укоренение. Другие ветви не изменились, отличаются от правильного так же, как исходное.
Задание 3. Бутстрэп
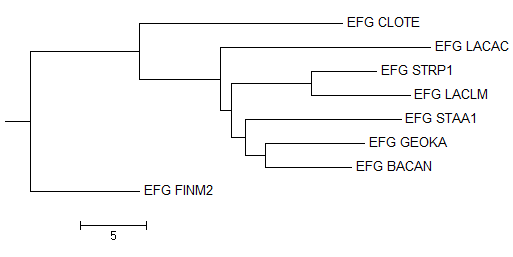
Провели бутстрэп-анализ белков с числом реплик, равным 100. Программа выдала два дерева - "Original tree" и "Bootstrap consensus tree", которые не отличались. На рисунке 3 они показаны. К правильному они не очень близко - LACAC в одной группе с Clostridia, причем FINM2 и CLOTE считаются более молодой группой, которая отошла от их общего с LACAC предка. Не могу сказать, что неправильная ветвь имеет бОльшую поддержку, но таковую она имеет.