Реконструкция филогенетических деревьев
Задание 1
В таблице представлено, в каких таксонах находятся бактерии, выбранные в предыдущем практикуме.
| Название | Мнемоника | Тип | Класс | Отряд | Семейство |
| Bacillus anthracis | BACAN | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Clostridium tetani | CLOTE | Firmicutes | Clostridia | Clostridiales | Clostridiaceae |
| Finegoldia magna | FINM2 | Firmicutes | Clostridia | Clostridiales | Clostridiales Family XI. Incertae Sedis |
| Geobacillus kaustophilus | GEOKA | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Lactobacillus acidophilus | LACAC | Firmicutes | Bacilli | Lactobacillales | Lactobacillaceae |
| Lactococcus lactis | LACLM | Firmicutes | Bacilli | Lactobacillales | Streptococcaceae |
| Staphylococcus aureus | STAA1 | Firmicutes | Bacilli | Bacillales | Staphylococcaceae |
| Streptococcus pyogenes | STRP1 | Firmicutes | Bacilli | Lactobacillales | Streptococcaceae |
Напомним структуру дерева:
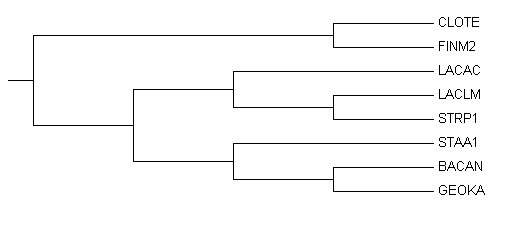
В таблице представлены нетривиальные ветви и таксоны, которые они разделяют:
| {CLOTE, FINM2} vs {LACAC, LACLM, STRP1, STAA1, BACAN, GEOKA} | Классы Bacilli и Clostridia |
| {CLOTE, FINM2, STAA1, BACAN, GEOKA} vs {LACAC, LACLM, STRP1} | Отряд Lactobacillales |
| {LACLM, STRP1} vs {CLOTE, FINM2, LACAC, STAA1, BACAN, GEOKA} | Семейство Streptococcaceae |
| {STAA1, BACAN, GEOKA} vs {CLOTE, FINM2, LACAC, LACLM, STRP1} | Отряд Bacillales |
| {BACAN, GEOKA} vs {STAA1, STRP1, LACLM, LACAC, FINM2, CLOTE} | Семейство Bacillaceae |
Задание 2
Для реконструкции филогенетического дерева было выбрано семество белков - факторов элонгации трансляции G (Мнемоника - EFG). Для получения записей в fasta-формате использовалась команда
seqret sw:EFG_BACAN
В JalView получили выравнивание, используя программу Muscle with Defaults, в "блочной" форме (Format > Wrap в меню), с блоками шириной 100 остатков, с раскраской по проценту идентичности. В выравниваниях можно найти диагностические позиции, по которым можно судить, к какому таксону принадлежат бактерии. Позиции для класса Bacilli отмечены на выравниваниях красным, для отряда Lactobacillales - желтым, семейтсва Lactobacillaceae - оранжевым, для семейства Streptococcaceae - бордовым, для семейства Staphylococcaceae - зеленым.

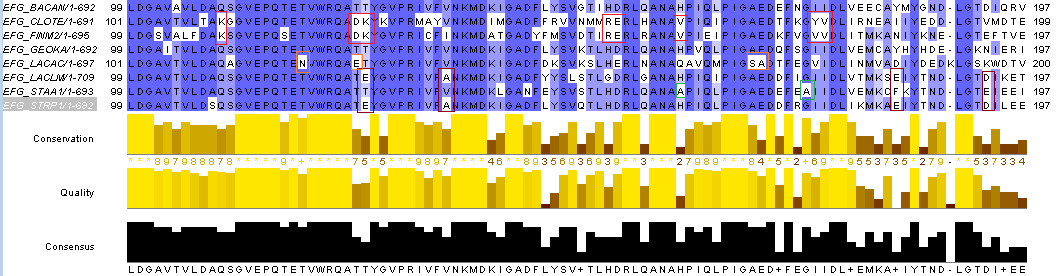



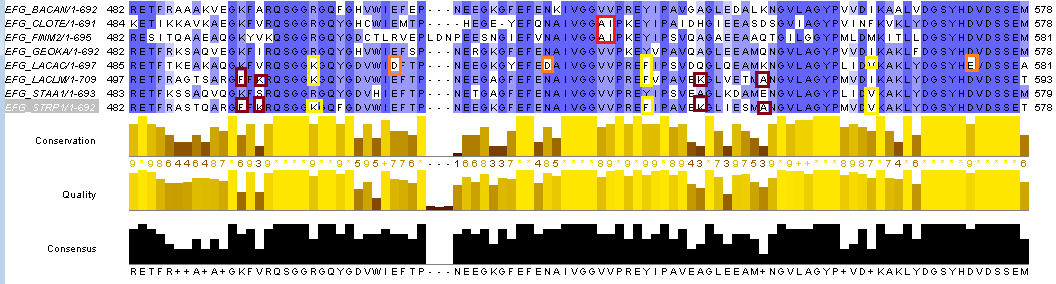


Филогенетическое дерево в JalView можно построить четяремя способами. Далее представлены изображения деревьев, построенных каждым способом, сходства и отличия их от настоящего.
Average Distance Using % Identity
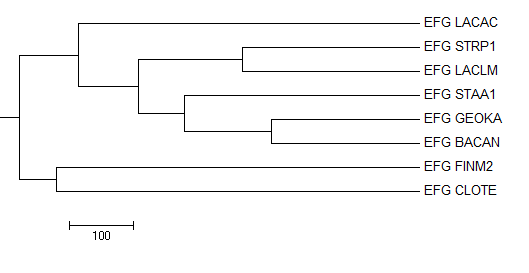
Общие ветви
- {CLOTE, FINM2} vs {LACAC, LACLM, STRP1, STAA1, BACAN, GEOKA}
- {BACAN, GEOKA} vs {STAA1, STRP1, LACLM, LACAC, FINM2, CLOTE}
- {STAA1, BACAN, GEOKA} vs {CLOTE, FINM2, LACAC, LACLM, STRP1}
- {LACLM, STRP1} vs {CLOTE, FINM2, LACAC, STAA1, BACAN, GEOKA}
Отличия от правильного дерева
- {CLOTE, FINM2, LACAC} vs {LACLM, STRP1, STAA1, BACAN, GEOKA}
В правильном дереве была ветвь {CLOTE, FINM2, STAA1, BACAN, GEOKA} vs {LACAC, LACLM, STRP1}.
Average Distance Using BLOSUM62
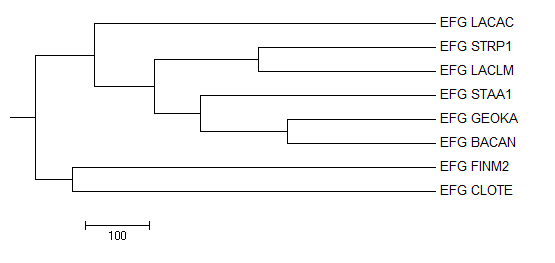
Дерево полностью совпадает с предыдущим.
Neighbour Joining Using % Identity
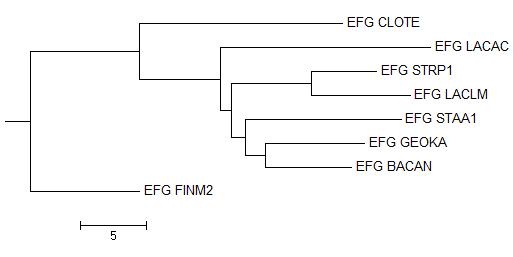
Общие ветви
- {CLOTE, FINM2} vs {LACAC, LACLM, STRP1, STAA1, BACAN, GEOKA}
- {LACLM, STRP1} vs {CLOTE, FINM2, LACAC, STAA1, BACAN, GEOKA}
- {BACAN, GEOKA} vs {STAA1, STRP1, LACLM, LACAC, FINM2, CLOTE}
- {STAA1, BACAN, GEOKA} vs {CLOTE, FINM2, LACAC, LACLM, STRP1}
Отличия от правильного дерева
- {CLOTE, FINM2, LACAC} vs {LACLM, STRP1, STAA1, BACAN, GEOKA}
В правильном дереве была ветвь {CLOTE, FINM2, STAA1, BACAN, GEOKA} vs {LACAC, LACLM, STRP1}.
Neighbour Joining Using BLOSUM62
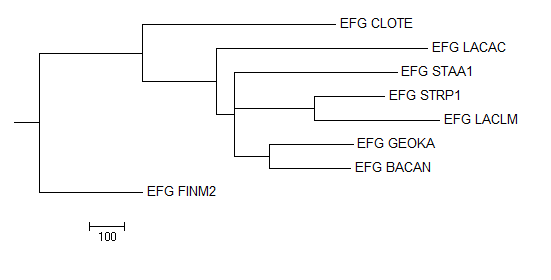
Дерево не является бинарным.
Общие ветви
- {BACAN, GEOKA} vs {STAA1, STRP1, LACLM, LACAC, FINM2, CLOTE}
- {LACLM, STRP1} vs {CLOTE, FINM2, LACAC, STAA1, BACAN, GEOKA}
- {CLOTE, FINM2} vs {LACAC, LACLM, STRP1, STAA1, BACAN, GEOKA}
Отличия от правильного дерева
- {CLOTE, FINM2, LACAC} vs {LACLM, STRP1, STAA1, BACAN, GEOKA}
В правильном дереве были две отличные от реконструированного ветви:
- {STAA1, BACAN, GEOKA} vs {CLOTE, FINM2, LACAC, LACLM, STRP1}
- {CLOTE, FINM2, STAA1, BACAN, GEOKA} vs {LACAC, LACLM, STRP1}
Было построено дерево с помощью программы MEGA.
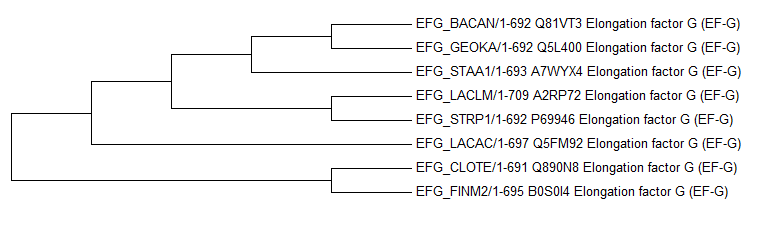
Общие ветви
- {CLOTE, FINM2} vs {LACAC, LACLM, STRP1, STAA1, BACAN, GEOKA}
- {BACAN, GEOKA} vs {STAA1, STRP1, LACLM, LACAC, FINM2, CLOTE}
- {STAA1, BACAN, GEOKA} vs {CLOTE, FINM2, LACAC, LACLM, STRP1}
- {LACLM, STRP1} vs {CLOTE, FINM2, LACAC, STAA1, BACAN, GEOKA}
Отличия от правильного дерева
- {CLOTE, FINM2, LACAC} vs {LACLM, STRP1, STAA1, BACAN, GEOKA}
В правильном дереве была ветвь {CLOTE, FINM2, STAA1, BACAN, GEOKA} vs {LACAC, LACLM, STRP1}.