Занятие 8. Особенности мембранных белков
Структуры мембранных белков
Существует два основных типа вторичной структуры трансмембранных белков: альфа-спирали и "бочонки" из бета-листов, или баррели. В таблице представлены основные параметры шести случайных белков: трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков . Для выполенения задания использовались базы данных OPM и TCBD.
Таблица 1. Описание трансмембранных белков с известной 3D структурой
| PDB код | Тип (спираль, баррель) |
Какая мембрана (внутренняя или внешняя, организм, органелла) |
Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 4A2N | Спираль | Мембрана архей(заякорен с внутренней стороны) Methanosarcina acetivorans (Метилтрансфераза) |
30.6 ± 1.2 | 22 |
| 2j58 | Спираль | Внутренняя мембрана грам-отрицательных бактерий Escherichia coli (Транспортер полисахаридов) |
30.6 ± 1.2 | 29 |
| 2UUH | Спираль | Мембрана ЭПР Homo sapiens (Лейкортринсинтаза) |
29.4 ± 1.1 | 18.5 |
| 1UUN | Баррель | Внешняя мембрана грам-отрицательных бактерий Mycobacterium smegmatis (Порин MspA) |
40.7 ± 2.1 | 9.5 |
| 1AF6 | Баррель | Внешняя мембрана грам-отрицательных бактерий Escherichia coli (Мальтотропин) |
25.1 ± 0.7 | 9 |
| 2JK4 | Баррель | Внешняя мембрана митохондрий Homo sapiens (Потенциал-зависимый анионный канал) |
23.4 ± 2.3 | 7 |
Можно заметить, что у трансмембранных бета-баррелей толщина гидрофобной части и число остатков в трансмембранном участке, как правило, меньше. Это связано с тем, что у числа остатков в альфа-спирали есть определенный минимальный порог, и в альфа-спираль "умещается" больше аминокислотных остатков.
Отбор гомологов белка 4EZC
Название данного белка - Urea transporter 1 (PDB ID - 4EZC, Uniprot - Q5QF96 (UT1_BOVIN)). Он принадлежит Bos taurus (Бык дикий) и участвует в транспорте мочевины.
Для поиска гомологов использовали программу blastp. Поиск проводился по базе данных RefSeq, , e-value = 1, максимальное число хитов = 1000. BLAST выдал 275 последовательностей, из них выбрали 15 гомологов из разных групп (среди которых были Членистоногие(Насекомые(Перепончатокрылые)), Млекопитающие (Приматы, Грызуны, Парнопытные, Китообразные),Бесчерепные и т д) с e-value<=1e-6. Проанализировав записи о белках в RefSeq, пришли к выводу, что гены, кодирующие белок, находятся в ядре.
Основная информация о белке 4EZC
Основная информация о структуре белка содржится в таблице. Была использована база данных OPM.
Таблица 2.Описание структуры трансмембранного белка UT1_BOVIN ((идентификатор PDB 4EZC, цепь A)
| PDB ID | Организм | Тип мембраны | TC-код | Угол наклона спиралей к нормали | Количество трансмембранных спиралей |
| 4EZC | Bos taurus | Плазматическая мембрана эукариот | 1.A.28 | 0 ± 0° | 30 (В каждой цепи по 10) |
Расшифровка кода TCDB: 1 - Каналы/ Поры; A - альфа-спиральные каналы; 28 - семейство транспортеров мочевины.
Анализ множественного выравнивания белков
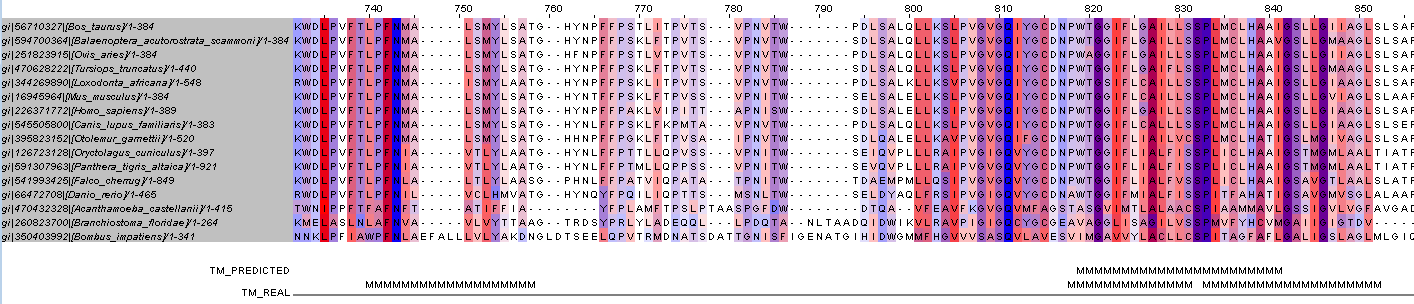
Осуществили множественное выравнивание белков с помощью программы Muscle. К выравниванию была добавлена аннотация TM_REAL, в которой было отмечено буквой "М", какие участки исходного белка являются альфа-спиралями. Эти участки нашли благодаря тому, что привязали к последовательности исходного белка его структуру.
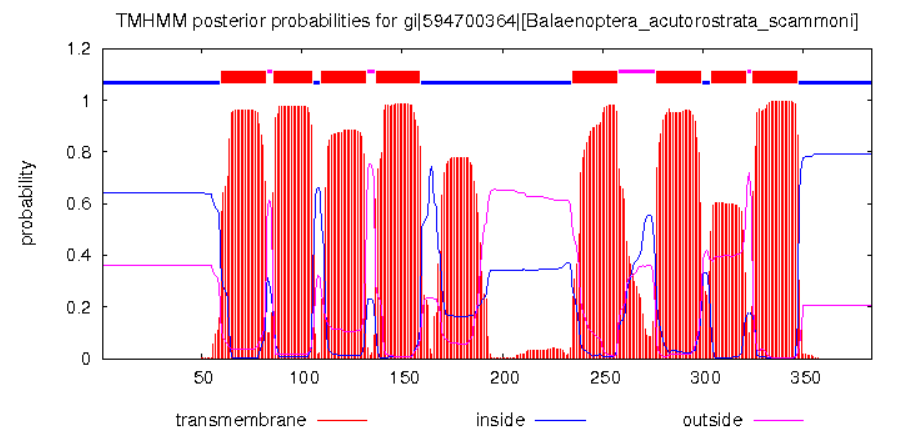
Затем к гомологу, принадлежащему Balaenoptera acutorostrata scammoni, была применена программа TMHMM, предсказывающая вторичную структуру:
# gi|594700364|[Balaenoptera_acutorostrata_scammoni] Length: 384 # gi|594700364|[Balaenoptera_acutorostrata_scammoni] Number of predicted TMHs: 8 # gi|594700364|[Balaenoptera_acutorostrata_scammoni] Exp number of AAs in TMHs: 178.63055 # gi|594700364|[Balaenoptera_acutorostrata_scammoni] Exp number, first 60 AAs: 0.99152 # gi|594700364|[Balaenoptera_acutorostrata_scammoni] Total prob of N-in: 0.63961 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 inside 1 59 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 TMhelix 60 82 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 outside 83 85 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 TMhelix 86 105 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 inside 106 109 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 TMhelix 110 132 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 outside 133 136 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 TMhelix 137 159 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 inside 160 234 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 TMhelix 235 257 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 outside 258 276 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 TMhelix 277 299 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 inside 300 303 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 TMhelix 304 321 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 outside 322 324 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 TMhelix 325 347 gi|594700364|[Balaenoptera_acutorostrata_scammoni] TMHMM2.0 inside 348 384
В аннотацию было добавлено поле TM_PREDICTED, в котором буквой "М" были отмечены альфа-спирали, предсказанные программой.
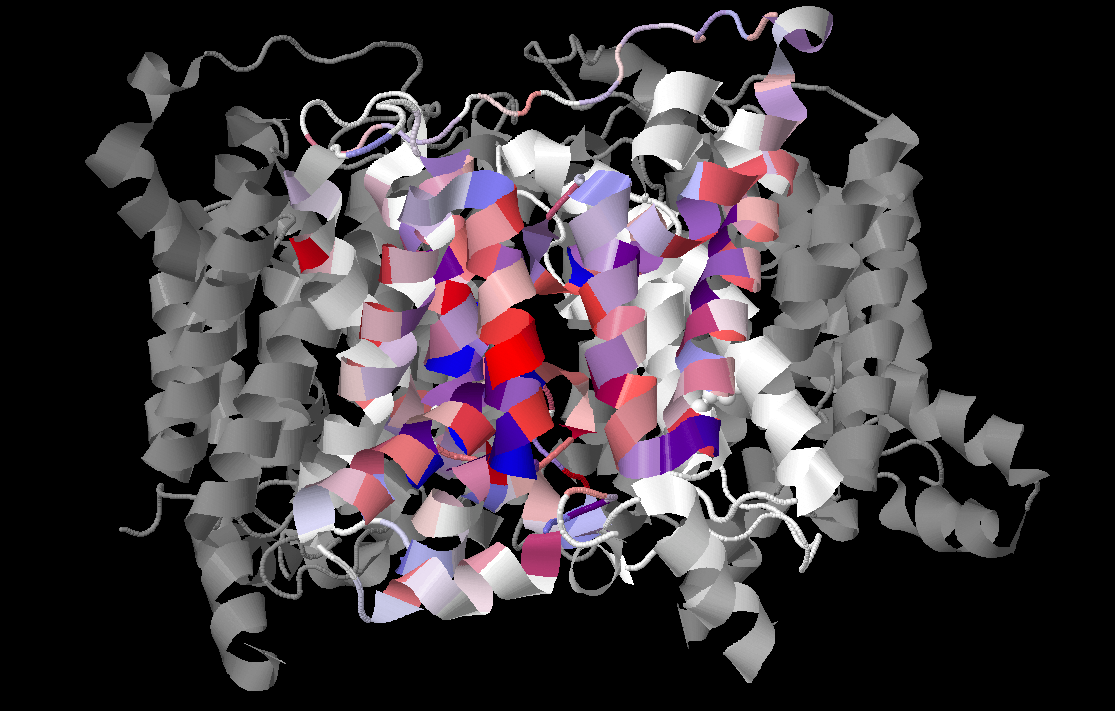
Для выравнивания была выбрана цветовая схема "Hydrophobicity"позволяющая визуально различать гидрофобные и гидрофильные остатки (гидрофобные остатки выделены красным цветом), причем интенсивность цвета зависит от того, насколько позиция консервативна (порог - 20%). Изображение выравнивания с отмеченными участками альфа-спиралей можно увидеть на рисунке 2 (показаны только те участки выравнивания, где были найдены альфа-спирали), а на рисунке 3 показана вторичная структура белка UT1_BOVIN. Раскрашена по гидрофобности только цепь А (так как она рассматривалась в предыдущих заданиях), другие цепи покрашены серым. Часть белка, ориентированная внутрь клетки, находится снизу.
Проект с выравниванием содержится в файле alignment.jar


Основные выводы по множественному выравниванию:
Большинство участков, встречающихся в альфа-спиралях, консервативны, но встречаются и неконсервативные фрагменты. Часто в альфа-спирялях встречаются остатки гидрофобных аминокислот - лейцина, изолейцина, валина, аланина, фениалаланина, метионина, глицина и пролина (консервативны).
Нередко встречаются остатки полярных аминокислот - серина, треонина, гистидина, аспарагина, тирозина, причем, как правило, они консервативны. Возможно, их присутствие связано с функцией белка (транспортер мочевины).
Про участки между спиралями можно сказать, что есть и консервативные, и неконсервативные, но первых больше.
С помощью программы TMHMM было найдено 8 альфа-спиралей из 10. В целом, результат программы и реальная структура совпадают, хотя спирали предсказанные и реальные бывают достаточно сильно смещены.