Задание 1. Построение и визуализация электронной плотности (ЭП)
Выбор структуры белка
Для анализа был выбран белок пиридоксалькиназа Bacillus subtilis (PDB code 2I5B). Его структура получена с разрешением 2.8 A
Данный белок соответствует следующим условиям:
- Белок есть в базе данных EDS, а это значит, что для белка доступны экспериментальные данные. Файл с электронной плотностью (structure factors) был скачан с сайта PDB.
- Для белка известно более 5 структурных гомологов с RMSD от 0.8 до 3.0 ангстрем и N_align от 50 до 90% числа аминокислотных его остатков. (Проверка осуществлялась с помощью PDBeFold) RMSD (Root Mean Square Deviation) это оценка среднего расстояния между C α-атомами соответствующих остатков при суперпозиции структур. Nalgn – длина выравнивания, т. е. число сопоставленных C α-атомов.
Визуализация электронной плотности
Для визуализации структуры использовалась программа PyMol. Ниже представлены изображения электронной плотности вокруг полипептидной цепи для уровней электронной плотности 1.0 σ, 1.5 σ и 2.0 σ на расстоянии 2 ангстрема. Для визуализации использовались следующие команды:
select backbone, name c+n+o+na isomesh backbone_map, 2i5b_map, 1, backbone, carve =2 isodot backbone_map, 2i5b_map, 1, backbone, carve =2 isomesh backbone_map, 2i5b_map, 1.5, backbone, carve = 2 isomesh backbone_map, 2i5b_map, 2, backbone, carve=2
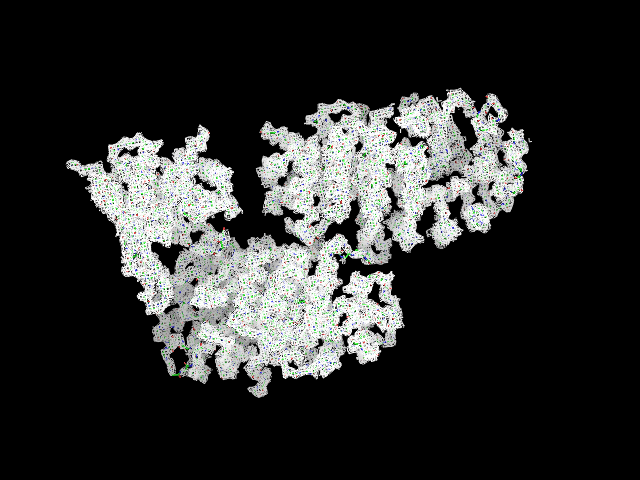
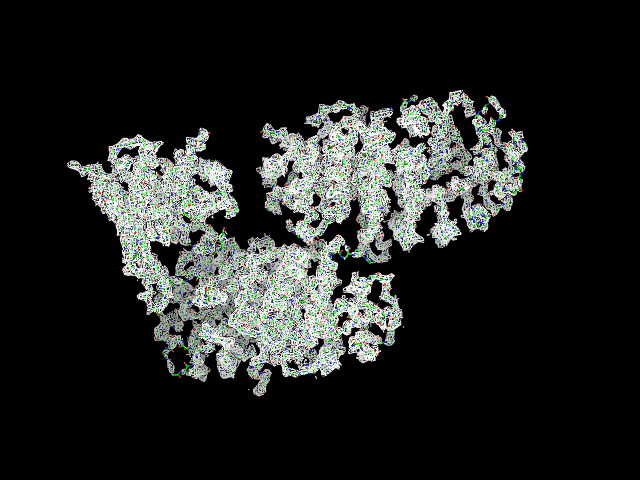
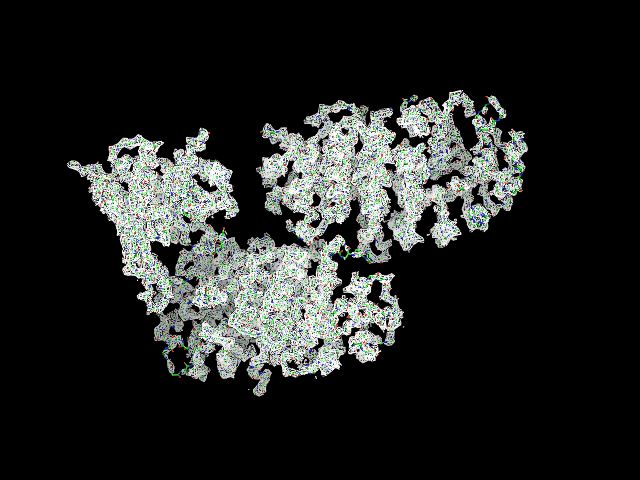
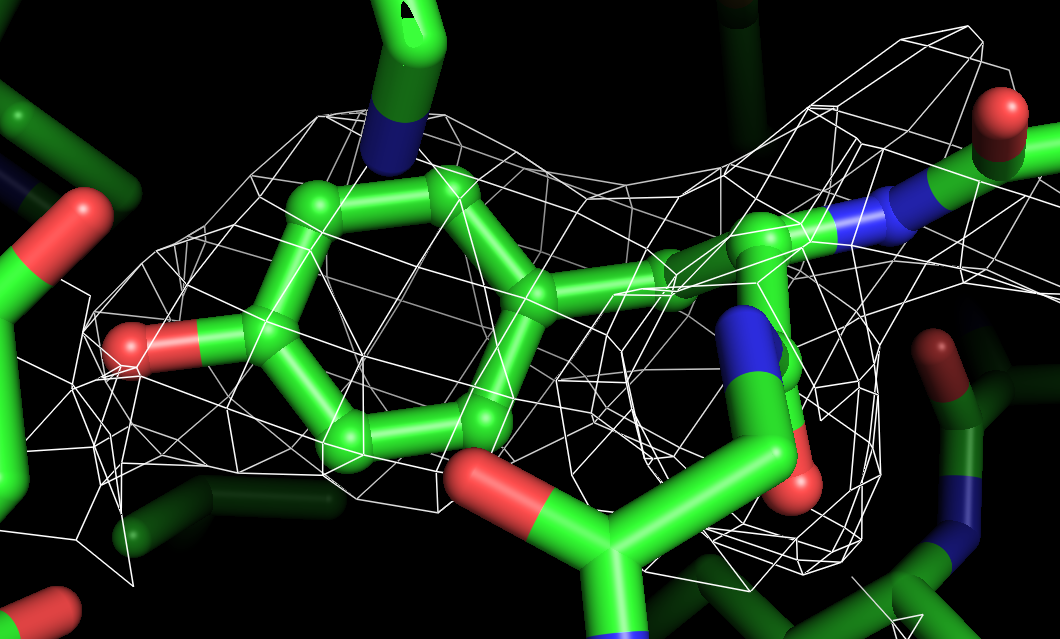
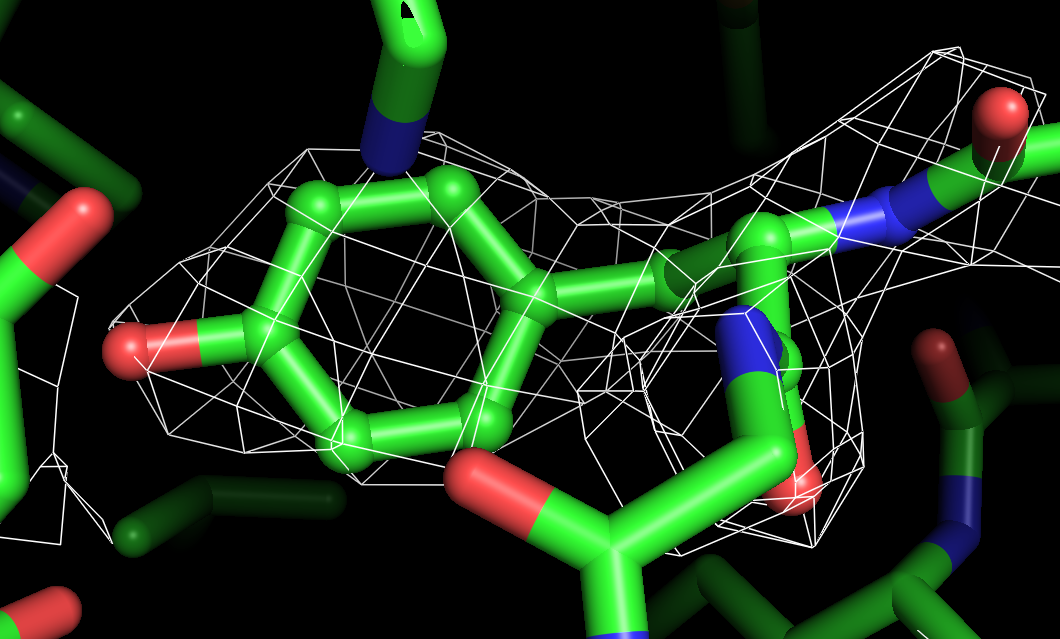
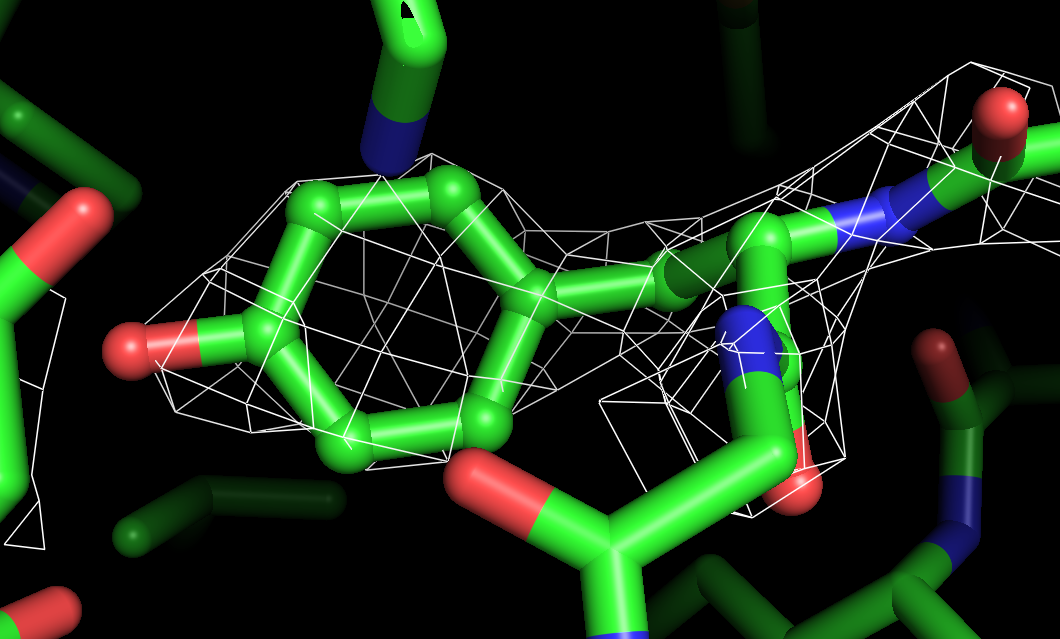
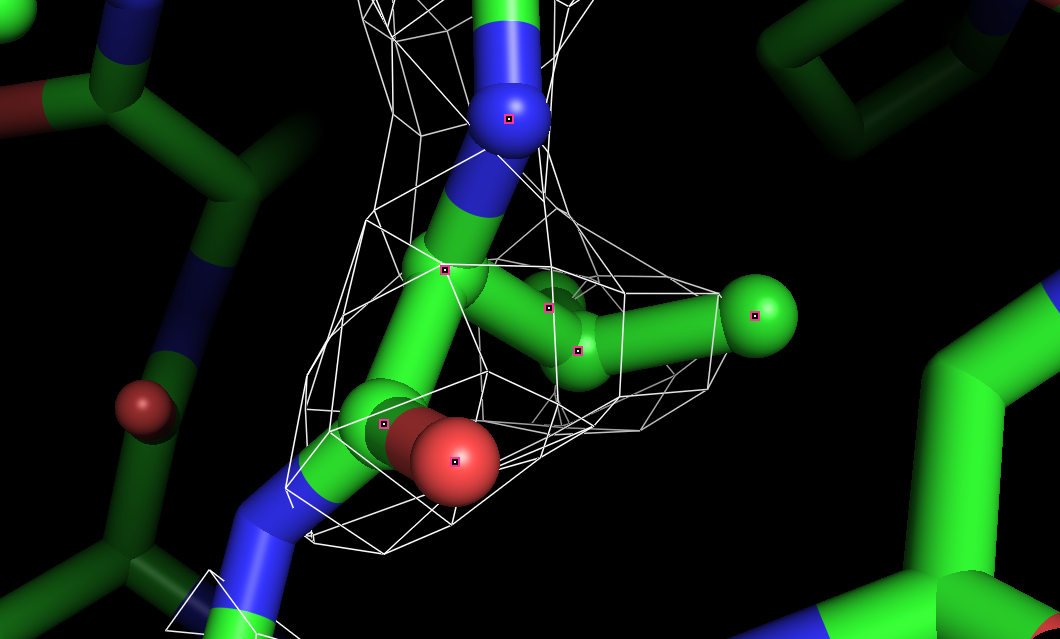
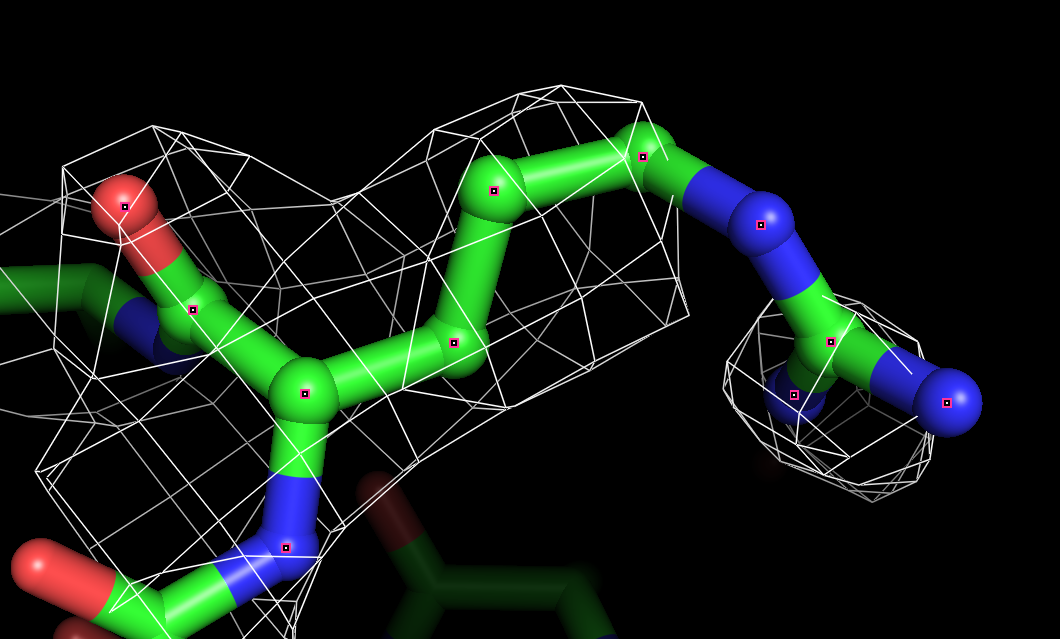
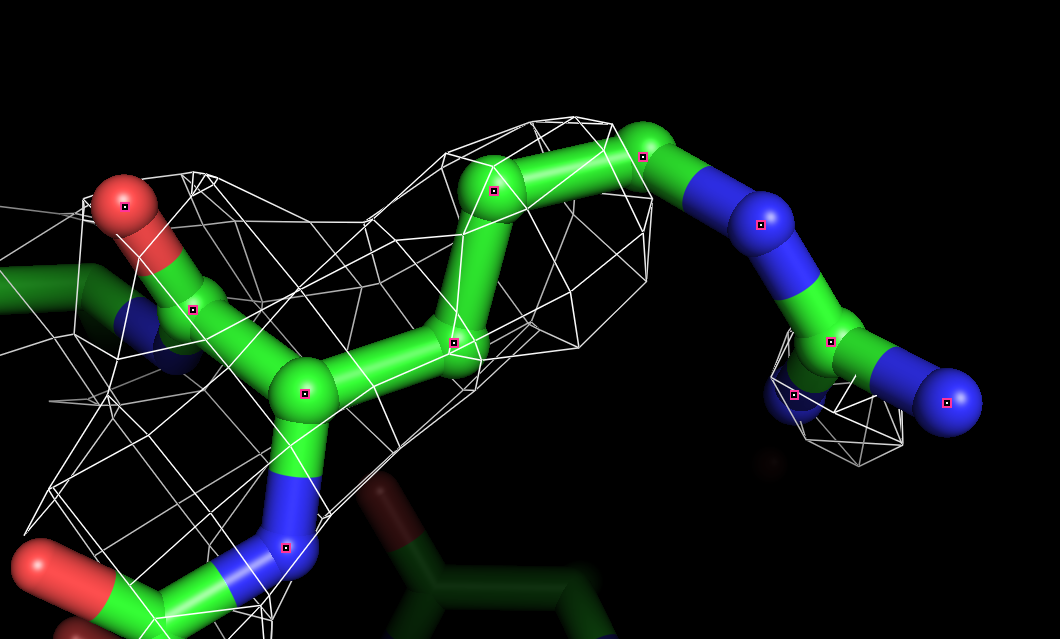
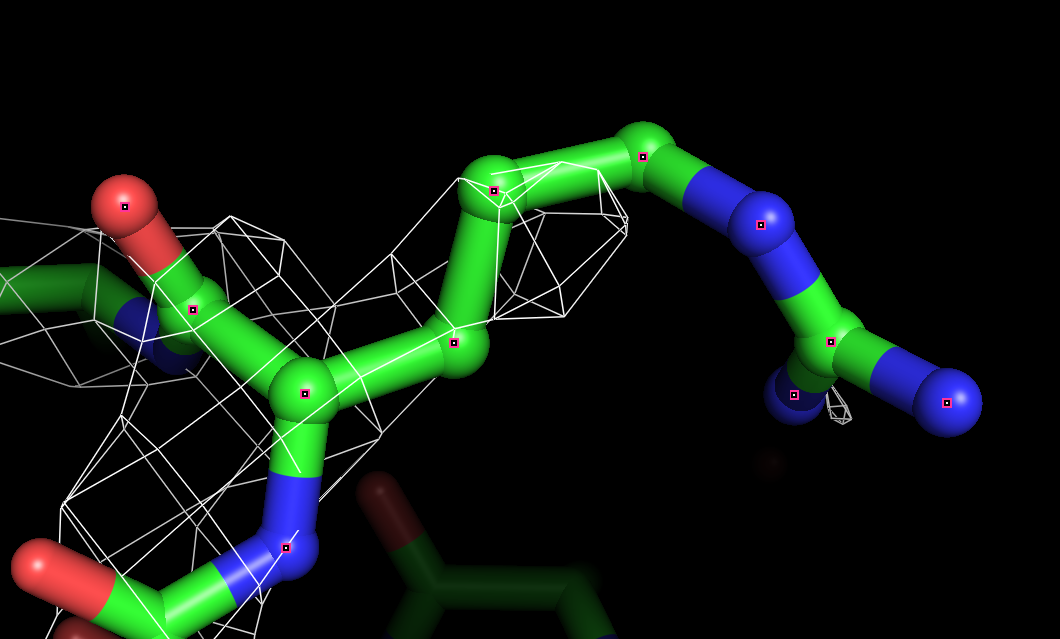
Электронная плотность аминокислотных остатков Thr:33, Val:40, Arg:63 цепи A для уровней подрезки 1.0 σ, 1.5 σ, 2.0 σ представлены ниже.
 |
 |
 |
| |
|
|
 |
 |
 |
| |
|
|
 |
 |
 |
| |
|
|
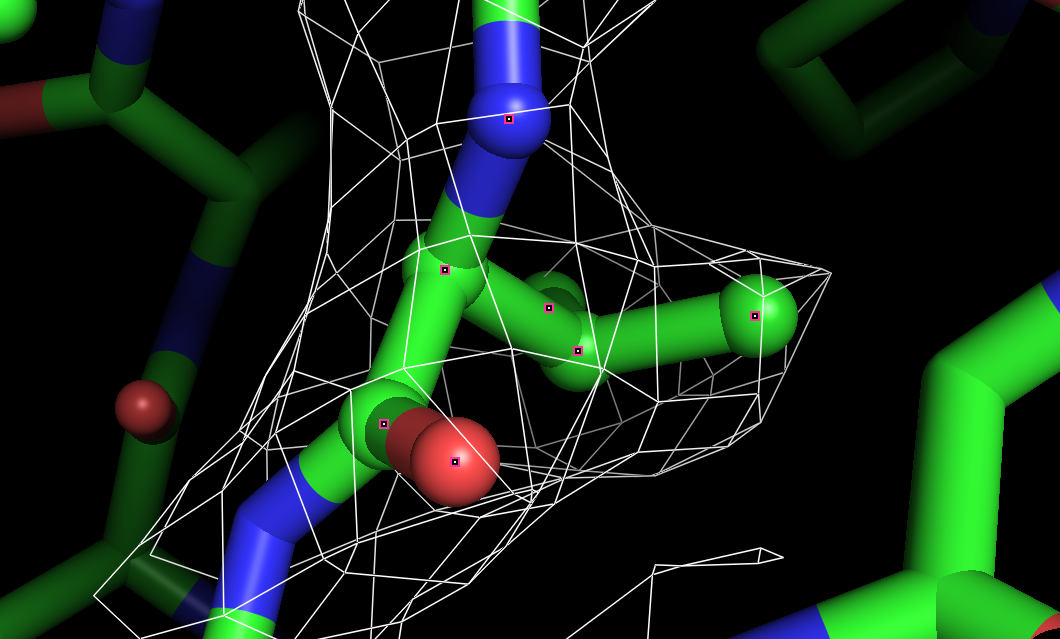
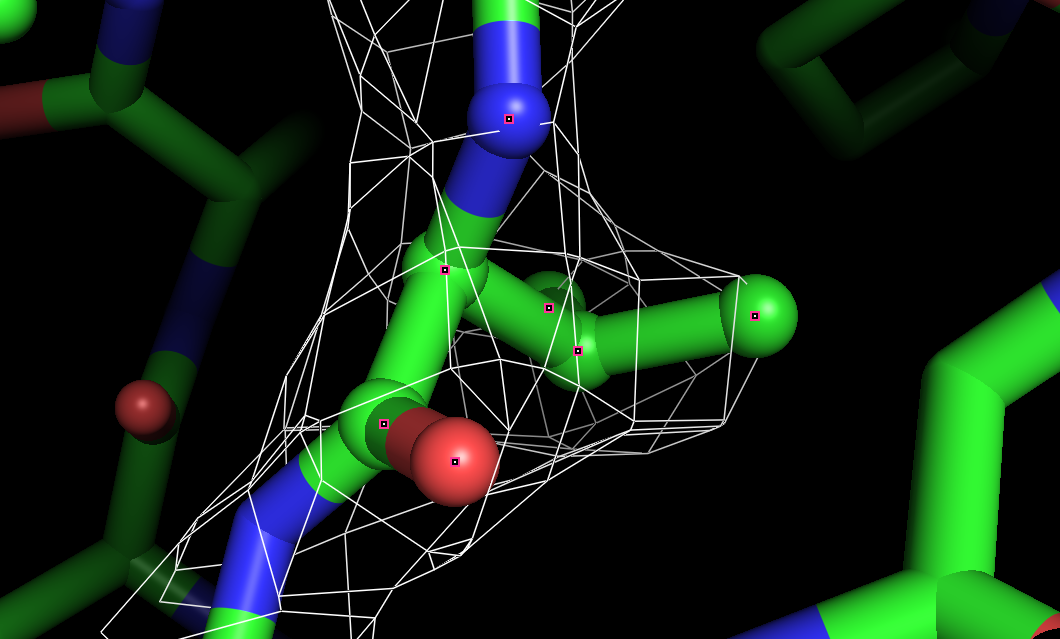
Координаты атомов соответствуют сгущениям электронной плотности. По сгущениям электронной плотности можно увидеть боковые группы аминокислотных остатков, хотя есть и несоответствия: например, электронной плотности нет вокруг атомов азота аргинина, при подрезке на уровне 2 не видно атом кислорода тирозина. Однако центры отдельных атомов увидеть нельзя, для этого не хватает разрешения.
Для получения изображений использовались следующие команды:
select thr, resi 33 and chain A show spheres, thr set sphere_scale, 0.2, thr isomesh map, 2i5b_map, 1.0, thr, carve=2.5 color gray30, map set mesh_width, 0.5
В целом, разрешения структуры достаточно для того, чтобы наблюдать большинство атомов боковых групп аминокислотных остатков.