import pybel
from os import system
Получим геометрию порфирина с помощью pybel.
def m3d(smiline):
mol=pybel.readstring('smi',smiline)
mol.addh()
mol.make3D(steps=1000)
return mol
##сделать мол-тип из смайла пибелем
porph = 'C1=CC2=CC3=CC=C(N3)C=C4C=CC(=N4)C=C5C=CC(=N5)C=C1N2'
mol = m3d(porph)
##получение 3d-координат с помощью pybel
mol.write(format='pdb',filename='pybel_porph.pdb',overwrite=True)
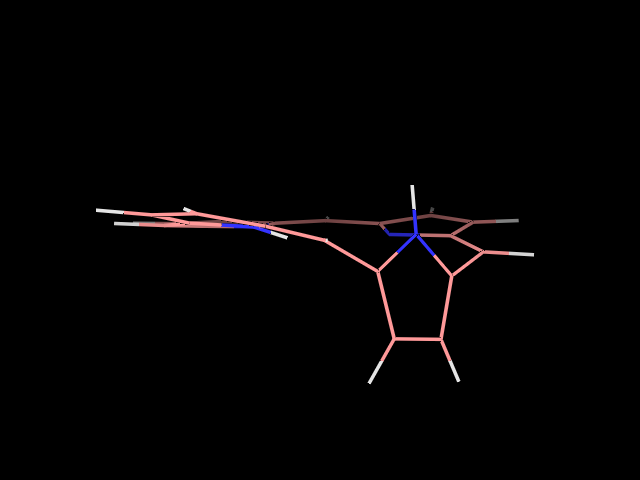
Как видно из рисунка, геометрия порфирина не соответствует реальности. Порфирин должен быть плоским, а получился изогнутым. Проведем оптимизацию геометрии порфирина разными способами.
def mopac(smiline,typea,name):
mol=pybel.readstring('smi',smiline)
mol.addh()
mol.make3D(steps=1000)
mop=mol.write(format='mopin',filename=name+typea+'.mop',opt={'k':typea+' CHARGE=%d' %mol.charge}, overwrite=True)
mopac_command = '/home/preps/golovin/progs/mopac/MOPAC2016.exe '+name+typea+'.mop'
system(mopac_command)
opt=pybel.readfile('mopout',name+typea+'.out')
for i in opt:
print i
i.write(format='pdb',filename=name+typea+'.pdb', overwrite=True)
##функция, позволяющая провести оптимизацию геометрии молекулы
mopac(porph,'AM1','porph')

Как видно из рисунка, опитимизированная AM1 геометрия порфирина тоже не соответствует реальности. Порфирин должен быть плоским, а получился изогнутым.
mopac(porph,'PM6','porph')
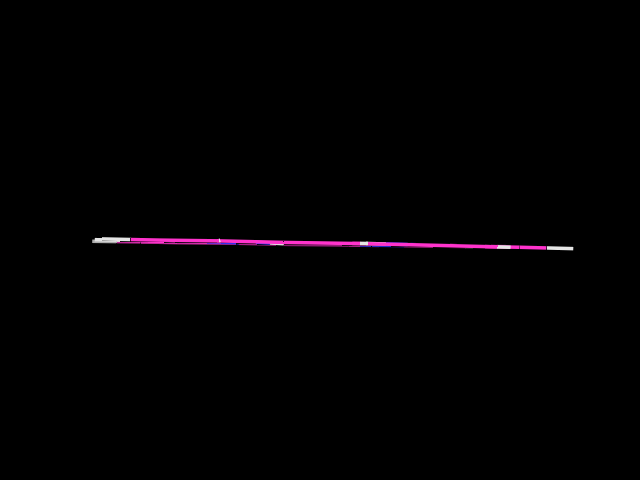
Опитимизированная PM6 геометрия порфирина соответствует реальности.
Рассчитаем возбужденные состояния порфирина и на основе этих данных прикинем спектр поглощения молекулы.
##возбужденный порфирин
def mopac_v(smiline,typea,name):
mol=pybel.readstring('smi',smiline)
mol.addh()
mol.make3D(steps=1000)
mop=mol.write(format='mopin',filename=name+typea+'_v2.mop',opt={'k':typea+' CHARGE=%d' %mol.charge}, overwrite=True)
addfile = open(name+typea+'_v2.mop','a')
addfile.write('\n'+'cis c.i.=4 meci oldgeo'+'\n')
addfile.close()
mopac_command = '/home/preps/golovin/progs/mopac/MOPAC2016.exe '+name+typea+'_v2.mop'
system(mopac_command)
opt=pybel.readfile('mopout',name+typea+'_v2.out')
for i in opt:
print i
i.write(format='pdb',filename=name+typea+'_v2.pdb', overwrite=True)
mopac_v(porph,'AM1','porph')
На основе приведенных в конце файла значений энергий для электронных переходов и формулы Е=h*v посчитан спектр:
outfile = open('porphPM6_v2.out', 'r')
lines = outfile.readlines()[-21:-13]
outfile.close()
energies = [float(line.split()[1]) for line in lines]
print energies, 'eV'
wavelengths = [1239.84193/e for e in energies]
frequencies = [e/4.135667516 for e in energies]
print 'frequncy,THz\t wavelength,nm\tenergy,eV'
for i in range(len(energies)):
print '%.4f \t %.4f \t %.4f' % (frequencies[i]*1000, wavelengths[i], energies[i])
Тиминовые димеры.
td_pdb = './td.pdb'
td = m3d(td_pdb)
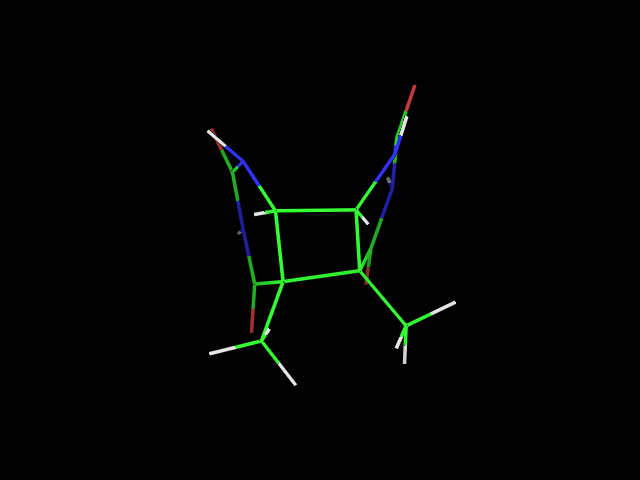
Начальная геометрия.
mop_td=td.write(format='mopin',filename='td.mop',opt={'k':'PM6 CHARGE=0 XYZ'}, overwrite=True)
%%bash
export MOPAC_LICENSE='/home/preps/golovin/progs/mopac/'
/home/preps/golovin/progs/mopac/MOPAC2016.exe td.mop
opt=pybel.readfile('mopout','td.out')
for i in opt:
print i
i.write(format='pdb',filename='td_01.pdb', overwrite=True)
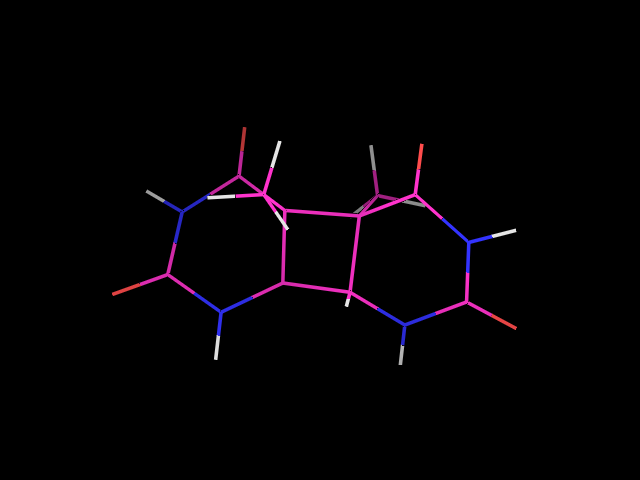
После оптимзации с зарядом 0
opt=pybel.readfile('mopout','td.out')
for i in opt:
print i
mop_td2=i.write(format='mopin',filename='td2.mop',opt={'k':'PM6 CHARGE=2 XYZ'}, overwrite=True)
%%bash
export MOPAC_LICENSE='/home/preps/golovin/progs/mopac/'
/home/preps/golovin/progs/mopac/MOPAC2016.exe td2.mop
opt=pybel.readfile('mopout','td2.out')
for i in opt:
print i
i.write(format='pdb',filename='td_02.pdb', overwrite=True)
После оптимзации с зарядом +2
opt=pybel.readfile('mopout','td2.out')
for i in opt:
print i
mop_td3=i.write(format='mopin',filename='td3.mop',opt={'k':'PM6 CHARGE=0 XYZ'}, overwrite=True)
%%bash
export MOPAC_LICENSE='/home/preps/golovin/progs/mopac/'
/home/preps/golovin/progs/mopac/MOPAC2016.exe td3.mop
opt=pybel.readfile('mopout','td3.out')
for i in opt:
print i
i.write(format='pdb',filename='td_03.pdb', overwrite=True)
После повторной оптимзации с зарядом 0. Как видно из рисунка, тиминовый димер не вернулся в исходную конформацию и развалился на две отдельные молекулы.
Энергии после оптимизаций:
Заряд 0: -560.52178 KJ/MOL
Заряд +2: 1226.23201 KJ/MOL
Заряд 0: -712.72844 KJ/MOL
Ab inito вычисления: Gamess US
Были построены и оптимизированны геометрии азулена и нафталена.
def orca_opt(smiline,name):
mol=pybel.readstring('smi',smiline)
mol.addh()
mol.make3D(steps=1000)
mop=mol.write(format='orcainp',opt={'k':'!HF RHF OPT 6-31G'}, overwrite=True)
out = mop.replace('! insert inline commands here ', '!HF RHF OPT 6-31G')
outfile = open('./'+name+'_opt_orca.inp', 'w')
outfile.write(out)
outfile.close()
orca_command = '/srv/databases/orca/orca '+name+'_opt_orca.inp'+' | tee orca-opt_'+name+'.log '
system(orca_command)
# opt=pybel.readfile('orca',name+typea+'.out')
# for i in opt:
# print i
# i.write(format='pdb',filename=name+typea+'.pdb', overwrite=True)
azu = 'C1=CC=C2C=CC=C2C=C1'
orca_opt(azu,'azu')
nap = 'C1=CC=C2C=CC=CC2=C1'
orca_opt(nap,'nap')
После этого были посчитаны энергии 2 разными способами:
def orca_hf(name):
mol=pybel.readfile('orca','orca-opt_'+name+'.log')
for i in mol:
print i
mop=i.write(format='orcainp',opt={'k':''}, overwrite=True)
out = mop.replace('! insert inline commands here ', '!HF RHF 6-31G')
outfile = open('./'+name+'_hf_orca.inp', 'w')
outfile.write(out)
outfile.close()
orca_command = '/srv/databases/orca/orca '+name+'_hf_orca.inp'+' | tee orca-hf_'+name+'.log '
system(orca_command)
def orca_dft(name):
mol=pybel.readfile('orca','orca-opt_'+name+'.log')
for i in mol:
print i
mop=i.write(format='orcainp',opt={'k':''}, overwrite=True)
out = mop.replace('! insert inline commands here ', '!DFT RHF 6-31G')
outfile = open('./'+name+'_dft_orca.inp', 'w')
outfile.write(out)
outfile.close()
orca_command = '/srv/databases/orca/orca '+name+'_dft_orca.inp'+' | tee orca-dft_'+name+'.log '
system(orca_command)
orca_hf('azu')
orca_dft('azu')
orca_hf('nap')
orca_dft('nap')
Полученные энергии:
| Метод | E Naphthalene | E Azulene | ΔE , kCal/mol |
| HF | -119.22071755 Eh = -74812.145738927 kCal/mol | -383.02130965 Eh = -240349.4141593 kCal/mol | 165537 kCal/mol |
| DFT | -130.6359515382 Eh = -81975.3147444394 kCal/mol | -382.007338449 Eh = -239713.275215 kCal/mol | 157737 kCal/mol |
Из эксперимента известно, что энергия изомеризации нафталина в азулен составляет 35.3±2.2 kCal/mol. А посчиталась ересь. Возможно, потому что при расчете нафталена Хартри-Фоком ORCA крашнулась, и при расчете в DFT обеих молекул тоже.