Выравнивание пространственных структур
Построение выравнивания вручную, используя наложенные 3D-структуры
Был проанализирован файл с четырьмя, наложеннными друг на друга, участками последовательностей. При помощи GeneDoc было построено выравнивание,
исходя из того, что в одной колонке могут быть лишь те остатки, которые реально выравниваются в пространстве. Если же, к примеру, два участка не совмещены, то они
записываются в выравнивание последовательно. Полученное выравнивание можно посмотреть здесь.
Далее на основе данного выравнивания было построено ещё одно, в котором за счет снижения пространственной достоверности повысилась
схожесть последовательностей и уменьшилось количество гэпов. Чтобы показать, где реально выравниваются последовательности, была добавлена строка Cons:
буква H означает выравнивание всех последовательностей, М - только трех.
Сравнение пространственного выравнивания с обычным, построенным программой ClustalW
Выравнивание последовательностей было так же загружено в GeneDoc. Сначала сравнивалась консервативность выравниваний:
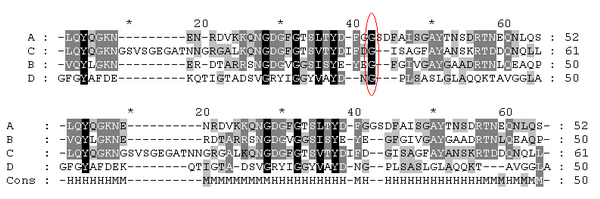
Как можно заметить в целом оба выравнивания очень похожи. ClustalW, призванный искать самые оптимистичные решения, лишь нашел ещё одну консервативную позицию - глицин 178 (нумерация по последовательности А). В пространстве эти четыре глицина выглядят так:
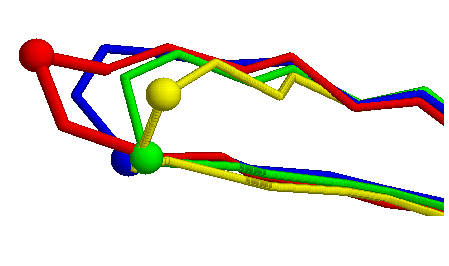
Очевидно, что выравниваются только два из них, что и отображено в нашем выравнивании.
Далее было проверено насколько хорошо ClustalW выровнял трехмерные структуры. Для этого его результат сравнивали с первым вариантом ручного выравнивания:

Оказалось, что ClustalW смог "увидеть" только одно серьезное различие в структуре - петлю цепи С (показана синим). Ещё два отклонения (оба в цепи D) он выравнял, не заметив. Желтым выделен нестандартный поворот, бронзой - альтернативный конец (впрочем это лишь кусок последовательности, так что "конец" - понятие условное). Сирень показывает позицию в которой все 4 последовательности совпадают, но, вероятней всего, цепь D в неё попала случайно (в структуре показаны шариками). Наконец, сложная ситуация с поворотом (изумруд) кажется попыталась разрешиться, но правильно были выровнены только два остатка: изолейцин и серин.
В целом результат неплохой, учитывая, что ClustalW обучен выравнивать лишь первичную структуру аминокислотной последовательности. А большое количество невыровненных им участков говорит о том, что третичная структура более консервативна, чем первичная.