Мини-обзор генома бактерии Pseudomonas brassicacearum
ОПИСАНИЕ
Бактерия P.brassicacearum - почвенная бактерия из семейства Pseudomonadaceae. Данная работа освещает вопросы качественного и количественного состава генома данного организма и носит, в основном, описательный характер.
КЛЮЧЕВЫЕ СЛОВА
Pseudomonas brassicacearum, Brassica napus, геном, протеом
ВВЕДЕНИЕ
Целью данного обзора является изучение генома и протеома Pseudomonas brassicacearum (рис. 1). Это грамотрицательная почвенная бактерия, впервые выделенная из ризосферы масличного рапса Brassica napus, от которой она и получила свое название. Клетки бактерий являются палочками по морфологии (1.0–1.5 μm в длину, около 0.5 μm в диаметре).[1]
Классификация:
- Phylum Proteobacteria
- Class Gammaproteobacteria
- Order Pseudomonadales
- Family Pseudomonadaceae
- Genus Pseudomonas
- Pseudomonas brassicacearum
Наиболее известные штаммы P.brassicacearum продуцируют антимикробные соединения и обладают активностью в отношении фитопатогенных микробов.[2]
МЕТОДЫ
Информация о протеоме бактерии была получена из базы данных NCBI Genome[4]. В работе использовались умения работы с сервисом Google Sheets. При создании таблицы в Google Sheets. При создании таблицы в Google Sheets использовались такие возможности ПО, как возможность импортировать файл *feature_table.txt, его разбиение по столбцам, форматирование различных спецсимволов в ячейках, форматирование диапазона ячеек, сортировка столбцов и строк, использование фильтров по значениям. Использование метода COUNTIFS для подсчета количества ячеек, соответствующих заданному условию (длина белка больше или равна нижней границы интервала и меньше его верхней границы). Для подсчета средней длины белка в протеоме использована функция AVERAGE с форматированием результирующей ячейки для округления значения. Минимальное и максимальное значения длин продуктов генов было найдено при помощи функций MAX и MIN соответственно. Использованы методы работы с файлами, словари, реализация циклов в Python для подсчета числа нуклеотидов, частоты кодонов, GC-skew.
РЕЗУЛЬТАТЫ
Общая информация:
1) Общий объем генома - 6738544 пар оснований, что входит в рамки обычной длины прокариотического генома. В Таблице 1 приведено количество встреченных нуклеотидов и частота. Количество нуклеотидов С приблизительно равно количеству G, а количество А - количеству Т. Из чего можно сделать вывод, что для последовательности соблюдается правило Чаргаффа.
2) GC-состав равен 0.6083 и является достаточно высоким для типа Gammaproteobacteria.[5]
3) Количество генов в геноме P.brassicacearum - 6100. Из них кодирующих белки генов - 5883. Распределение генов по функциям можно увидеть в Таблице 2.
4) Число гипотетических белков в геноме “hypothetical protein” бактерии - 677. Процентное содержание - 11,5 % от общего количества кодирующих белки последовательностей.
5) Рибосомных белков в протеоме P.brassicacearum 60 штук. Из них на “+” - цепи - 13, на “-”-цепи - 47. (Их список можно увидеть на листе “ribosomal prots” таблицы Genome features)
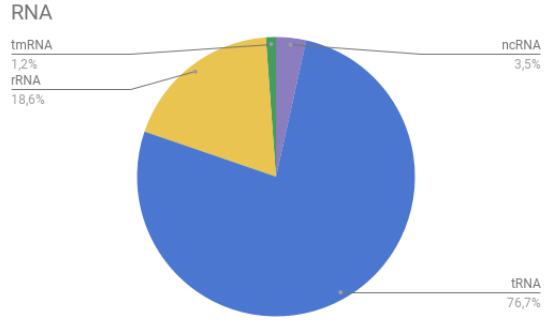
6) Всего генов, кодирующих РНК - 85, их распределение показано на рис. 2
| Нуклеотид | Количество | Частота |
|---|---|---|
| A | 1320561 | 0.1960 |
| C | 2050075 | 0.3042 |
| G | 2049305 | 0.3041 |
| T | 1318603 | 0.1957 |
| Что кодирует | Цепь | Количество | Всего |
|---|---|---|---|
| Белки | + | 2974 | 5883 |
| - | 2909 | ||
| Псевдогены | + | 23 | 45 |
| - | 22 | ||
| Какую-либо РНК | + | 27 | 86 |
| - | 59 |
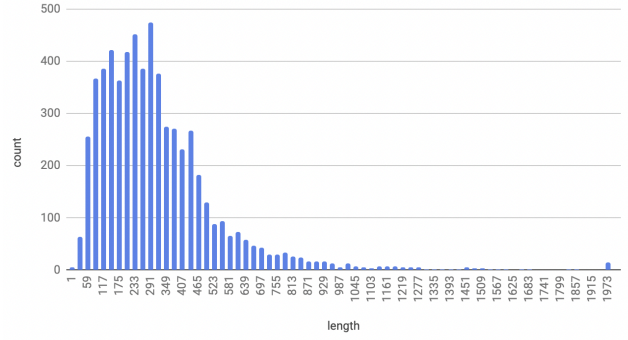
Гистограмма длин белков
Гистограмма длин белков представлена на Рисунке 3. Из гистограммы видно, что бактерия больше всего синтезирует те белки, конечная длина которых от 120 до 320. Средняя длина белка в протеоме 333. Наибольшая длина белка - 4910, наименьшая - 23. В протеоме P.brassicacearum имеется небольшое число больших белков, их длина превышает среднюю в 10 – 15 раз. Они относятся к классу вторичных метаболитов и могут отвечать за синтез противомикробных соединений.[6] Белки с наибольшей длиной перечислены в таблице 3.
| Белок | Длина |
|---|---|
| hemagglutinin repeat-containing protein | 4910 |
| non-ribosomal peptide synthetase | 4648 |
| LapA family giant adhesin | 4512 |
| filamentous hemagglutinin N-terminal domain-containing protein | 4434 |
| non-ribosomal peptide synthetase | 4328 |
| filamentous hemagglutinin family protein | 4188 |
| non-ribosomal peptide synthetase | 3401 |
| retention module-containing protein | 3347 |
Использование кодонов
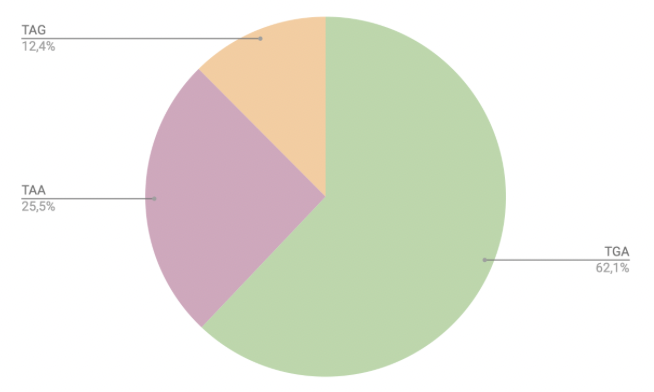
Распределение старт-кодонов приведено в таблице 4. Наиболее часто используемым старт-кодоном является ATG, также довольно часто встречаются кодоны TTG и GTG. Остальные кодоны встречаются значительно реже. Вероятно, такие старт-кодоны получаются в результате мутации и не влияют на инициацию транскрипции.На рисунке 4 показано распределение стоп-кодонов Стоп-кодом чаще всего является TGA, однако остальные кодоны (TAG и TAA) тоже довольно часто используются - в 37,9% случаев. В 18 генах стоп-кодон встречается не только в конце последовательности, в 16 случаях встречаются кодоны TGA и TAG, они кодируют аминокислоты селеноцистеин и пирролизин соотвественно и не являются терминирующими.[7]
| Кодон | Количество | Частота |
|---|---|---|
| ATG | 5308 | 0,895 |
| GTG | 398 | 0,067 |
| TTG | 153 | 0,026 |
| CTG | 19 | 0,003 |
| Другие | 50 | 0,008 |
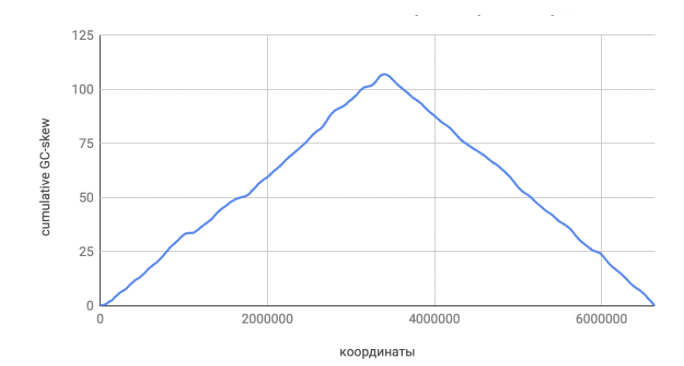
GC-skew
На рисунке 5 показан график GC-skew cumulative. Расчёт GC-skew в окне заданной ширины производится по формуле:
GC-skew = (G-C)/(G+C)
GC-skew cumulative для позиции считается как сумма всех GC-skew, посчитанных ранее. Точка минимума на графике соответствует началу репликации – oriC. Ее кордината - 0. Максимум на графике должен соответствовать точке терминации репликации – ter. Ее координата - 3400000
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
Геном, таблица со списком белков, графиками и расчетами, программы, использованные в данной статье, доступны по ссылке
БЛАГОДАРНОСТЬ
Автор выражает особую благодарность преподавателям информатики за предоставленные знания и важные советы по оформлению статьи.
СПИСОК ИСТОЧНИКОВ
1. Achouak, W. et al. Pseudomonas brassicacearum sp. nov. and Pseudomonas thivervalensis sp. nov., two root-associated bacteria isolated from Brassica napus and Arabidopsis thaliana. Int. J. Syst. Evol. Microbiol. 50, 9–18 (2000).
2. Nelkner, J. et al. Genetic Potential of the Biocontrol Agent Pseudomonas brassicacearum (Formerly P. trivialis) 3Re2-7 Unraveled by Genome Sequencing and Mining, Comparative Genomics and Transcriptomics. Genes 10, 601 (2019).
3. Zachow, C., Müller, H., Monk, J. & Berg, G. Complete genome sequence of Pseudomonas brassicacearum strain L13-6-12, a biological control agent from the rhizosphere of potato. Stand. Genomic Sci. 12, 1–7 (2017).
4. Index of /genomes/all/GCF/008/370/715/GCF_008370715.1_ASM837071v1. Index of /genomes/all/GCF/008/370/715/GCF_008370715.1_ASM837071v1.
5. Lightfield, J., Fram, N. R. & Ely, B. Across bacterial phyla, distantly-related genomes with similar genomic GC content have similar patterns of amino acid usage. PLoS One 6, e17677 (2011).
6. Rokni-Zadeh, H., Mangas-Losada, A. & De Mot, R. PCR detection of novel non-ribosomal peptide synthetase genes in lipopeptide-producing Pseudomonas. Microb. Ecol. 62, 941–947 (2011).
7. Pyrrolysine and Selenocysteine Use Dissimilar Decoding Strategies. J. Biol. Chem. 280, 20740–20751 (2005).