Cравнительный анализ канонической ДНК и стеблей тРНК
1. Генерация структуры DNA
Для генерации файлов были использованы следующие команды:
fiber -seq=GATCGATCGATCGATCGATC -a gatc-a.pdb
fiber -seq=GATCGATCGATCGATCGATC -b gatc-b.pdb
fiber -z gatc-z.pdb
Затем для поли-GC-Z-спирали я ввёла 20 пар оснований.
Полученные модели можно скачать здесь:
2. Сравнение модели с экспериментальными данными
Для этого задания я решила взять pdb файл B-формы DNA 1BNA. В B-форме бороздки определяются достаточно легко: большая бороздка значительно шире малой. На рисунке показан цитозин (мое основание для задания), находящийся в 3 положении цепи ДНК.Изображение получено при помощи MarvinSketch.
- В сторону большой бороздки обращены атомы: С3.C4, С3.C5, С3.C6, С3.N4.
- В сторону малой бороздки обращены атомы: С3.C2, С3.O2, С3.N1, С3.N3.
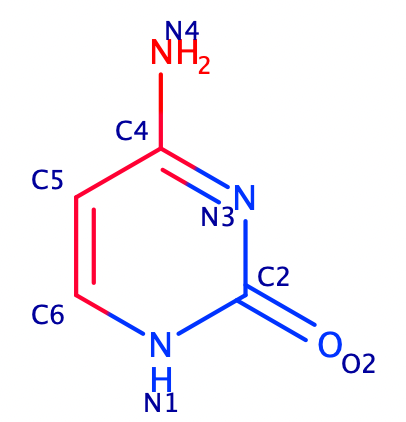 Рис. 1. Цитозин 1BNA. Красные атомы
Рис. 1. Цитозин 1BNA. Красные атомы
смотрят в сторону большой бороздки, а синие - в сторону малой.
Затем я изучила полученные при помощи fiber структуры, используя модуль nglview в Python. Полученные данные внесены в таблицу 1.
Таблица 1. Параметры разных структур DNA
|
A-форма |
B-форма |
Z-форма |
| Тип спирали |
Правая |
Правая |
Левая |
| Шаг спирали, Å |
28,03 |
33,75 |
42,50 |
| Число оснований на виток |
11 п.о. |
10 п.о. |
12 п.о. |
| Ширина большой (глубокой) бороздки, Å |
7,98 (A10:A - G25:B) |
17,21 (C12:A - A26:B) |
13,21 (C38:A - C46:B) |
| Ширина малой бороздки, Å |
16,81 (A10:A - G33:B) |
11,69 (T15:A - A30:B) |
7,20 (G37:A - G47:B) |
3. Структура RNA
В этом задании было необходимо определить параметры структуры РНК с помощью программ find_pair и analyze пакета 3DNA. Я рассматривал структуру с PDB ID: 1H4S. Чтобы можно было работать с пакетом, конвертируем в старый формат:
remediator --old ''1H4S.pdb'' > ''1H4S_old.pdb''
Затем определяем спаренные основания и положение спиралей в структуре:
find_pair -t 1H4S_old.pdb stdout | analyze
В результате получились файлы 1H4S_old.out и stacking.pdb.
Теперь рассмотрим неканонические пары в файле 1H4S_old.pdb. Всего в РНК присутствует 6 неканонических пар. Видно, что в RNA присутствуют псевдоуридин, 5-метилурацил.
Неканонические пары:
T:..49_:[..G]G-*---U[..U]:..65_:T
T:..54_:[5MU]t-**--G[..G]:..58_:T
T:..55_:[PSU]P-**+-G[..G]:..18_:T
T:..44_:[..G]G-**--A[..A]:..26_:T
T:..14_:[..A]A-**--U[..U]:...8_:T
T:..15_:[..G]G-**+-C[..C]:..48_:T
Координаты стеблей:
(4...7 - 66...69)
(49...53 - 61...65)
(38...44 - 26...32)
(10...13 - 22...25)
Дополнительные водородные связи:
T:..54_:[5MU]t-**--G[..G]:..58_:T
T:..55_:[PSU]P-**+-G[..G]:..18_:T
T:..14_:[..A]A-**--U[..U]:...8_:T
T:..15_:[..G]G-**+-C[..C]:..48_:T
T:..19_:[..G]G-----C[..C]:..56_:T
С помощью программы analyze пакета 3DNA было получено значение торсионных углов для выбранной тРНК, полученные данные были обработаны при поомощи Python. С результатами можно ознакомиться в таблице 2.
Таблица 2. Сравнение средних значений торсионных углов модели с теоретическими.
| Углы |
α |
β |
γ |
δ |
ε |
ζ |
χ |
| A-форма (теоретическая) |
62 |
173 |
52 |
88/3 |
178 |
-50 |
-160 |
| B-форма (теоретическая) |
63 |
171 |
54 |
123/131 |
155 |
-90 |
-117 |
| Цепь 1 (среднее) |
-10,9 |
49,3 |
45 |
86 |
-146,1 |
-63,2 |
-104,5 |
| Цепь 2 (среднее) |
-58,9 |
63,2 |
57,8 |
88 |
-149,4 |
-69,8 |
-157,1 |
Из таблицы видно, что полученные значения торсионных углов в молекуле сильно отличаются от "канонических", характерных для ДНК.