2. Поиск ДНК-белковых контактов в заданной структуре
В данном упражнении было необходимо определить следующие множества атомов в Jmol: кислорода 2'-дезоксирибозы (set1), кислорода в остатке фосфорной кислоты (set2), азота в азотистых основаниях (set3).
Для описания контактов разной природы был написан скрипт, в котором укзаано, какие атомы считать полярными и неполярными. Далее последовательно было использовано несколько команд select (атомы покрашены в цвета для удобства), определяющих следующие взаимодействия:
1) "полярные" (азот, кислород) атомы ДНК и белка, расположенные на расстоянии не более 3.5 Å;
2) "неполярные" (углерод, фосфор, сера) атомы ДНК и белка, расположенные на расстоянии не более 4.5 Å.
Результаты поиска приведены в таблице 2.
| Контакты атомов белка с |
Полярные |
Неполярные |
Всего |
| остатками 2'-дезоксирибозы |
2 |
8 |
10 |
| остатками фосфорной кислоты |
10 |
2 |
12 |
| остатками азотистых оснований со стороны большой бороздки |
8 |
14 |
22 |
остатками азотистых оснований со стороны малой бороздки |
0 |
0 |
0 |
Можно заметить, что среди контактов белка с остатками сахара преобладают неполярные; это можно объяснить тем, что в остатке дезоксирибозы больше углерода, чем кислорода. Аналогично объясняются и преимущественно полярные взаимодействия фосфатных остатков.
Далее нужно было получить схему ДНК-белковых контактов. Для этого я перевела PDB в старый формат записи и далее воспользовалась nucplot:
remediator --old 1MHD.pdb > 1MHD_old.pdb
nucplot 1MHD_old.pdb
Полученный документ в формате Postscript я перевела в JPG. Результаты на рис. 2.
На полученной схеме нужно было выбрать и показать с помощью Jmol:
-
Аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК - это Leu71 (как один из остатков с наибольшим числом взаимодействий - 2)
-
Аминокислотный остаток, по моему мнению, наиболее важный для распознавания последовательности ДНК. Я считаю, что это Arg74 цепи A, так как этот остаток, по-видимому, образует две водородные связи с Gua2004(D) (доноры водорода - атомы NH1 и NH2 остатка аргинина, акцепторы водорода - атомы N7 и O6 остатка дезоксигуанозинфосфата), а значит, это позволяет белку относительно надёжно и специфично узнавать определённое азотистое основание (гуанин).что может играть значительную роль в распознавании последовательности.
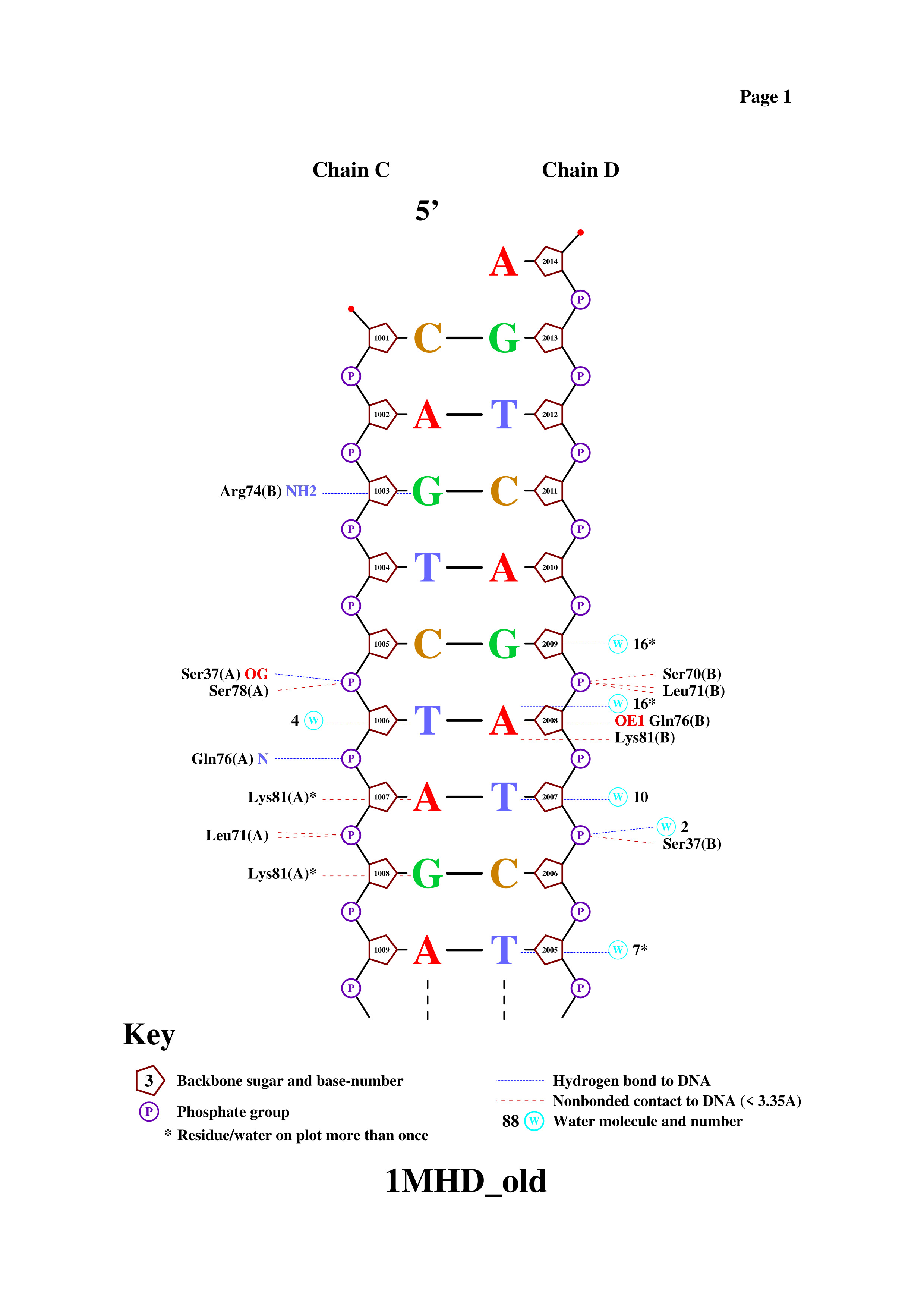 Рис. 2. Выдача nucplot.
Рис. 2. Выдача nucplot.
Сами контакты были визуализированны с помощью JMol.
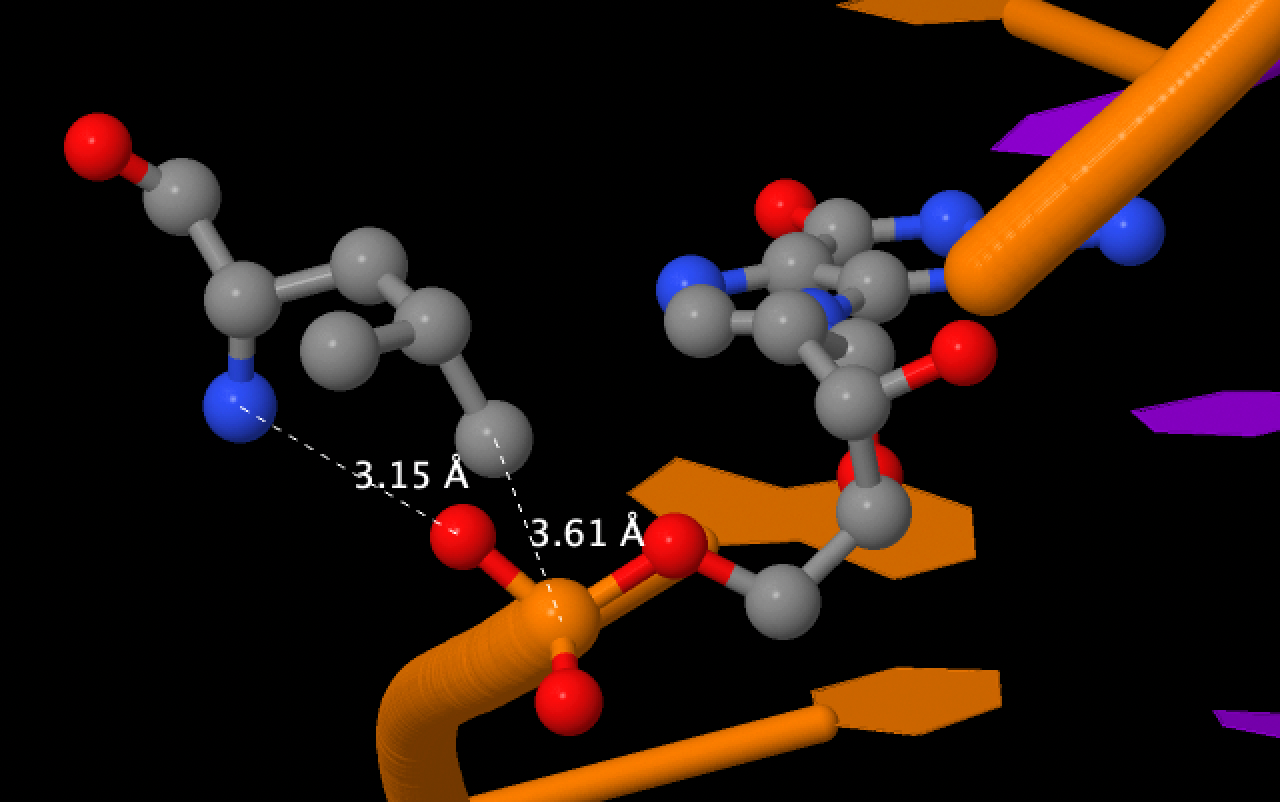 Рис. 3. Контакты с Leu71(A) с фосфатным остатком в составе ДНК.
Рис. 3. Контакты с Leu71(A) с фосфатным остатком в составе ДНК.
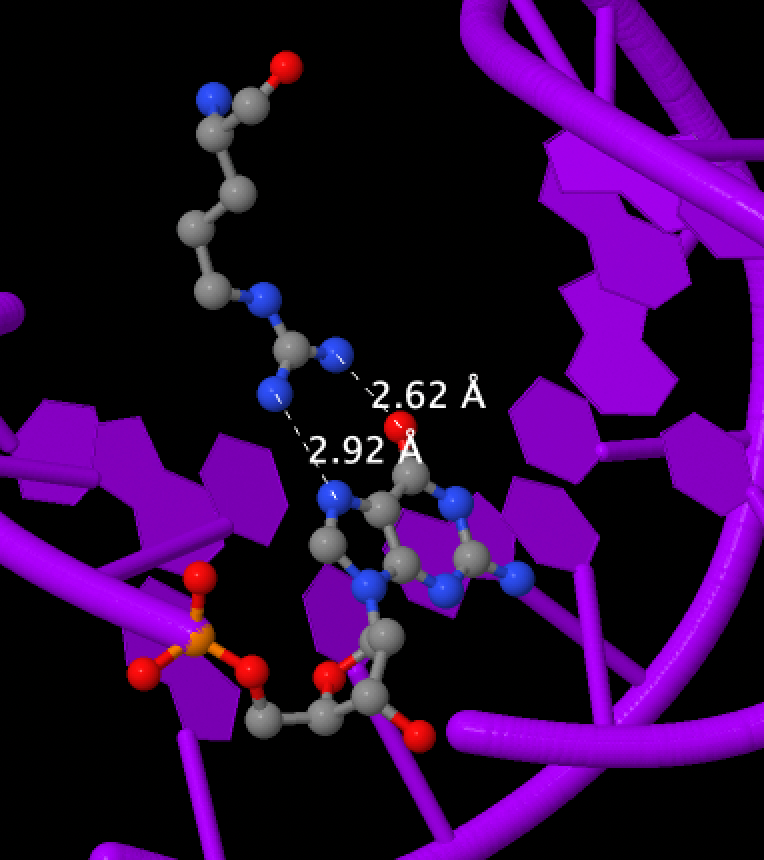 Рис. 4. Контакты с Arg74(A) с Gua2004(D) с возможностью образования двух водородных связей.
Рис. 4. Контакты с Arg74(A) с Gua2004(D) с возможностью образования двух водородных связей.