Нуклеотидный BLAST
Выбор сборки
Мной был выбран контиг 580 длиной 9734 bp. Он не помещен ни в одну из хромосом (unplaced scaffold). Идентификатор: NW_025964923
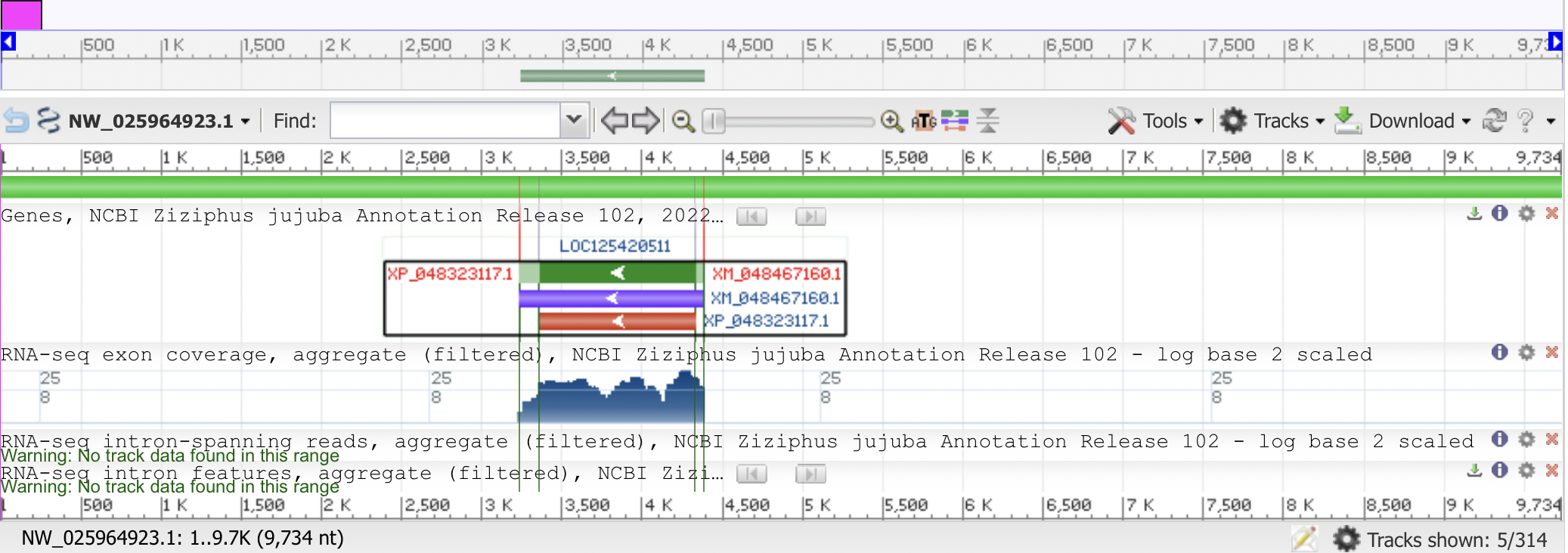 Рис. 1. Схема расположения генов на выборанном контиге
Рис. 1. Схема расположения генов на выборанном контиге
На схеме (Рис.1) видим, что зелёным отмечен сам ген (1 ген), светло-зелёным — 5'- и 3'-нетранслируемые области, фиолетовым — соответствующая мРНК (1 мРНК) и красным — кодирующая последовательность (1 CDS). Длина кодирующей последовательности: 975 нуклеотидов. Ген кодирует беллок, похожий на кислотную эндохитиназу.
Посмотреть файл в fasta-формате можно по ссылке.
Поиск в BLAST
Далее был проведен поиск последовательностей по выбранному контигу с использованием сервиса BLAST и алгоритмами blastn, megablast, blastx, tblastx и исключением некоторых таксонов.
Таблица 1. Сравнение алгоритмов BLAST
| Алгоритм поиска |
Выбор таксонов |
Длина слова |
Количество находок |
Таксономия находок |
Состав находок |
Пример использования алгоритма |
| blastn |
Я решила убрать таксон Двудольные (Magnoliopsida (taxid:3398)) на Spermatophyta blast падал почему-то |
11 |
500/500 - при попытке поставить большее кол-во находок выдает уведомление о превыешении лимита использования CPU |
Нашлись представители различных групп. 147 находок относятся к Костистым рыбам, 50 к Амниотам, 199 к Членистоногим. |
Во все выравнивания попали разные участки контига, трудно отследить зависимость, но могу отметить, что выравнивания с кодирующей последовательностью встречаются гораздо реже, чем с некодирующей. |
Можно использовать, чтобы определить принадлежность орагнизма к тому или иному таксону, а также, чтобы сравнивать геномы не близкородственных организмов. |
| megablast |
Я решила исключить таксон Семенные растения Spermatophyta (taxid:58024) |
28 |
11/1000 |
Получился довольно неожиданный разброс организмов. Беспозвоночные, Позвоночные, Кинетопластиды |
Для всех находок хорошо выровнялся участок длиной около 30-35 нуклеотидов в начале кодирующей последовательности контига. |
Осуществляет быстрый поиск среди очень похожих последовательностей. Мо |
| blastx |
Я решила исключить таксон Gunneridae (taxid:91827) (все таксоны выше по рангу, к сожалению, не позволяли провести поиск) |
6 |
714 |
Сразу заметна проблема поиска - все находки принадлежат только к таксону Magnoliopsida |
Среди находок много белков hevamine-A-like, это тоже гликозил гидролаза. Остальные также эндохитиназы или гипотетические белки. |
Определение кодирующих последовательностей |
| tblastp |
При всех попытках убрать какой-либо таксон (даже при поиске по любым таксонам) сайт падал с ошибкой нехватки вычислительных ресурсов для обработки запроса, в итоге NCBI сочли мои запросы подозрительными и сказали, что не будут временно осуществлять их :( |
Можно использовать для предсказания генов в последовательностях |
Поиск гомологов рРНК в геноме эукариот на основе гена E.coli
Сначала при помощи команды makeblastdb -in GCA_020796205.1_ASM2079620v1_genomic.fna -dbtype nucl была создана база данных на основе генома моего организма.
Для выполнения этой задачи выбран blastn, т.к. проводится поиск по нуклеотидным последовательностям, гены рРНК не являются белок-кодирующими.
Использованные команды
blastn -task blastn -evalue 0.05 -query rRNA1.txt -db GCA_020796205.1_ASM2079620v1_genomic.fna -out blast_rRNA1.txt -outfmt 7
blastn -task blastn -evalue 0.05 -query rRNA2.txt -db GCA_020796205.1_ASM2079620v1_genomic.fna -out blast_rRNA2.txt -outfmt 7
Параметр -evalue установлен, чтобы убрать плохие находки, параметр -outfmt для удобной выдачи в виде таблицы.
16S рРНК входит в состав малой субъединицы, 23S рРНК в состав большой субъединицы прокариотической рибосомы
В результате первого blast в топе находок (с наименьшим e-value) последовательности хлоропластного и митохондриального генома, что неудивительно, так как в хлоропластах и митохондриях находятся прокариотические рибосомы. Однако не могу не отметить, что в хлоропластной (CM036903.1) и митохондриальной (CM036902.1)последовательности отсутствуют какие-либо аннотированные гены рРНК. Для отстальных находок с малым e-value аннотированные гены рРНК также отсутствуют.
В результате второго blast в топе находок последовательность хлоропластного генома (CM036903.1) и митохондриального генома (CM036902.1), что тоже вполне естественно.
Сложно увидеть какие-либо закономерности в гомологии последовательностей, так как аннотация генома плоховата(