Для работы использованы гомологи АТФ-связывающей субъединицы протеазы Clp E.coli (fasta), принадлежащие бактериям, приведенным в таблице ниже.
| Название |
Мнемоника |
| Clavibacter michiganensis |
CLAMS |
| Corynebacterium diphtheriae |
CORDI |
| Corynebacterium efficiens |
COREF |
| Leifsonia xyli |
LEIXX |
| Nocardioides sp. |
NOCSJ |
| Rhodococcus jostii |
RHOJR |
| Rubrobacter xylanophilus |
RUBXD |
| Streptomyces avermitilis |
STRAW |
Составление списка гомологов
Для выполнения задания была создана локальная база данных по выбранным бактериям с помощью следующих команд:
cat CLAMS.fasta CORDI.fasta COREF.fasta LEIXX.fasta NOCSJ.fasta RHOJR.fasta RUBXD.fasta STRAW.fasta > proteomes.fasta
makeblastdb -dbtype prot -in proteomes.fasta -out bac_db
blastp -query clpx_ecoli.fasta -num_threads 4 -db bac_db -evalue 0.001 -out blast_res.txt (файл)
В результате поиска были получены следующие находки:
Blast hit Bit-score E-value
sp|Q820F8|CLPX_STRAW ATP-dependent Clp protease ATP-binding subun... 535 0.0
sp|Q0SGZ3|CLPX_RHOJR ATP-dependent Clp protease ATP-binding subun... 534 0.0
sp|A1SME0|CLPX_NOCSJ ATP-dependent Clp protease ATP-binding subun... 528 0.0
tr|Q1AVT0|Q1AVT0_RUBXD ATP-dependent Clp protease ATP-binding sub... 524 0.0
sp|Q8FN57|CLPX_COREF ATP-dependent Clp protease ATP-binding subun... 518 0.0
sp|Q6NFU7|CLPX_CORDI ATP-dependent Clp protease ATP-binding subun... 518 0.0
sp|Q6AFZ6|CLPX_LEIXX ATP-dependent Clp protease ATP-binding subun... 509 1e-180
sp|B0RAS4|CLPX_CLAMS ATP-dependent Clp protease ATP-binding subun... 491 2e-173
tr|Q1AU05|Q1AU05_RUBXD ATPase AAA-2 OS=Rubrobacter xylanophilus (... 52.0 4e-07
tr|Q0S6Y7|Q0S6Y7_RHOJR Chaperone protein ClpB OS=Rhodococcus jost... 47.8 9e-06
tr|Q8FMH5|Q8FMH5_COREF Putative endopeptidase Clp ATP-binding cha... 47.0 1e-05
tr|Q0S8C7|Q0S8C7_RHOJR ATP-binding subunit of ATP-dependent Clp p... 47.0 1e-05
tr|Q6NFB1|Q6NFB1_CORDI ATP-dependent Clp protease ATP-binding sub... 45.8 4e-05
tr|Q1AY82|Q1AY82_RUBXD ATPase AAA-2 OS=Rubrobacter xylanophilus (... 43.5 2e-04
tr|Q6ACQ0|Q6ACQ0_LEIXX ATP-dependent zinc metalloprotease FtsH OS... 43.1 2e-04
tr|Q82QV8|Q82QV8_STRAW Putative AAA family ATPase OS=Streptomyces... 41.6 4e-04
tr|A1SDV1|A1SDV1_NOCSJ ATP-dependent zinc metalloprotease FtsH OS... 42.0 5e-04
tr|Q82EE9|Q82EE9_STRAW ATP-dependent zinc metalloprotease FtsH OS... 41.6 7e-04
tr|Q82EB8|Q82EB8_STRAW Putative ATP-dependent Clp protease OS=Str... 41.6 9e-04
tr|B0RHW4|B0RHW4_CLAMS ATP-dependent zinc metalloprotease FtsH OS... 41.2 0.001
Находками с лучшим E-value являются АТФ-зависимые протеазы Clp, а также все найденные белки обладают АТФазной активностью.
Реконструкция и визуализация
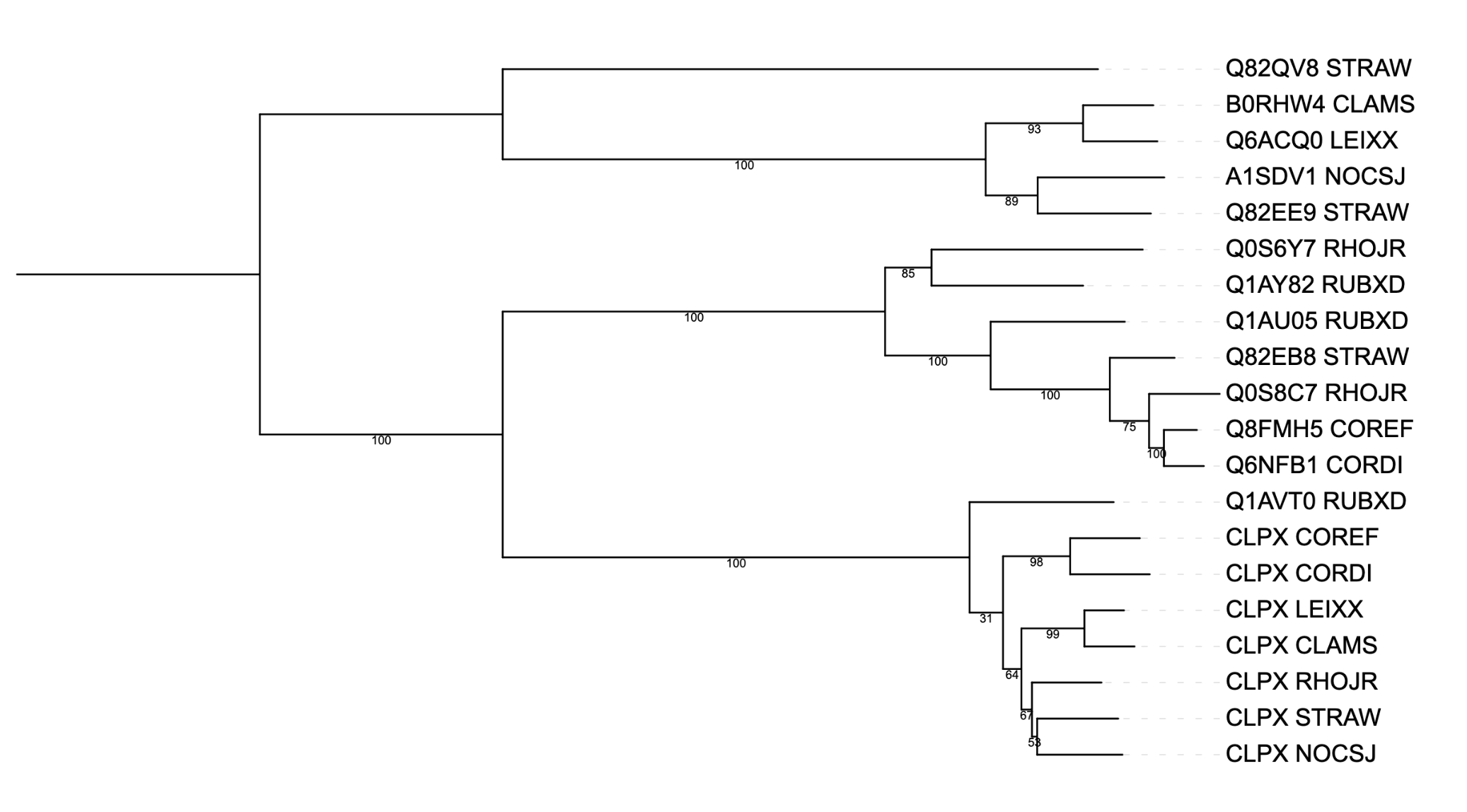 Рис. 1. Реконструированное дерево находок
Рис. 1. Реконструированное дерево находок
C помощью алгоритма FastMe было построено дерево. На дереве видны следующие пары предполагаемых ортологов: CLPX_CLAMS и CLPX_LEIXX, CPLX_CORDI и CLPX_COREF, B0RHW4_CLAMS и Q6ACQ0_LEIXX, и пары паралогов: Q82EE9_STRAW и Q82QV8_STRAW, CLPX_NOCSJ и A1SDV1_NOCSJ, CLPX_LEIXX и Q6ACQ0_LEIXX.
На полученном дереве хорошо обособлены группа ортологов Clp протеаз и группа цинковых металлопротеаз FtsH (Рис. 2):
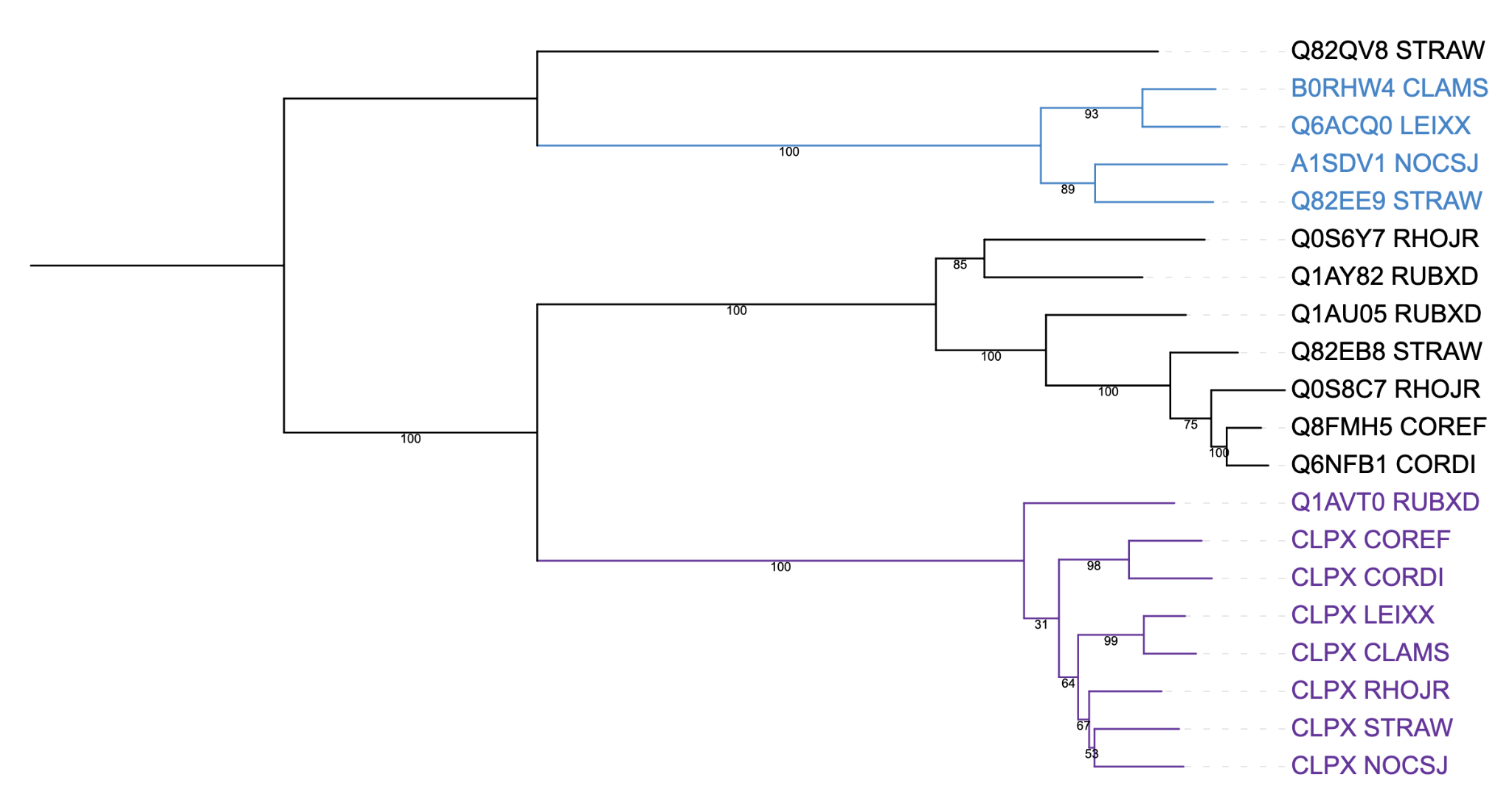 Рис. 2. Дерево с покрашенными ортологами. Фиолетовым - ортологичная группа Clp протеаз, голубым - группа цинковых металлопротеаз FtsH
Рис. 2. Дерево с покрашенными ортологами. Фиолетовым - ортологичная группа Clp протеаз, голубым - группа цинковых металлопротеаз FtsH
Схлопнем указанные ветви (Рис. 3). В голубую ветвь (цинковая металлопротеаза FtsH) вошло 4 листа, в фиолетовую — 8 листьев.
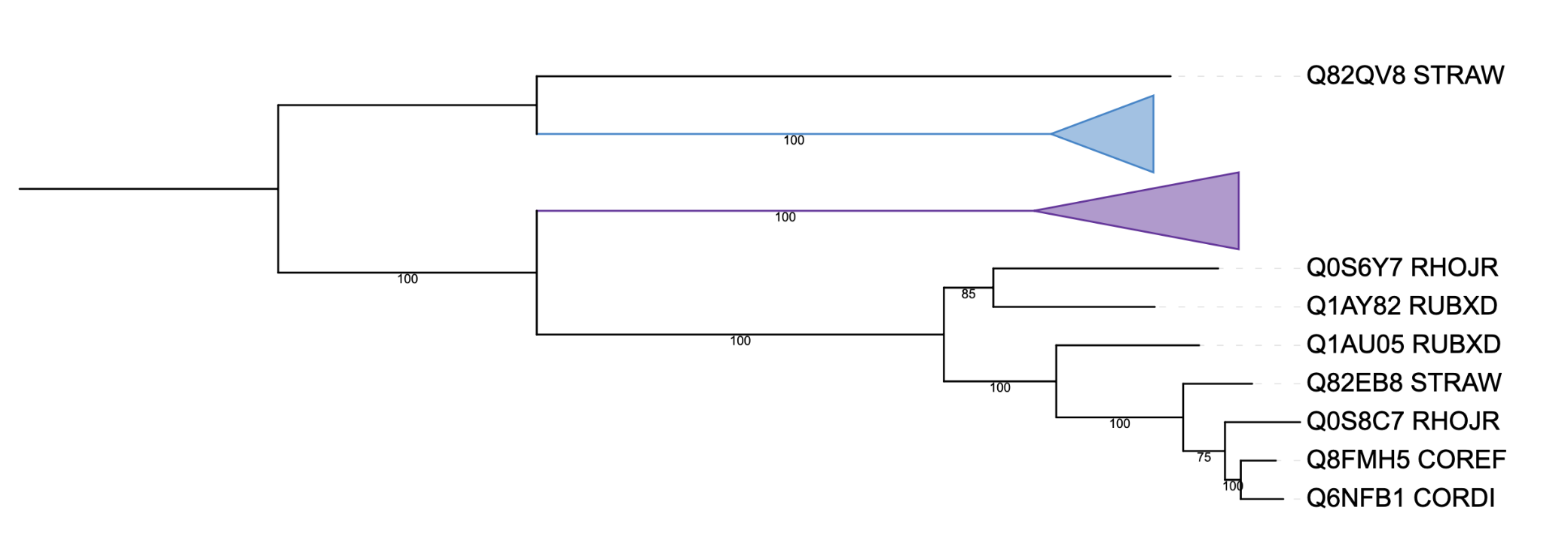 Рис. 3 Дерево со схлопнутыми ветвями.
Рис. 3 Дерево со схлопнутыми ветвями.
Клада Clp протеаз почти полностью соответствует исходной филогении бактерий, за ислючением положения ветви RHOJR, в изначальной топологии она образует нетривиальную ветвь CORDI, COREF, RHOJR} против {RUBXD, CLAMS, LEIXX, NOCSJ, STRAW} и соответствует кладе Mycobacteriales. Внутри клады металлопротеаз топология также совпадает с исходным деревом.