Часть первая. Выравнивания.
Для работы были выбраны шесть последовательностей гомологичных белков семейства у эукариот, архей и бактерий. В таблице ниже представлены данные
`
Таблица 1. Выбранные для задания белки
| Запись | Имя белка | Домен | Организм |
| B7J7X9 | Chaperone protein DnaK (HSP70) | Bacteria | Acidithiobacillus ferrooxidans |
| P41797 | Heat shock protein SSA1 | Eukaryota | Candida albicans |
| Q0W874 | Chaperone protein DnaK (HSP70) | Archaea | Methanocella arvoryzae |
| P9WMJ8 | Chaperone protein DnaK (HSP70) | Bacteria | Mycobacterium tuberculosis |
| Q3IUI0 | Chaperone protein DnaK (HSP70) | Archaea | Natronomonas pharaonis |
| P16474 | 78 kDa glucose-regulated protein homolog (GRP-78) | Eukaryota | Saccharomyces cerevisiae |
Hsp70 - семейство белков теплого шока, взаимодействующих с синтезируемой на рибосомах полипептидной цепью, предотвращая преждевременное неправильное
сворачивание незрелой полипептидной цепи, и участвующих в транспорте белка к органеллам.
Выравнивание, сделанное в JalView с помощью Tcoffee with Defaults, раскрашенное ClustalX с параметром Identity Threshold = 100%.
Используя опцию Tcoffee with Defaults, было смоделировано выравнивание последовательностей белков с раскраской по схеме ClustalX, при Identity Threshold = 100%.
Функциональная схожесть: изолейцин и валин - алифатические, гидрофобные остатки; аланин и глицин - алифатические, небольшие остатки; лейцин и изолейцин - изомеры.
Разметка: красным выделен пример абсолютно консервативных позиций (100%), зеленым - абсолютно функционально консервативных позиций, синим - позиции с гэпом.
Остальные данные получены с помощью программы infoalign пакета EMBOSS.
| Имя последовательности | Длина последовательности | Длина выравнивания | Количество гэпов | Длина гэпов | Идентичные | Сходные | Процент гэпов |
| P9WMJ8|DNAK_MYCTO | 625 | 761 | 16 | 139 | 428 | 61 | 43.76 |
| Q3IUI0|DNAK_NATPD | 656 | 761 | 16 | 105 | 402 | 93 | 47.17 |
| P16474|GRP78_YEAST | 682 | 761 | 11 | 79 | 395 | 80 | 48.10 |
| Q0W874|DNAK_METAR | 623 | 761 | 19 | 138 | 411 | 78 | 46.00 |
| B7J7X9|DNAK_ACIF2 | 634 | 761 | 20 | 127 | 417 | 77 | 46.20 |
| P41797|HSP71_CANAL | 656 | 761 | 12 | 105 | 390 | 82 | 48.75 |
Таблица 2
| Имя последовательности | Кол-во 100% консервативных позиций | Процент | Кол-во 70% консервативных позиций | Процент | Кол-во функционально консервативных позиций | Процент |
| P9WMJ8|DNAK_MYCTO | 172 | 22.6 | 315 | 43.2 | 270 | 35.5 |
| Q3IUI0|DNAK_NATPD | 172 | 22.6 | 307 | 43.1 | 270 | 35.5 |
| P16474|GRP78_YEAST | 172 | 22.6 | 293 | 41.4 | 270 | 35.5 |
| Q0W874|DNAK_METAR | 172 | 22.6 | 303 | 42.8 | 270 | 35.5 |
| B7J7X9|DNAK_ACIF2 | 172 | 22.6 | 304 | 42.9 | 270 | 35.5 |
| P41797|HSP71_CANAL | 172 | 22.6 | 288 | 41.8 | 270 | 35.5 |
Jalview проект
Часть вторая. Эволюция.
Для моделирования возникновения мутаций в белке был выбран гистон H2B.1 Saccharomyces cerevisiae. Текст скрипта bash, работающего на EMBOSS и добавляющего 7
случайных мутаций в каждом поколении. Для построения выравнивания я использовала программу JalView, алгоритм TcoffeeWS, раскраску ClusterX с
порогом идентичности 100%.
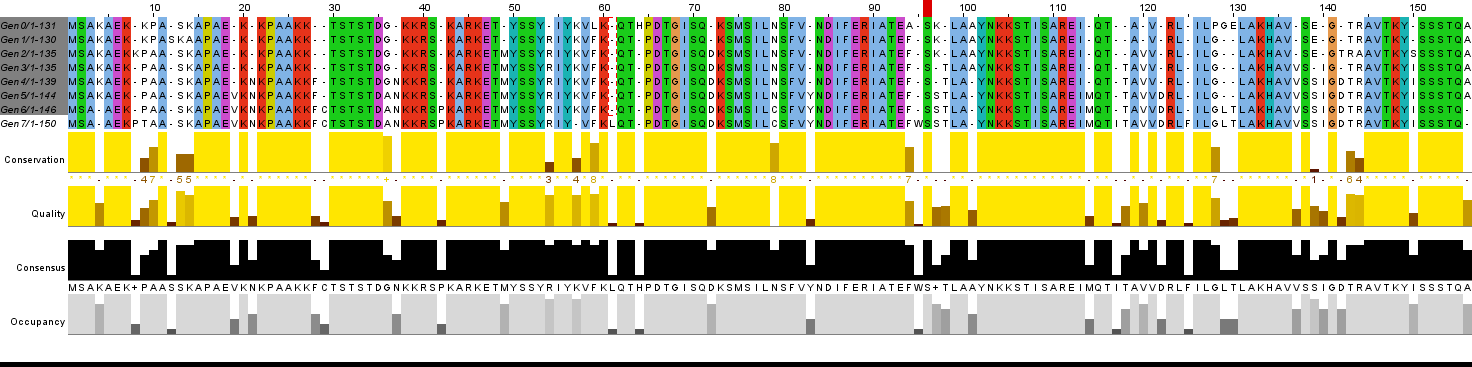
Исправленное вручную выравнивание
Рис 2. Исправленное выравнивание. Gen0- исходная последовательность, Gen1-Gen7 - видоизмененные потомки.
TCoffeeWS с 100% идентичность.
Мутации были воспроизведены таким образом, что только семь точечных мутаций произошло в каждом из семи рассматриваемых пооколений.
Информация о первых десяти позициях содержится в таблице 2.
Taблица 2. Данные о первых десяти мутациях в искусственно мутированном белке.
| Позиция |
Мутация |
Родительское поколение |
Дочернее поколение
|
| 4 |
Deletion of K |
Gen5 |
Gen6
|
| 8 |
Insertion of K |
Gen1 |
Gen2
|
| 9 |
Substitution of K by P |
Gen1 |
Gen2
|
| 10 |
Substitution of P by A |
Gen1 |
Gen2
|
| 12 |
Insertion of S |
Gen0 |
Gen1
|
| 19 |
Insertion of V |
Gen4 |
Gen5
|
| 21 |
insertion of N |
Gen3 |
Gen4
|
| 28 |
Insertion of F |
Gen3 |
Gen4
|
| 29 |
Insertion of C |
Gen5 |
Gen6
|
| 36 |
Substitution of G by A |
Gen4 |
Gen5
|