Поиск по сходству.
BLAST, E-VALUE
Задание 1. Проверка гомологичности белков, найденных поиском по сходству
Для выполнения задания была взята последовательность белка ANU26136.1 — селеноцистеин-специфичного фактора
элонгации трансляции из бактерии Planococcus sp. Cервис BLAST (разновидность blastp: protein—>protein) был запущен
на последовательностях из таксонов Euglena, Trichomonas, Sediminibacillus (для разнообразия значений E-value).
Распределение скоров выравниваний по выдаче отображено на Рисунке 1.
Рис. 1. Скоры выравниваний из выдачи blastp
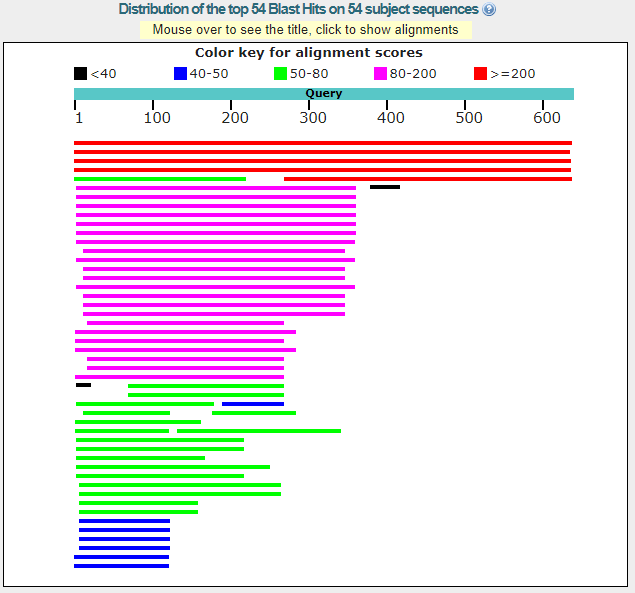
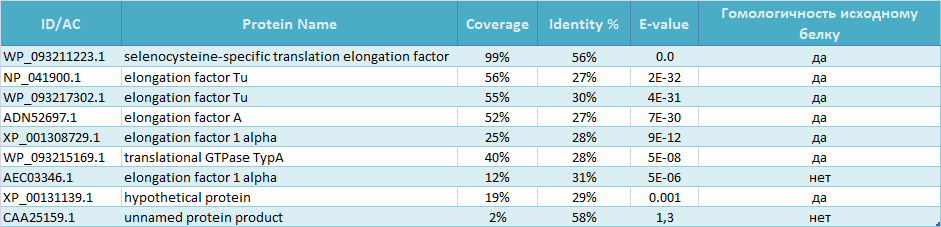
По итогам работы была составлена Таблица 1, в которой указаны параметры девяти отобранных находок из выдачи.
Последовательности отбирались так, чтобы в выборке встречались находки разного качества (из разных интервалов скоров), что хорошо отражено как в значениях E-value,
так и в проценте покрытия (Coverage) и проценте сходства (Identity %).
Таблица 1. Выборка из находок BLAST
Для определения гомологичности выровненные участки последовательностей были скачаны и выровнены с последовательностью исходного белка (первая в выравнивании, обозначим как #0).
Получившееся выравнивание приведено на Рисунке 2 (скачать jalview-проект).
Рисунок 2. Множественное выравнивание исходной последовательности с 9 находками
Для удобства анализа и наглядности представления результатов была добавлена разметка блоков выравнивания в поле BLOCKS.
Эти блоки выделялись без учета последовательностей из blast #4 (ADN52697.1), #7 (AEC03346.1), #9 (CAA25159.1). Изображение выравнивания
со скрытыми последовательностями #4,7,9 можно посмотреть здесь.
Таким образом, для оставшихся шести находок (#1-3, 5, 6, 8) мы наблюдаем как минимум шесть достоверных блоков выравнивания с большим количеством как абсолютно, так и функционально
консервативных колонок, и это дает основания говорить о гомологичности их исходной последовательности (#0).
Рассмотрим последовательность #4 (ADN52697.1). Блок 1 будет частично нарушен при добавлении её к шести последовательностям, по которым строилась разметка. Но, однако, ей можно без проблем расширить блоки 2-6,
что, на мой взгляд, является достаточно веским основанием считать ее также гомологичной исходной последовательности.
Последовательности же #7 и 9 при добавлении к вышеописанным шести нарушат все шесть блоков, кроме (опять же, частично) первого. Наличие единственного блока, пусть и достоверного, не может дать свидетельств в силу гомологичности
этих последовательностей нашей исходной, поэтому в соответсвующую колонку таблицы было занесено "нет", что подразумевает не доказательство негомологичности как таковой, а недостаток у нас данных "за" гомологичность.
Задание 2. Описание перестроек между парой белков с гомологичными участками
Для выполнения задания использовалась база данных Pfam. Выбранное доменное семейство — Vps53_N (PF04100), белки которого участвуют в процессах мембранного транспорта. Две выбранные архитектуры показаны на Рисунке 3.
Рис. 3. Выбранные архитектуры

В качестве представителей белков с данными архитектурами были выбраны белки A0A0V0UQY3_9BILA (с более простой II-ой архитектурой) и
A0A0V1MAD6_9BILA (с усложненной I архитектурой). Для этих последовательностей был запущен blastp в варианте "align two sequences".
Карта локального сходства (dot matrix) приведена на Рисунке 4.
Рис. 4. Карта локального сходства
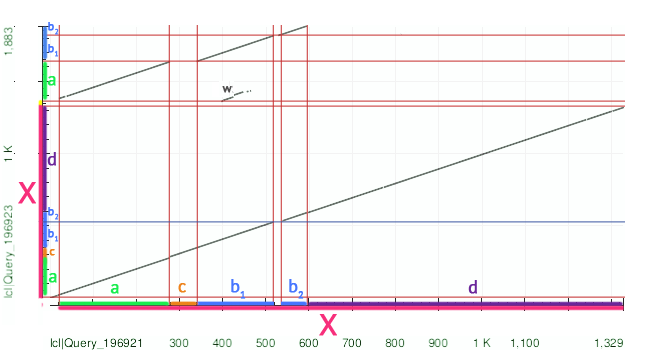
На карте одинаковые участки обозначены соответствующими цветами и буквенными обозначениями. На оси Х отложена последовательность с II архитектурой, на оси Y — с I-ой.
С учетом обозначений можем записать:
OX (II arch) A0A0V0UQY3_9BILA = X
Oy (I arch) A0A0V1MAD6_9BILA = X + {*} + a + b1 + b2,
где X = a + c + b1 + b2 + d; {*} — небольшая жёлтая вставка
Эволюционные события при переходе от посл-ти по Ох к посл-ти по Oy:
- делеция небольшого участка между b1 и b2;
- вставка небольшого участка {*} (жёлтым по Oy);
- дупликация участка a+b1+b2 (сперва, вероятно, дупликация a+c+b1+b2, затем делеция с);
- интересно отметить участок карты w: его наличие говорит о сходстве в последовательностях a и b1,
поэтому можно предполагать их эволюционную связь: одна могла развиться из другой.
Дополнение к вопросу о гомологии.
Я считаю возможным говорить о гомологии этих белков т.к. считаю их достаточно похожими для этого. Но гораздо более уверенно можно говорить о гомологичности доменов a и b1, b2.
|



