Предсказание вторичной структуры заданной тРНК
В этом задании было необходимо сравнить реальную вторичную структуру заданной тРНК, найденную с помощью find_pair, с предсказанными
структурами.
Поиск инвертированных повторов проводился с помощью программы einverted из пакета EMBOSS,
которая получает на вход последовательность нуклеотидов, а на выходе дает файлы sequence.fasta (содержит информацию о найденных комплементарных
участков) и sequence.inv (содержит информацию о предполагаемых водородных связях на этих участках).
При использовании программы с параметрами по умолчанию, файл sequence.fasta оказался пустым, поэтому понадобилось подбирать параметры вручную.
Наиболее значимым параметром оказался Minimum score threshold, который по умолчанию был
равен 50.
Наиболее близкие к реальной структуре предсказания были получены при threshold 14 и меньше. Наилучший результат, которого
удалось достичь - полное предсказание для одного из стеблей и частичное, с несовпадением конца и начала, для другого.
Далее была использована программа RNAfold из пакета Viena Rna Package, которая
реализует алгоритм Зукера. RNAfold принимает на вход последовательность РНК и
рассчитывает вторичную структуру РНК с минимальной свободной энергией.
Точки обозначают неспаренные нуклеотиды, круглые скобки - спаренные.
Квадратные и фигурные скобки обозначают взаимодействия, формирующие псевдоузлы.
Для моей структуры RNAfold сработала достаточно хорошо с первого раза. Были предсказаны 3 из 4 стеблей и один из стеблей был предсказан
со сдвигом на один нуклеотид.
| Реальная и предсказанная вторичные структуры тРНК из файла 1i9v.pdb |
|---|
| Участок структуры | Позиции в структуре (результаты find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера |
|---|
| Акцепторный стебель | 5'-1-7-3'
5'-66-72-3'
всего 7 пар |
предсказано 0 пар из 7 реальных | предсказано 7 пар из 7 реальных, стебель предсказан полностью |
| D-стебель | 5'-10-13-3'
5'-22-25-3'
всего 4 пары |
предсказано 0 пар из 4 реальных | предсказано 4 пары из 4 реальных, стебель предсказан полностью |
| T-стебель | 5'-49-53-3'
5'-61-65-3'
всего 5 пар |
предсказано 5 пар из 5 реальных, стебель предсказан полностью, начало и конец совпадают |
предсказано 4 пары из 5 реальных + одна лишняя пара, начало и конец стебля не совпадают |
| Антикодоновый стебель | 5'-40-44-3'
5'-26-30-3'
всего 5 пар |
предсказано 5 пар из 5 реальных, но начала и концы стеблей в реальной и
предсказанной структурах не совпадают, предсказано 5 лишних пар | предсказано 5 пар из
5 реальных, стебель предсказан полностью |
| Общее число канонических пар нуклеотидов | 23 | 13 | 23 |
Предсказанная вторичная структура тРНК с минимальной свободной энергией, RNAfold
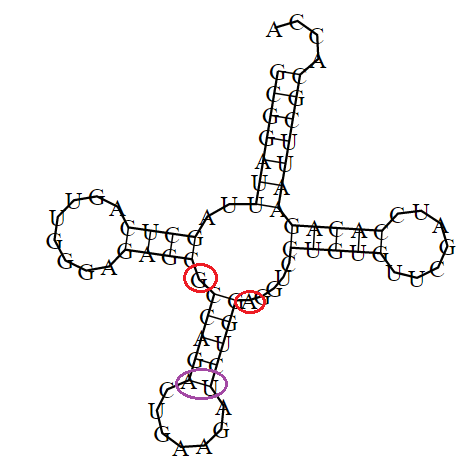
Красным отмечена недостающая пара, фиолетовым - лишняя.
Поиск ДНК-белковых контактов в структуре 1MDM
В упражнении 1 было необходимо задать множества атомов кислорода 2'-дезоксирибозы,
атомов кислорода в остатке фосфорной кислоты и атомов азота в азотистых основаниях
с помощью команды define, а затем создать скрипт, последовательно
отображающий изображения всего ДНК-белкового комплекса, только ДНК в проволочной модели и
той же модели, но с выделенными шариками множествами атомов set1, set2 и set3.
Результаты работы представлены в скрипте.
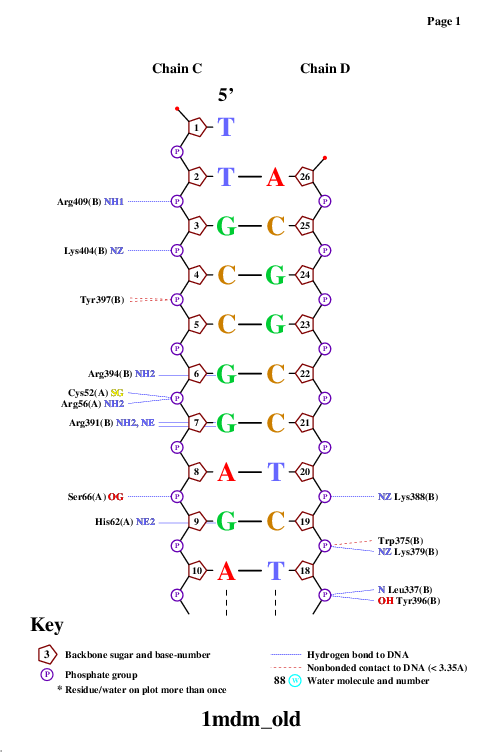
В следующем задании было необходимо найти ДНК-белковые контакты в заданной
структуре (PDB ID: 1MDM) и сравнить количество контактов разной природы.
Полярными считались атомы кислорода и азота, а неполярными - атомы углерода, фосфора и серы.
Под полярным контактом понималась ситуация, когда расстояние между полярным атомом белка и
полярным атомом ДНК меньше 3.5A. Аналогично, под неполярным контактом
подразумевалась пара неполярных атомов на расстоянии меньше 4.5A.
Текст скрипта
Результаты работы представлены в таблице.
| Контакты атомов белка с: | Полярные | Неполярные | Всего |
|---|
| остатками 2'-дезоксирибозы | 7 | 51 | 58 |
| остатками фосфорной кислоты | 17 | 16 | 33 |
| остатками азотистых оснований со стороны большой бороздки | 3 | 16 | 19 |
| остатками азотистых оснований со стороны малой бороздки | 4 | 9 | 13 |
Таким образом, наибольшее количество контактов в заданном комплексе реализуется
между белком и остатками 2'-дезоксирибозы, также много контактов с остатками фосфорной кислоты.
На основании данных таблицы, мы можем сделать вывод, что белок
наиболее охотно контактирует с остовом ДНК, и менее охотно - с остатками азотистых оснований.
Скорее всего это объясняется пространственным расположением.
Далее я получила схему ДНК-белковых контактов в комплексе 1MDM с помощью nucplot.
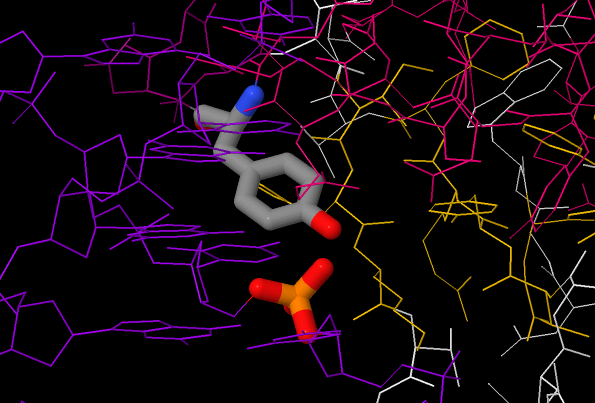
На картинке видно, что наибольшее количество связей образует tyr397.
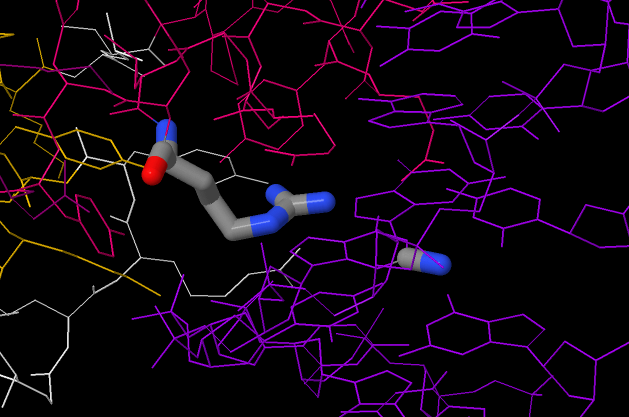
В качестве распознавателя, возьмем аминокислоту, которая непосредственно связывается с азотистым основанием, то есть arg394.
В выводе было 3 изображения, однако 2 из них приходились на другие цепи белка.
Рис. 1. Связывание arg394 с N2 гуанина. (картинка сверху)
*В правой части находится гуанин, который как раз и участвует в образовании контакта с аргинином.
Рис. 2. Связывание tyr397 с фосфатом. (картинка снизу)
*В нижней части находится фосфат, который участвует в образовании контактов с тирозином.



© Raldugina Vasilisa 2016 |