ВЫРАВНИВАНИЕ ГЕНОМОВ
АНАЛИЗ КРУПНЫХ ГЕНОМНЫХ ПЕРЕСТРОЕК
В данном практикуме рассматривались геномы бактерии Streptococcus suis
. Streptococcus suis - вид грамположительных бактерий. Бактерия
эндемична почти во всех странах с обширной свиноводческой промышленностью. S.
suis способна передаваться людям от свиней.
Естественной средой S. suis у свиней являются верхние дыхательные пути, особенно миндалины и полости носа,
а также пищевые и половые пути.
В Таблице 1 приведена основная информация о трех геномах Streptococcus suis, использовавшихся в построении выравнивания.
Таблица 1.
| ID | Штамм | Длина генома | Тип |
| NC_012926.1 | BM407 | 2146229 bp | кольцевой |
| CP002465.1 | JS14 | 2137435 bp | кольцевой |
| CP002640.1 | SS12 | 2096866 bp | кольцевой |
Данные хромосомы были выравнены при помощи blast2seq, алгоритма blastn. Параметры задавались по умолчанию. Ниже представлены
карты локального сходтва для трех выравниваний геномов:
Рис.1 Карта локального сходства геномов NC_012926.1 и CP002465.1
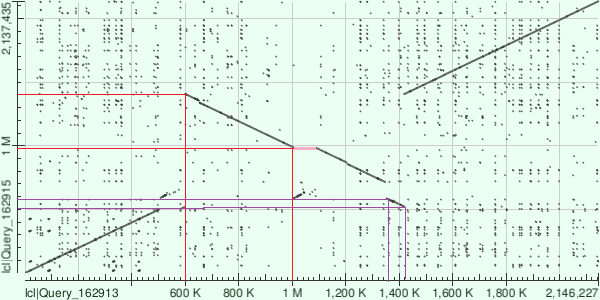 Рис.2 Карта локального сходства геномов CP002640.1 и NC_012926.1
Рис.2 Карта локального сходства геномов CP002640.1 и NC_012926.1
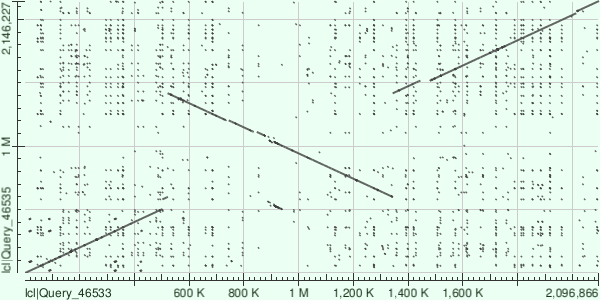 Рис.3 Карта локального сходства геномов CP002640.1 и CP002465.1
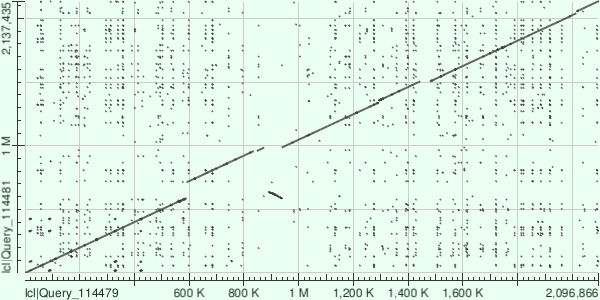
Рис.3 Карта локального сходства геномов CP002640.1 и CP002465.1
Ниже в Таблице 2 представлена основаная информация о трех построенных выравниваниях:
| Выравнивание | NC_012926.1 - CP002465.1 | CP002640.1 - NC_012926.1 | CP002640.1 - CP002465.1
|
| Ident% | 99% | 99% | 99% |
| E-value | 0.0 | 0.0 | 0.0 |
| Max score | 4.520e+05 | 3.196e+05 | 3.307e+05 |
| Query cover | 95% | 95% | 96% |
| Число находок | 2368 | 2314 | 2391 |
| Карта |
 |
 |
 |
| E-value | 0.0 | 0.0 | 0.0 |
| Max score | 4.520e+05 | 3.196e+05 | 3.307e+05 |
| q. start - q. end | 1770388 - 2015764 | 1780714 - 1954414 | 1079423 - 1258629 |
АНАЛИЗ КРУПНЫХ ГЕНОМНЫХ ПЕРЕСТРОЕК
Рис.1 Карта локального сходства геномов NC_012926.1 и CP002465.1
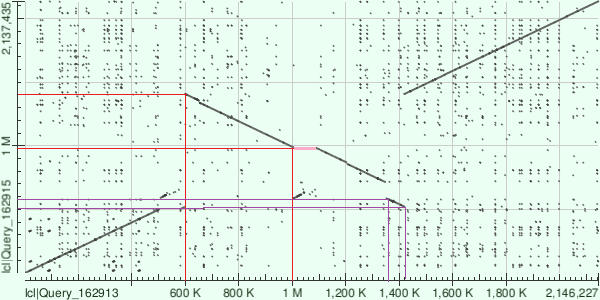 На данной карте по горизонтали расположен геном NC_012926.1 (ось Х, геном 1), а по вертикали CP002465.1 (ось Y, геном 2).
Участок, выделенный красным повернут на 180 градусов, относительно второго генома.
То есть в одном из геномов произошла инверсия данного участка.
Участок, выделенный розовым отсутствует в геноме, располагающемся на оси X, что говорит либо о делеции, либо о вставке в геноме по оси Y.
Участок, выделенный фиолетовым располагается в геноме 1 раньше, чем в геноме 2, что говорит о транслокации участка в одном из геномов.
Видно, что гомологичные участки занимют большую часть геномов.
На данной карте по горизонтали расположен геном NC_012926.1 (ось Х, геном 1), а по вертикали CP002465.1 (ось Y, геном 2).
Участок, выделенный красным повернут на 180 градусов, относительно второго генома.
То есть в одном из геномов произошла инверсия данного участка.
Участок, выделенный розовым отсутствует в геноме, располагающемся на оси X, что говорит либо о делеции, либо о вставке в геноме по оси Y.
Участок, выделенный фиолетовым располагается в геноме 1 раньше, чем в геноме 2, что говорит о транслокации участка в одном из геномов.
Видно, что гомологичные участки занимют большую часть геномов.
Рис.2 Карта локального сходства геномов CP002640.1 и NC_012926.1
 На карте по горизонтали расположени геном CP002640.1, по вертикали NC_012926.1.
Помимо этого на карте можно наблюдать инверисию крупного участка приблизительно с 550 тыс. нуклеотида по 1350 тыс. (по горизонтали).
На карте по горизонтали расположени геном CP002640.1, по вертикали NC_012926.1.
Помимо этого на карте можно наблюдать инверисию крупного участка приблизительно с 550 тыс. нуклеотида по 1350 тыс. (по горизонтали).
Рис.3 Карта локального сходства геномов CP002640.1 и CP002465.1
 По горизонтали расположен геном CP002640.1, по вертикали CP002465.1.
Крупных перестроек не наблюдается. Есть несколько негомологичных участков.
Таким образом, основываясь на картах локального сходства, можно сделать вывод, что эти геномы достаточно
близки друг к другу, между ними не происходило крупных перестроек. А геном NC_012926.1,
наоборот, отличает от обоих наличие инверсии. Таким образом, можно предположить, что штамм BM407 эволюционно более новый.
По горизонтали расположен геном CP002640.1, по вертикали CP002465.1.
Крупных перестроек не наблюдается. Есть несколько негомологичных участков.
Таким образом, основываясь на картах локального сходства, можно сделать вывод, что эти геномы достаточно
близки друг к другу, между ними не происходило крупных перестроек. А геном NC_012926.1,
наоборот, отличает от обоих наличие инверсии. Таким образом, можно предположить, что штамм BM407 эволюционно более новый. |







