Ралдугина Василиса Студентка Факультета биоинженерии и биоинформатики МГУ имени М.В. Ломоносова  |
Обо мне |
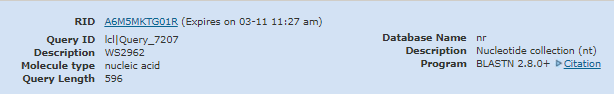
Нуклеотидный Blast
Последовательность, прямое прочтение: straight.fasta
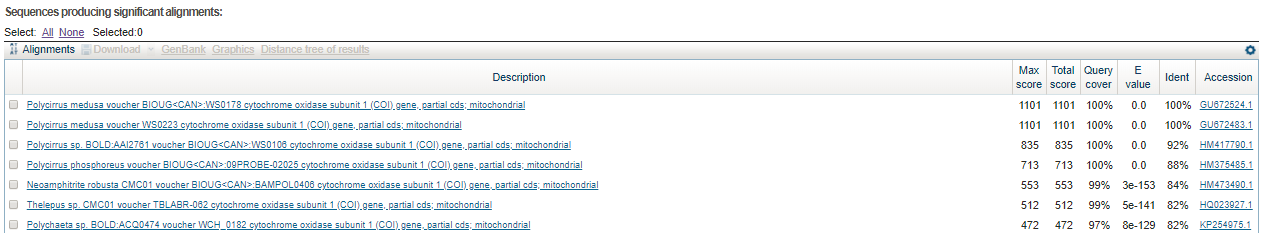
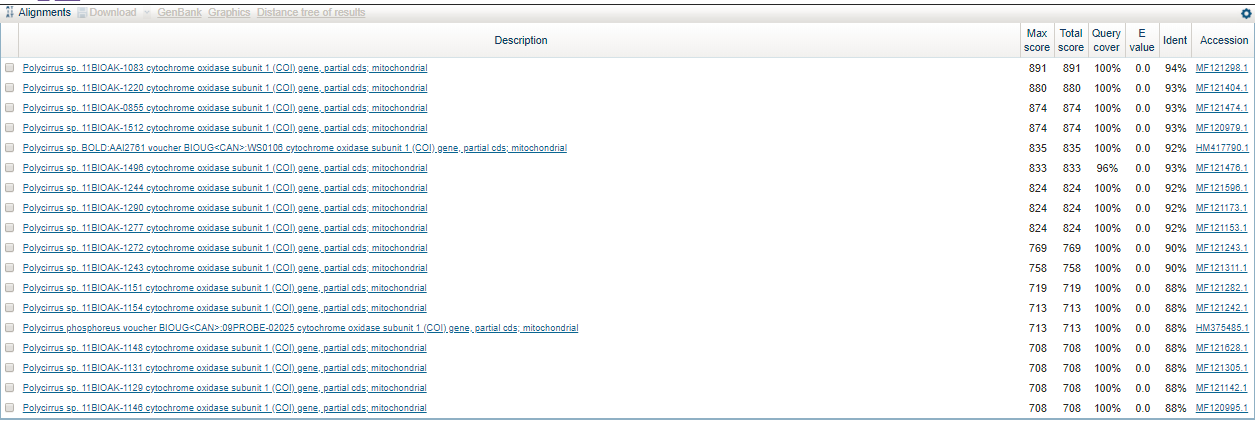
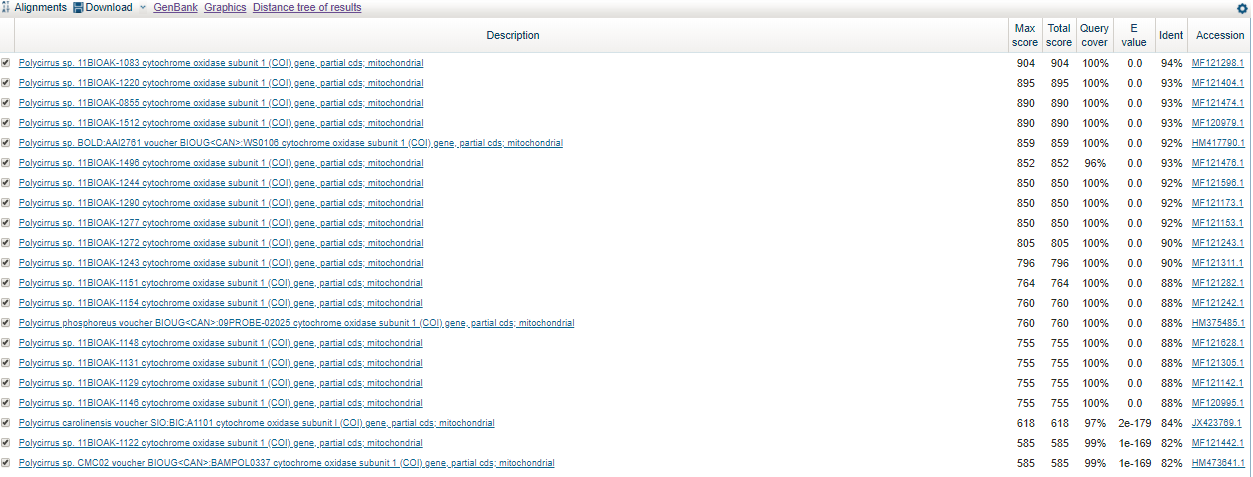
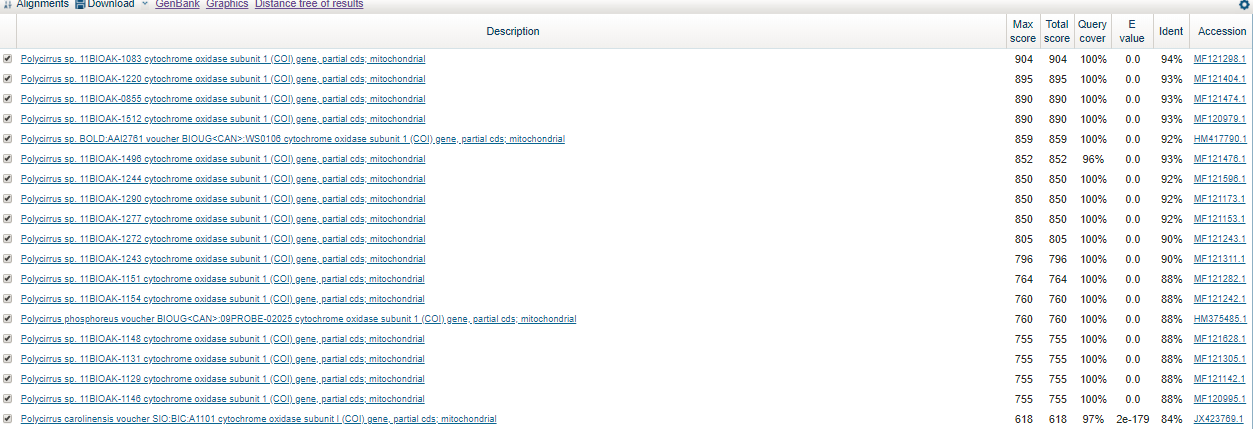
Максимально сходство с последовательностью, доступной по ссылке. В данном случае будут рассматриваться три алгоритма: blastn, megablast и discontiguous megablast. Чтобы сравнить их, нужно расширить область поиска. Результаты запроса по роду Polycirrus (taxid:516039): 18 последовательности для megablast, 44 для discontiguous megablast, 51 для blastn.
Все три алгоритма нашли одинаковые последовательности с максимальным сходством.
megablast, discontiguous megablast, discontiguous blastn Необходимо проверить наличие гомологов определенных белков в геноме организма X5 (Amoboaphelidium protococarum) c помощью локального BLAST.
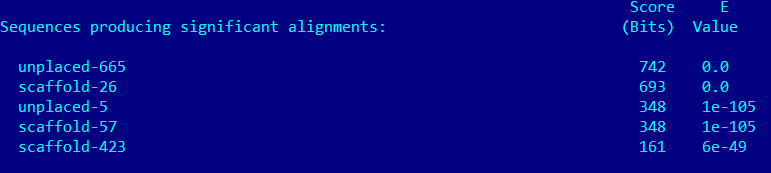
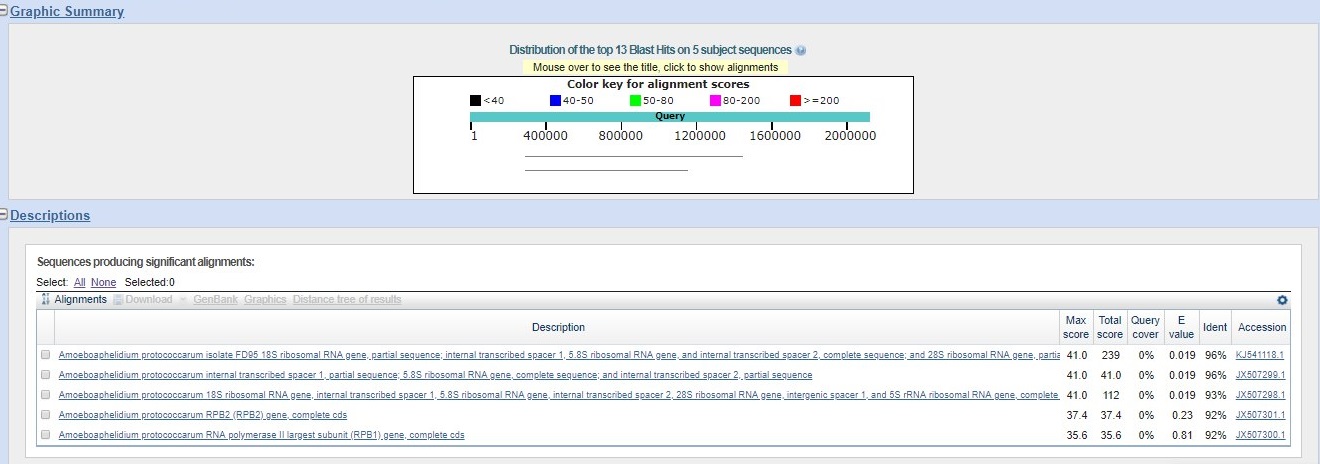
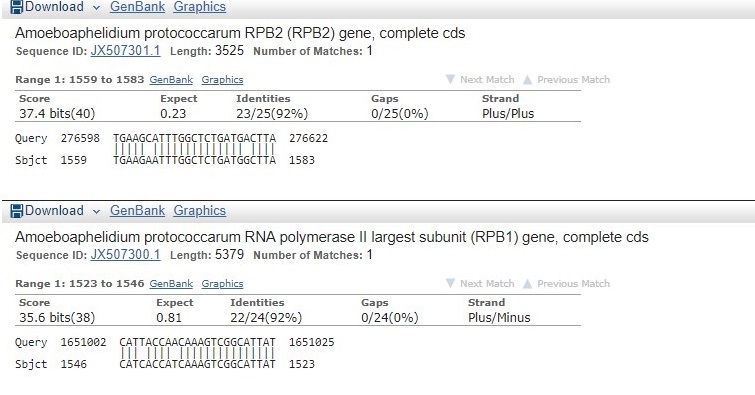
Задание выполнялось с помощью версии BLAST+, установленной на kodomo. Белок HSP71_YEASTБелок HSP71_YEAST - белок теплового шока. Консервативный шаперон HSP70. Результат работы программы tblastn в файле Лучший результат: Лучшая находка имеет хороший E-value и остальные параметры, таким образом, что эту находку можно назвать гомологом, и скорее всего она несет схожие функции. Белок PRPC_EMENIPRPC_EMENI - митохондриальная цитратсинтаза. Результат работы программы tblastn в файле Лучший результат: Находку можно охарактеризовать как гомологичную, т.к. параметры сходства я сочла достаточно высокими. Белок TBB_NEUCRTBB_NEUCR - тубулин, белок, участвующий в образовании микротрубочек Результат работы программы tblastn в файле Лучший результат: Параметры сходства снова достаточно хорошие, так что можно сказать, что данный белок является гомологом. С помощью команды infoseq пакета EMBOSS была получена информация о длинах контигов (infoseq X5.fasta -only -name -length.) Я выбрала последовательность scaffold-17 (длина 2125590 нуклеотидов), т.к. она достаточно большая для наличия в ней кодирующего белок гена, и поместила ее в отдельный файл (командой seqret X5.fasta:scaffold-17 -out scaf17.fasta). Далее был запущен blastn с параметрами по умолчанию и ограничению по таксону Amoeboaphelidium (taxid:1243176). Результат работы blastn Как видно из результатов, у нас есть 5 последовательностей генов с Identity 92-96%. Такой низкий Query cover обусловлен малыми размерами гена по сравнению с длиной общей последовательности. Я считаю, что можно утверждать, что данный контиг содержит найденные гены. Далее мной были выбраны гены белка, закодированные в скэффолде Рисунок Лучший в списке находок - Amoeboaphelidium protococcarum RPB2 (RPB2) gene, complete cds © Raldugina Vasilisa 2016 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||