Белки-гомологи - белки, имеющие общее происхождение.
Ортологи - белки, которые разошлись в результате видообразования. Таким образом,
белки-ортологи находятся в организмах разных видов и, как правило, выполняют схожие функции.
Паралоги - белки, образовавшиеся в результате дупликации генов. Следовательно, такие белки
существуют в одном организме и эволюционируют почти независимо друг от друга, а значит, могут сильно различаться
в функциях.
1. Поиск гомологов.
Поиск достоверных гомологов указанного белка вёлся среди протеомов перечисленных бактерий:
| Видовое название | Мнемоника | Таксономия |
| Serratia proteamaculans | SERP5 |
cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacterales; Yersiniaceae; Serratia |
| Proteus mirabilis | PROMH |
cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacterales; Morganellaceae; Proteus
|
| Saccharophagus degradans | SACD2 | cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Cellvibrionales; Cellvibrionaceae; Saccharophagus |
| Pseudomonas mendocina |
PSEMY |
cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Pseudomonadales; Pseudomonadaceae; Pseudomonas; Pseudomonas aeruginosa group |
| Rhizobium meliloti |
RHIME |
cellular organisms; Bacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Sinorhizobium/Ensifer group; Sinorhizobium |
| Brucella suis |
BRUSU |
cellular organisms; Bacteria; Alphaproteobacteria; Rhizobiales; Brucellaceae; Brucella |
| Neisseria meningitidis | NEIMA | cellular organisms; Bacteria; Proteobacteria; Betaproteobacteria; Neisseriales; Neisseriaceae; Neisseria |
| Aromatoleum aromaticum | AROAE |
cellular organisms; Bacteria; Proteobacteria; Betaproteobacteria; Rhodocyclales; Rhodocyclaceae; Aromatoleum |
Перед началом работы протеомы бактерий были собраны в единый файл
compiled.fasta. Последовательность белка, гомологи которого надо отыскать, была
сохранена в файл CLPX_ECOLI.fasta.
Затем из файла с протеомами была создана база данных для дальнейшего поиска гомологов.
Командная строка: makeblastdb -in compiled.fasta -dbtype prot
Далее был запущен поиск с помощью алгоритма BLASTP.
Командная строка: blastp -query CLPX_ECOLI.fasta -db compiled.fasta -evalue 0.001 -out
blast.out -outfmt 7 -num_alignments 50
Из найденных белков я выбрала самые достоверные гомологи (исходя из E-value).
Аминокислотные последовательности этих белков собраны в файл chosen.fasta.
Далее было построено выравнивание всех последовательностей.
Командная строка: muscle -in chosen.fasta -out align.fasta
На основе полученного файла методом Минимальной эволюции (Minimum Evolution) было построено
филогенетическое дерево.
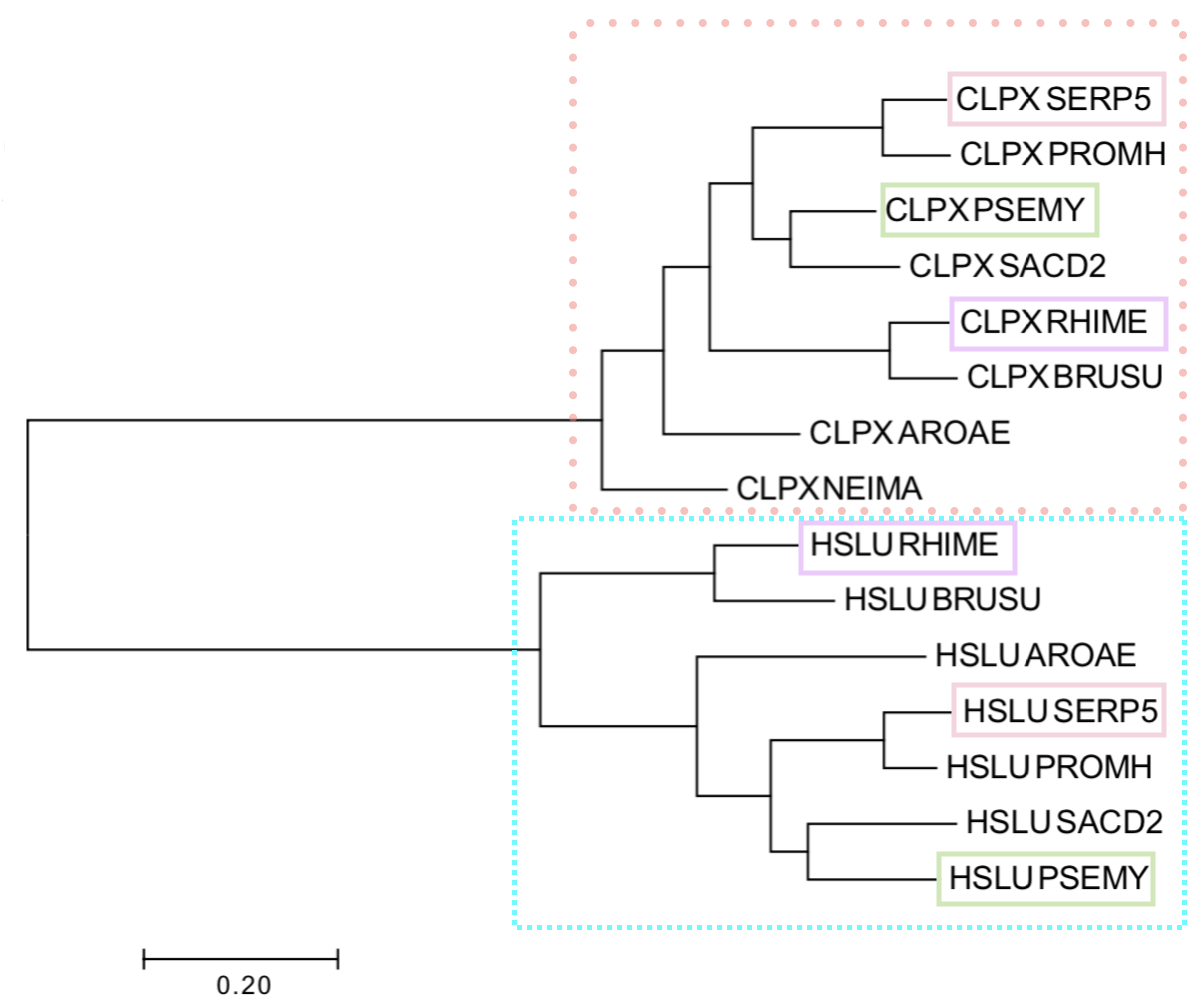
Рис.1. Филогенетическое дерево.
Оттенками розового и бирюзового отмечены ортологи - CLPX и HSLU (ATP-dependent protease ATPase subunit HslU; субъединица входящая в состав той же АТФ-зависимой протеазы).
Сиреневым отмечены паралоги RHIME, салатовым - паралоги PSEMY, светло-розовым - паралоги SERP5. Паралоги - белки, имеющие общего предка и находящиеся в организме одного вида, однако функционально разошедшиеся в ходе эволюции.


