- Смена активной директории и просмотр содержимого директорий.
- Посредством программы "putty" вошла на машину kodomo-count.cmm.msu.ru (по протоколу ssh).
- Выполнила команду "ls", увидела: Term_1 Term_2 from_aba public_html winscp.RND; - зашла таким образом в свою директорию на диске P.
- Выполнила команду "ls ..". Увидела список рабочих директорий всех студентов курса, т.е. поднялась по древу директорий на один шаг выше, нежели в предыдущий раз.
- Выполнив команду "cd ..", а затем команду "ls", увидела то же самое, что в предыдущий раз: список директорий студентов курса.
- Повторяла пару команд предыдущего пункта, пока картина не перестала меняться. Вывод: каждый раз эта пара отсылает в предыдущую директорию. Когда этот процесс прекратился - мы дошли до корневых директорий.
- Выполнила "cd" (без параметров), чтобы вернуться в домашнюю директорию. Выполнила "pwd", эта команда сообщает полное имя текущей директории.
- Сделала активной директорию Practice5: команда "cd X" делает активной поддиректорию X текущей директории. Просмотрела содержимое директории командой "ls".
- Создание и просмотр файлов.
В качестве активной - директория Practice5.
- Выполнила команду
seqret sw:p00000 -auto
подставив вместо "p00000" код доступа своего белка в банке Swiss-Prot. После этого выполнила команду "ls" и убедилась, что в директории появился новый файл: menc_ecoli.fasta. Выполнила команду "more X", подставив вместо X имя нового файла: menc_ecoli.fasta. Увидела содержание нового файла с последовательностью моего белка.
- Выполнила команду
entret sw:p00000 -auto
(аналогично заменив "p00000"). Выполнила команду "ls" и убедилась, что в директории появился новый файл: menc_ecoli.entret. Выполнила команду "more X", подставив вместо X имя нового файла. Увидела поля сведений о моём белке из банка Uniprot. Нажала несколько раз клавишу <Enter> , затем клавишу "пробел". Новый файл содержит полную информацию о белке из Uniprot.
- Выполнила команду
- Некоторые способы облегчения работы в командной строке.
- Понажимала клавиши "стрелка верх" и "стрелка вниз". С помощью них можно избавить себя от повторного набора ранее встречавшихся команд. Стрелкой вверх можно как бы пролистать прошлые команды, начиная от самой ближайшей и далее, стрелкой вниз - вернуться к пролистанным.
- Выполнила команду "history". Она выдаёт историю заданных команд, от самой первой и до самой последней.
- Выбрала в своей директории файл, первая буква названия которого отличается от первых букв всех других файлов. Вот, допустим, файл fullANDpart.fasta. Набрала в командной строке "more", затем после пробела — первую букву имени файла - f, затем нажала клавишу <Tab>. Замечательный результат! Команда выдаёт полное имя файла. Затем можно и <Enter> нажать, тогда мы увидим его содержание: последовательность белка и фрагмент этой последовательности в fasta-формате.
- Построила полное (глобальное) оптимальное выравнивание с помощью программы needle пакета EMBOSS: aligne.needle
Пользуясь программой seqret, создала файл с последовательностью белка, родственного моему - menc_mycle.
Построила полное оптимальное выравнивание двух белков — моего и родственного. Для этого выполнила команду:
needle menc_ecoli.fasta menc_mycle.fasta aligne.needle -auto
Содержимое файла aligne.needle: некоторые исходные данные о белках, выравнивание, процетный состав схожести, в том числе - тождественно равных промежутков, сходных, наличие гэпов.Задание штрафов за гэпы, отличных от заданных по умолчанию. Повторила предыдущую команду, но изменила имя выходного файла и опустила опцию "-auto". Программа спросила о значениях параметров. Дала параметрам вдвое большие значения, чем заданные по умолчанию (сообщаются в квадратных скобках: промежуток, открывающий штраф: 10.0; штраф расширения промежутка: 0.5) - 20.0 и 1.0 соответственно. Из-за изменения параметров выравнивание измнилось очень сильно: aligne1.needle
1е выравнивание (параметры по умолчанию) 2е выравнивание (параметры в 2 раза больше) длина 415 645 идентичность 85/415 (20.5%) 3/645 (0.5%) схожесть 123/415 (29.6%) 4/645 (0.6%) гепы 176/415 (42.4%) 636/645 (98.6%) оценка 160.5 11.0 Получение выдачи в формате, пригодном для импорта в GeneDoc. Запустила программу needlе, добавив опцию "
-aformat msf ". Открыла полученный файл программой GeneDoc. - Построила локальное (частичное) оптимальное выравнивание тех же последовательностей с помощью программы water пакета EMBOSS: aligne.water
- Попробовала поменять параметры так же, как в предыдущем упражнении - увеличить вдвое: aligne1.water
- Также попробовала уменьшить вдвое значение запрашиваемых параметров: aligne2.water
- Сравним полученные выравнивания
Получили несколько разных выравниваний для двух последовательностей.
Сравнение выравниваний — это не только и не столько сравнение количественных параметров выравниваний, сколько выяснение того, насколько одинаково сопоставление остатков одной последовательности остаткам второй.
Поэтому прежде всего ответим на следующие вопросы:- есть ли хотя бы один пример того, что одной и той же позиции первой последовательности в разных глобальных выравниваниях сопоставлены разные позиции второй последовательности?
- есть ли хотя бы один пример того, что одной и той же позиции первой последовательности в разных локальных выравниваниях сопоставлены разные позиции второй последовательности?
- есть ли хотя бы один пример того, что в одном глобальном выравнивании какой-либо позиции первой последовательности сопоставлена некоторая позиция второй, а в другом выравнивании против той же позиции оказался пропуск?
- есть ли хотя бы один пример того, что в одном локальном выравнивании какой-либо позиции первой последовательности сопоставлена некоторая позиция второй, а в другом выравнивании против той же позиции оказался пропуск?
- соответствуют ли оптимальные локальные выравнивания, построенными с использованием разных параметров,
одним и тем же фрагментам последовательностей?
- если сравнивать исходное выравнивания aligne.water с выравниванием с увеличенными вдвое параметрами aligne1.water, то можно заметить, что первое выравнивание целиком включает в себя участок второго;
- если сравнивать исходное выравнивания aligne.water с выравниванием с уменьшенными вдвое параметрами aligne2.water, можно заметить, что начинаются они с одних и тех же а.о., но, напротив, участок второго будет включать в себя участок первого, который заканчивается раньше;
- если сравнивать выравнивание с увеличенными вдвое параметрами aligne1.water с выравниванием с умешёнными вдвое параметрами aligne2.water, соответственно, можно заключить, что участок второго выравнивания включает в себя участок первого; таким образом, можно заключить, что в данном случае выравнивание с уменьшёнными вдвое параметрами промежутка, открывающего штраф и штрафа расширения промежутка является наиболее обширным, тогда как выравнивание с увеличенными параметрами - наименее; выравнивание с параметрами по умолчанию показывает в этом отношении средний результат. - совпадают ли локальные выравнивания с соответствующими частями глобальных выравниваний?
aligne.needle aligne1.needle aligne.water aligne1.water aligne2.water aligne.needle aligne1.needle aligne.water совпадает не полностью:
позиции №19-297- MENC_ECOLI
№21-269- MENC_MYCLE совпадают;
№11-18 и 298-314- MENC_ECOLI
№270-279- MENC_MYCLE -> нет.нет aligne1.water да. позиции №121-272- MENC_ECOLI
№95-243- MENC_MYCLEнет aligne2.water не совпадают нет
да, есть. Это вообще настолько разные выравнивания, что сравнивать их бесполезно, и второе aligne1.needle, полученное путём удвоения параметров первого aligne.needle по умолчанию (промежуток, открывающий штраф: 10.0; штраф расширения промежутка: 0.5), вообще содержит лишь 9 пар сопоставленных а.о.
да, есть - если сравнивать исходное выравнивание и выравнивание с параметрами в 2 раза меньше - aligne.water, aligne2.water - например, позиции 103 белка MENC_ECOLI в первом выравнивании соответствует позиция 88 белка MENC_MYCLE. Во втором же выравнивании этой позиции бека MENC_ECOLI соответствует уже другая позиция MENC_MYCLE - 95я;
- если сравнивать исходное выравнивание и выравнивание с параметрами в 2 раза больше - aligne.water, aligne1.water, - видим, что сопоставление всех аминокислот белка одинаково;
- если сравнивать выравнивание с параметрами в 2 раза больше и выравнивание с параметрами в 2 раза меньше - aligne1.water, aligne2.water,- можем найти как участки полного совпадения сопоставления аминокислот, так и различного сопоставления аминокислотных остатков. Например, в следующем фрагменте:
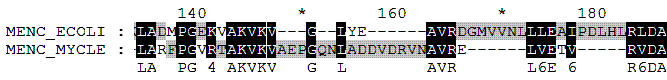
Как видно, по краям фрагмента сопоставления полностью совпадают, тогда как посередине - существенно расходятся как в сопоставлении а.о., так и в расстановке гэпов.
Как уже говорилось, полученные глобальные выравнивания aligne.needle - с параметрами по умолчанию - и aligne1.needle - с параметрами, увеличенными вдвое, - сравнивать бессмысленно. Конечно, есть такие примеры. Достаточно посмотреть на второе "выравнивание", где мы вообще с обоих концов увидим сплошные гэпы..
- если сравнивать исходное выравнивания aligne.water с выравниванием с увеличенными вдвое параметрами aligne1.water, таких примеров не встречается;
- если сравнивать исходное выравнивания aligne.water с выравниванием с уменьшенными вдвое параметрами aligne2.water, можно привести такой пример: в первом выравнивании позиции MENC_ECOLI: 57 соответствует позиция MENC_MYCLE: 52; во втором же этой позиции MENC_ECOLI: 57 соответствует пропуск - MENC_MYCLE: -;
- если сравнивать выравнивание с увеличенными вдвое параметрами aligne1.water с выравниванием с умешёнными вдвое параметрами aligne2.water, можно на том же фрагменте, который являлся примером несоответствия некоторых сопоставлений а.о. в выравниваниях, показать и наличие сопоставления пропуска тому а.о., которому в другом выравнивании сопоставлялся другой а.о.:
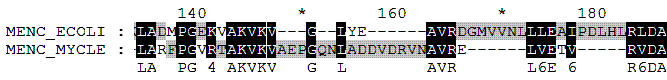
**aligne.needle - глобальное выравнивание с параметрами по умолчанию;
aligne1.needle - глобальное выравнивание с параметрами, увеличенными вдвое;
aligne.water - локальное выравнивание с параметрами по умолчанию;
aligne1.water - локальное выравнивание с параметрами, увеличенными вдвое;
aligne2.water - локальное выравнивание с параметрами, уменьшенными вдвое.
К блоку
Ко 2му семестру
На главную
© Шишкова Настя, 2008
| 1е выравнивание (параметры по умолчанию) | 2е выравнивание (параметры в 2 раза больше) | 3е выравнивание (параметры в 2 раза меньше) | |
| длина | 323 | 159 | 387 |
| идентичность | 85/323 (38.7%) | 49/159 (30.8%) | 110/387 (28.4%) |
| схожесть | 125/323 (38.7%) | 73/159 (45.9%) | 148/387 (38.2%) |
| гепы | 79/323 (24.5%) | 17/159 (10.7%) | 147/387 (38.0%) |
| оценка | 175.0 | 96.0 | 310.25 |