Практикум 7.
1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Из белков с β-листами в трансмембранной части в базе OPM я выбрала FpvA - рецептор пиовердина.
Информация об этом белке:
- Type: Transmembrane
- Class: Beta-barrel transmembrane
- Superfamily: Ligand-gated protein channels - лиганд-зависимые ионные каналы
- Family: Outer Membrane Receptor (OMR) - рецепторы на внешней мембране
- Name: Pyoverdine Outer Membrane Receptor FpvA - рецептор пиовердина FpvA на внешней мембране
- Species: Pseudomonas aeruginosa - синегнойная палочка
- Localization: Bacterial Gram-negative outer membrane - внешняя мембрана грам-отрицательных бактериий
- UniProt ID: FPVA_PSEAE
- PDB: 2iah
Что белок делает? FpvA выступает в качестве транспортера пиовердина, связанного с железом, в клетку, и подает сигналы для начала биосинтеза пиовердина в условиях дефицита железа.
В таблице ниже приведены координаты трансмембранных участков белка по данным OPM.
| Номер петли | OPM | DeepTMHMM |
|---|---|---|
| 1 | 278-286 | 279-286 |
| 2 | 290-299 | 290-298 |
| 3 | 307-316 | 309-316 |
| 4 | 330-338 | 330-337 |
| 5 | 344-353 | 346-353 |
| 6 | 397-404 | 396-403 |
| 7 | 410-418 | 412-419 |
| 8 | 456-464 | 455-463 |
| 9 | 472-480 | 473-481 |
| 10 | 530-538 | 530-539 |
| 11 | 543-551 | 543-551 |
| 12 | 569-578 | 570-579 |
| 13 | 583-590 | 583-590 |
| 14 | 615-624 | 616-623 |
| 15 | 629-636 | 629-636 |
| 16 | 672-680 | 673-681 |
| 17 | 684-692 | 685-692 |
| 18 | 712-720 | 713-720 |
| 19 | 728-736 | 728-735 |
| 20 | 761-770 | 763-769 |
| 21 | 776-781 | 775-782 |
| 22 | 806-813 | 807-813 |
Запускала DeepTMHMM для последовательности белка, взятой из UniProt.
Ссылка на файл с выдачей DeepTMHMM в формате .gff3
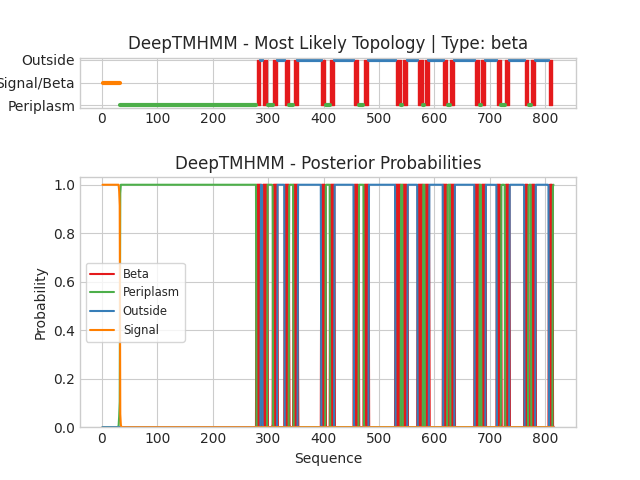
И DeepTMHMM, и OPM нашли 22 трансмембранных участка. Координаты для каждого из них отличаются незначительно, если есть различия, то в 1-3 аминокислоты. Вероятно данные отличия связаны с погрешностью предсказания.
2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Мне был предложен белок NavPaS, взаимодействующий с mu-дигитоксином.
Информация об этом белке:
- Type: Transmembrane
- Class: Alpha-helical polytopic - белки с альфа-спиралями в трансмембранной части
- Superfamily: Ion channel (VIC) superfamily - потенциал-зависимые ионные каналы
- Family: Voltage-gated calcium and sodium channels - потенциал-зависимые кальциевые и натриевые каналы
- Name: Channel NavPaS - NavPaS канал
- Species: Periplaneta americana - Американский таракан
- Localization: Eukaryotic plasma membrane - мембрана эукариотической клетки
- UniProt ID: SCNA1_PERAM
- PDB: 6a90
Что белок делает? NavPaS - потенциал-зависимый натриевый канал, играет важнейшую роль в работе нервной системы, является мишенью для нейротоксинов, например, тетродотоксина (TTX).
Mu-дигитоксин добывают из пустынного кустарникового паука (Diguetia canities). Он взаимодействует с NavPaS, что приводит к задержке инактивации пресинаптических потенциал-зависимых натриевых каналов, тем самым вызывает паралич у насекомых.
Запускала DeepTMHMM для последовательности белка, взятой из UniProt.
Ссылка на файл с выдачей DeepTMHMM в формате .gff3
| Номер петли | OPM | DeepTMHMM |
|---|---|---|
| 1 | 140-158 | 141-157 |
| 2 | 164-185 | 167-182 |
| 3 | 196-217 | 203-212 |
| 4 | 224-240 | 214-223 |
| 5 | 258-280 | 261-281 |
| 6 | 390-416 | 393-411 |
| 7 | 519-538 | 520-538 |
| 8 | 550-571 | 553-562 |
| 9 | 581-600 | 564-573 |
| 10 | 606-624 | 585-600 |
| 11 | 639-663 | 641-660 |
| 12 | 715-741 | 724-737 |
| 13 | 856-875 | 861-875 |
| 14 | 888-910 | 891-903 |
| 15 | 917-942 | 905-914 |
| 16 | 948-965 | 927-939 |
| 17 | 975-1003 | 982-999 |
| 18 | 1087-1115 | 1087-1107 |
| 19 | 1171-1189 | 1172-1189 |
| 20 | 1201-1225 | 1201-1221 |
| 21 | 1228-1250 | 1235-1250 |
| 22 | 1261-1279 | 1263-1272 |
| 23 | 1292-1320 | 1302-1318 |
| 24 | 1385-1411 | 1386-1406 |
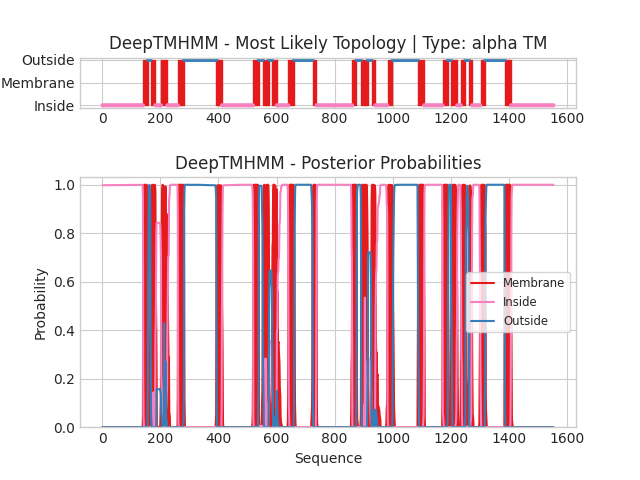
И DeepTMHMM, и OPM нашли 24 трансмембранных участка. Координаты практически всех этих участков перекрываются за исключением 4-го. Также 9-му и 15-му по OPM соответсвуют 10 и 16, предсказанные DeepTMHMM. При этом, судя по графичсекой выдаче, вероятности предсказания DeepTMHMM трансмембранных участков близки к 1, немного ниже вероятности для 4 и 9.