Практикум 8.
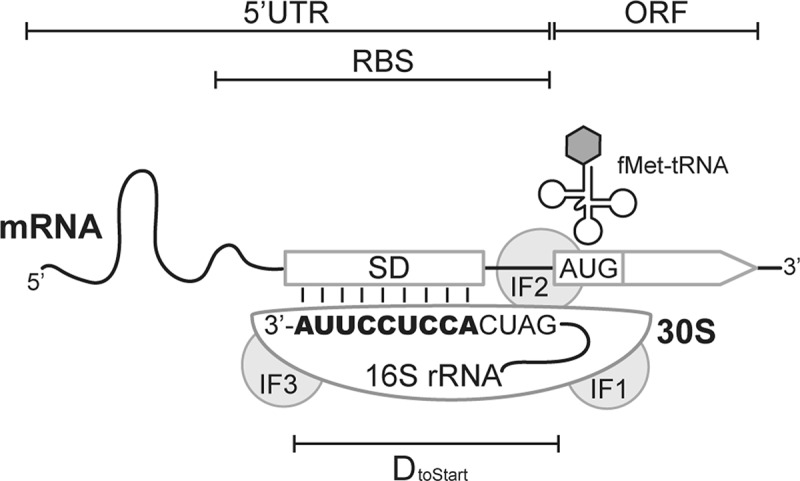
В качестве сигнала для исследования я выбрала последовательность Shine-Dalgarno (SD) - сайт посадки рибосомы в мРНК у бактерий, архей. Также обнаружена в некоторых генах хлоропластов и митохондрий.
Описание сигнала
- Название: Последовательность Шайна-Дальгарно
- Носитель: мРНК бактерий, архей.
- Кому адресован: малая субъединица рибосомы, которая содержит 16S рРНК с комплементарной последовательностью на 3' конце (анти - Шайна-Дальгарно).
- Реакция адресата: связывание SD с анти-SD приводит к тому, что рибосома совершает правильную посадку на мРНК - начальный кодон оказывается в P-сайте рибосомы, начинается трансляция.
- Сила сигнала: сила сигнала зависит от нескольких факторов.
Во-первых, от степени комплементарности между последовательностью SD и 16S рРНК, поскольку при более прочном связывании скорость посадки рибосомы на мРНК и инициации трансляции выше.
Вариации, мутации в последовательности могут влиять на эффективность связывания, а значит и трансляции.
Во-вторых, важно расстояние между SD и стартовым кодоном, которое должно быть равным ~8 нуклеотидам.
В общем, вероятно сила сигнала SD различна для разных генов в разных организмах. Хотя у большинства бактерий именно такой механизм является главным в инициации трансляции, существуют и другие механизмы.
Например есть прокариоты с мРНК, SD которых не способна стабильно связываться с анти-SD, а правильное связывание с P-сайтом объясняют особенностью структуры такой мРНК и работой рибосомальных белков и факторов инициации трансляции.
- Примеры сигнала:
- Escherichia coli, консенсусная последовательность - AGGAGGU
- В генах T4 вируса E. coli - GAGG
- Bacillus subtilis - AGGAGG
Источники
- - Shine J, Dalgarno L. The 3'-terminal sequence of Escherichia coli 16S ribosomal RNA: complementarity to nonsense triplets and ribosome binding sites. Proc Natl Acad Sci U S A. 1974 Apr;71(4):1342-6. doi: 10.1073/pnas.71.4.1342
- - Wen JD, Kuo ST, Chou HD. The diversity of Shine-Dalgarno sequences sheds light on the evolution of translation initiation. RNA Biol. 2021 Nov;18(11):1489-1500. doi: 10.1080/15476286.2020.1861406
Работоспособность сервисов для поиска сигнала
Специализированных работающих онлайн-сервисов для поиска только SD-последовательностей я не нашла, воспользовалась более "общим" сервисом.
uORF4u
- биоинформаический инструмент, предназначенный для аннотирования консервативных коротких открытых рамок считывания (upstream open reading frames, uORFs). У прокариот ключевым признаком для идентификации потенциально функциональных uORF является наличие последовательности SD перед стартовым кодоном.
Как он работает?
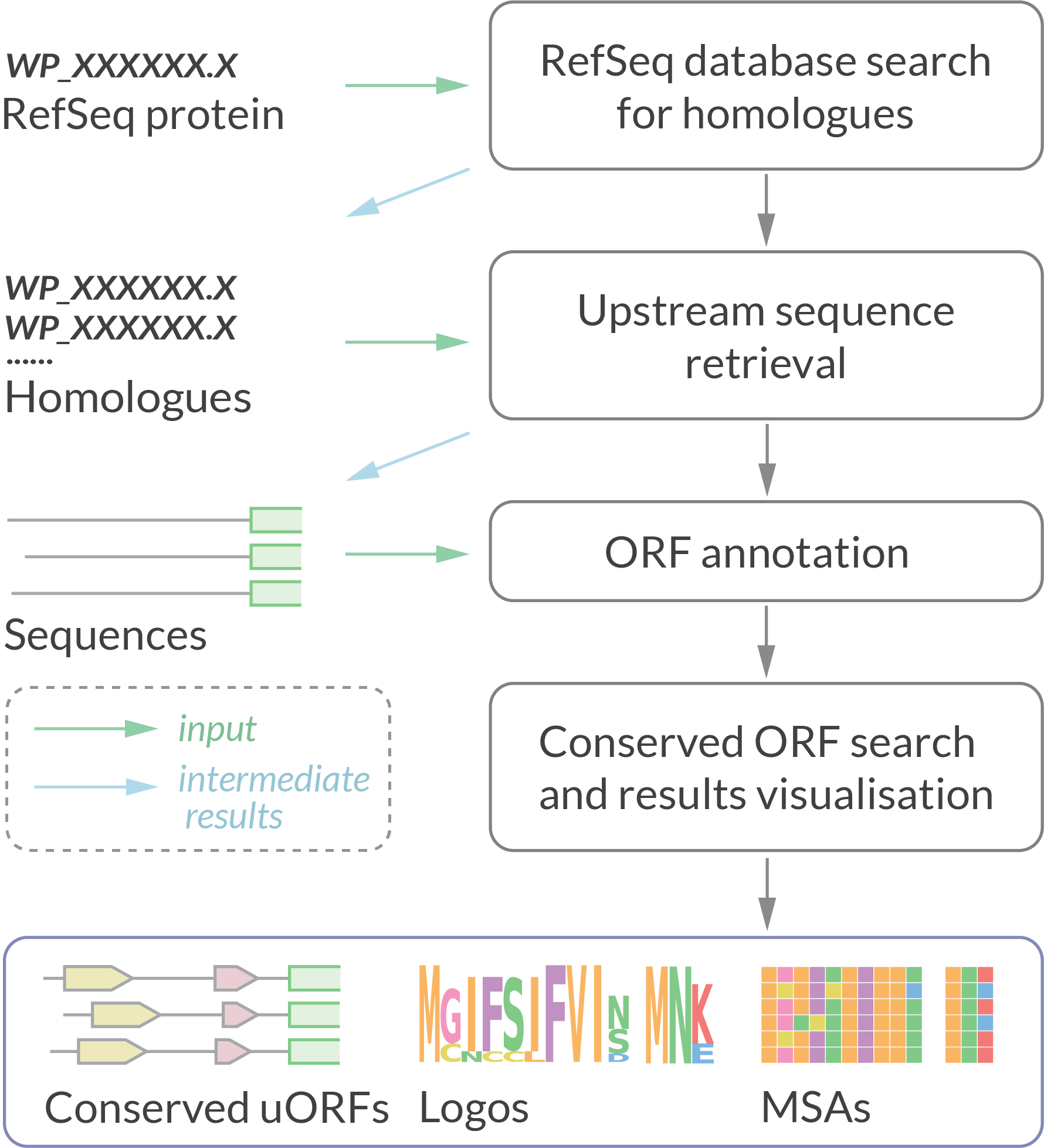
На вход алгоритму можно подавать a) последовательность аминокислот белка (или его ascension number в NCBI RefSeq), b) несколько последовательностей белков-гомологов (или их ascension number) c) несколько нуклеотидных последовательностей.
a) Сначала uORF4u выполняет BLASTP поиск по базе данных белков RefSeq для получения последовательностей гомологов. Далее следуют те же шаги, что и в b).
b) Используя полученный список uORF4u извлекает соответствующие upstream последовательности. Для эукариот участок upstream - это 5ʹ UTR-последовательность полного транскрипта, а для прокариот участок upstream - это заданная пользователем (по умолчанию 500 нуклеотидов) длина от стартового кодона главной ORF (main ORF, mORF). Полученные нуклеотидные последовательности сохраняются как промежуточный результат в формате FASTA. Далее алгоритм работает как в c).
c) Можно сразу подать набор нуклеотидных последовательностей. Авторы отмечают, что в таком случае uORF4u может быть использован для поиска консервативных ORF вообще, т. е. необязательно upstream от конкретной mORF. Но есть ограничения в рекомендуемой длине последовательностей (от 100 до 1000 нуклеотидов) и количестве последовательностей (от 10 до 1000).
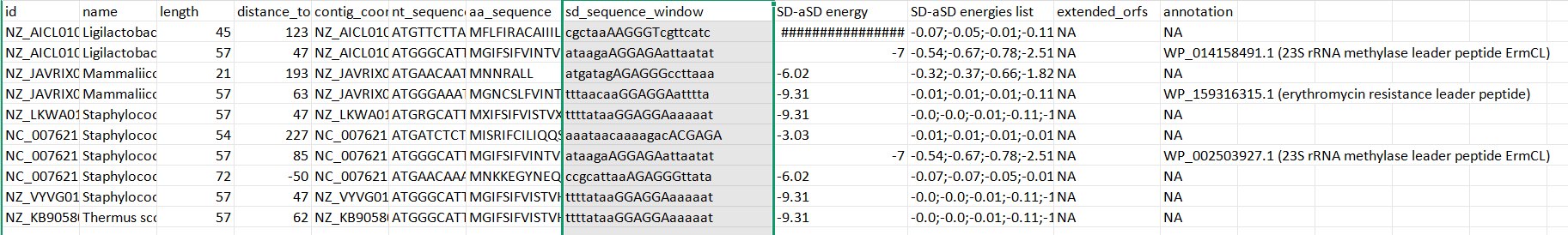
Далее идет аннотация, ORF определяется как область между стартовым кодоном и стоп-кодоном. Минимальная длина по умолчанию - 9 нуклеотидов. При анализе прокариотических последовательностей на этом этапе также происходит поиск последовательности SD в окне перед стартовым кодоном размером 20 нуклеотидов. Аннотация SD основана на расчете свободной энергии Гиббса SD - анти-SD взаимодействия. Свободная энергия Гиббса для SD - анти-SD взаимодействия рассчитывается как разница между энергией исходных молекул (мРНК и рРНК) и энергией их в косплексе. Среди найденных возможных рамок uORF4u ищет консервативные. Для этого используется "жадный алгоритм": uORF4u перебирает последовательности и стремится сделать сумму весов парных выравниваний между uORF максимальной. Выдача представляет собой результаты множественных выравниваний найденных консервативных uORFs: графики, logo последовательностей и файлы с множественными выравниваниями.
На сайте с веб-версией uORF4u предлагается посмотреть на работу алгоритма на примере гомологов 6 ErmC [https://server.atkinson-lab.com/uorf4u].

В архиве с результатом работы uORF4u есть папка annotated_ORFs, содержащая файлы .tsv, в которых находятся информация об аннотированных SD.
Ссылка на файл с объединенными аннотированными SD.