Практикум 9.
Моей бактерией была Streptomyces vietnamensis (NCBI RefSeq assembly: GCF_000830005.1)
Как и в практикуме 8 было решено взяться за последовательностю Шайна-Дальгарно (SD).
Воспользовалась с разрешения автора скриптом, который создает три файла: последовательности "окон" размером 25 нуклеотидов перед старт-кодоном (1), последовательности в 25 нуклеотидов после старт-кодона (2), гены наиболее консервативных белков, т.е. связанных с трансляцией и пр. (3).
Поскольку длина последовательности SD составляет примерно от 5 до 10 нуклеотидов (параметры -minw, -maxw) и интересовало нахождение мотива в единственном экземпляре (параметр -nmotifs) была использована следующая команда:
meme TRAIN.fasta -dna -nmotifs 1 -minw 5 -maxw 10
TRAIN.fasta был взят как файл с обучающей выборкой, в нем ожидаем выявить SD.
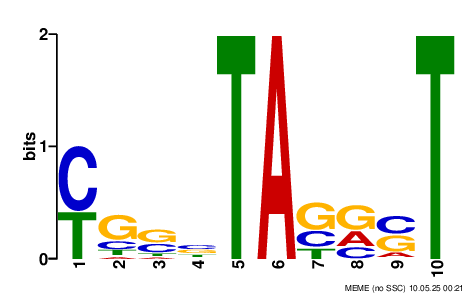
Выявленный мотив не соответствует каноническому консенсусу (AGGAGG), но схож с ним, содержит несколько остатков гуанина и аденина.
С помощью команд
fimo --oc fimo_pos -thresh 0.001 meme_out/meme.txt POSITIVE.fasta
fimo --oc fimo_neg -thresh 0.001 meme_out/meme.txt NEGATIVE.fasta
произвела поиск выявленного мотива с помощью FIMO, параметром -thresh задала p-value < 0.001 для находок.
Ссылка на выдачу для последовательностей с окном до старт-кодона - 393, после - 63.
Учитывая то, что в первом случае всего было 8216 последовательностей, у меня возникают сомнения в том, что YGSBTASRST - характерный для данной бактерии паттерн SD. Даже несмотря на то, что во втором случае находок гораздо меньше, чем в первом.