Практикум 10.
Выравнивание геномов
Для выполнения этого практикума были взяты полные сборки хромосом двух бактерий рода Enterococcus: Enterococcus faecium (NZ_CP039729.1) и Enterococcus hirae (NZ_CP023011.2). Эти организмы были найдены с помощью "Browse by Organism" в NSBI.
Были построены карты локального сходства с помощью Megablast и BLASTN.
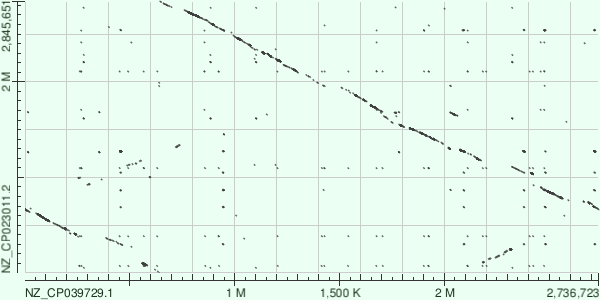
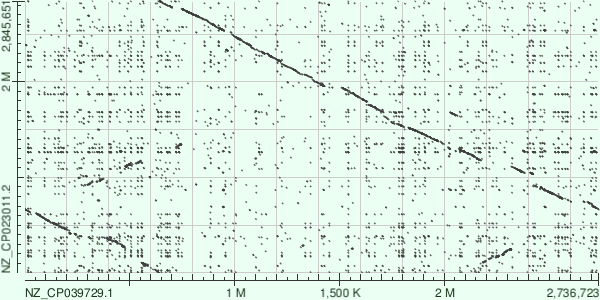
При использовании алгоритма blastn появилось много "шума" на фоне. Это означает большое количество повторов.
Можно заметить что на карте получилось 2 убывающих диагонали. То, что их получилось 2, можно объяснить тем, что хромосомы кольцевые, и разрыв одной из хромосом для секвенирования произошёл не в том месте, где произошёл разрыв другой. То, что они убывают,можно объяснить тем, что произошла инверсия. Также можно заметить несколько вставок/делеций с 250К до 400К, с 1400К до 1550К и тд.