ПРАКТИКУМ 9
Глобальное парное выравнивание гомологичных белков
Отобрав все белки с одинаковой мнемоникой функции у организмов Escherichia coli (strain K12) и Bacillus subtilis (strain 168) мной были выбраны три пары: BIPA, CDD и PURU
Таблица 1. Характеристики глобального парного выравнивания трёх пар белков
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
|---|---|---|---|---|---|---|---|
| Large ribosomal subunit assembly factor BipA |
BIPA_ECOLI | BIPA_BACSU | 1746.0 | 53.9% | 74.0% | 5 | 2 |
| Cytidine deaminase | CDD_ECOLI | CDD_BACSU | 101.0 | 13.9% | 20.5% | 176 | 8 |
| Formyltetrahydrofolate deformylase |
PURU_ECOLI | PURU_BACSU | 601.5 | 41.4% | 59.3% | 34 | 7 |
Таблица 2. Характеристики локального парного выравнивания трёх пар белков
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|
| Large ribosomal subunit assembly factor BipA | BIPA_ECOLI | BIPA_BACSU | 1751.5 | 54.9% | 75.2% | 0 | 0 | 99.0 | 98.2 |
| Cytidine deaminase | CDD_ECOLI | CDD_BACSU | 108.0 | 32.8% | 47.4% | 11 | 5 | 36.4 | 83.8 |
| Formyltetrahydrofolate deformylase |
PURU_ECOLI | PURU_BACSU | 611.5 | 45.0% | 63.9% | 20 | 5 | 95.4 | 91.0 |
Анализируя, приведенные выше таблицы можно с увереностью сказать, что белки BIPA и PURU гомологичны, так как имеют процент идентиности явно превышающий 25%,
соответсвующий случайному совпадению. Несмотря на то, что последовательности не полностью совпадают, прослеживаются консервативные участки, с высокой схожестью, к примеру для пары BIPA - это
участки: 8-32; 51-63; 69-80 и 358-377 (последовательности E. Coli) в глобальном выравнивание этой пары. Для белков PURU гомологичность прослеживается в участке
206-233 (последовательности E. Coli) для локального выравнивания этой пары.
В данном случае выравнивание Smith & Waterman несильно влияет на получаемые результаты схожести, лишь несколько повышая ее и сокрощая количество гэпов,
да и высокий процент покрытия белков выравнивания подтверждает незначительные изменения в локальном выравнивание относительно глобального.
Ситуация с парой CDD отлична от вышеизложенной, при глобальном выравние мы наблюдаем очень низкий процент схожести и большое
количесвто гэпов. Однако, число инделей слабо отличается, от гомологичных последовательностей, поэтому точно сказать об отсутсвии гомологии нельзя.
Локальное выравнивание этих последовательностей показывает более высокий процент совпадений, и меньшее число гэпов
(даже меньше чем у пары PURU с достаточно высокой степенью схожести). Но несмотря на это, найти четких
гомологичных участков не удалось.
Дополнительно я решил сравнить пространственные структуры белков puru_ecoli и puru_bacsu, чтобы точнее определить возможную гомологию.
Для этого использовалась программа PyMOL. Белок E. Coli покрашен в синий цвет, а его малые молекулы в оранжевый, белок Bacillus subtilis в красный, относящиеся к нему малые молекулы в циановый.
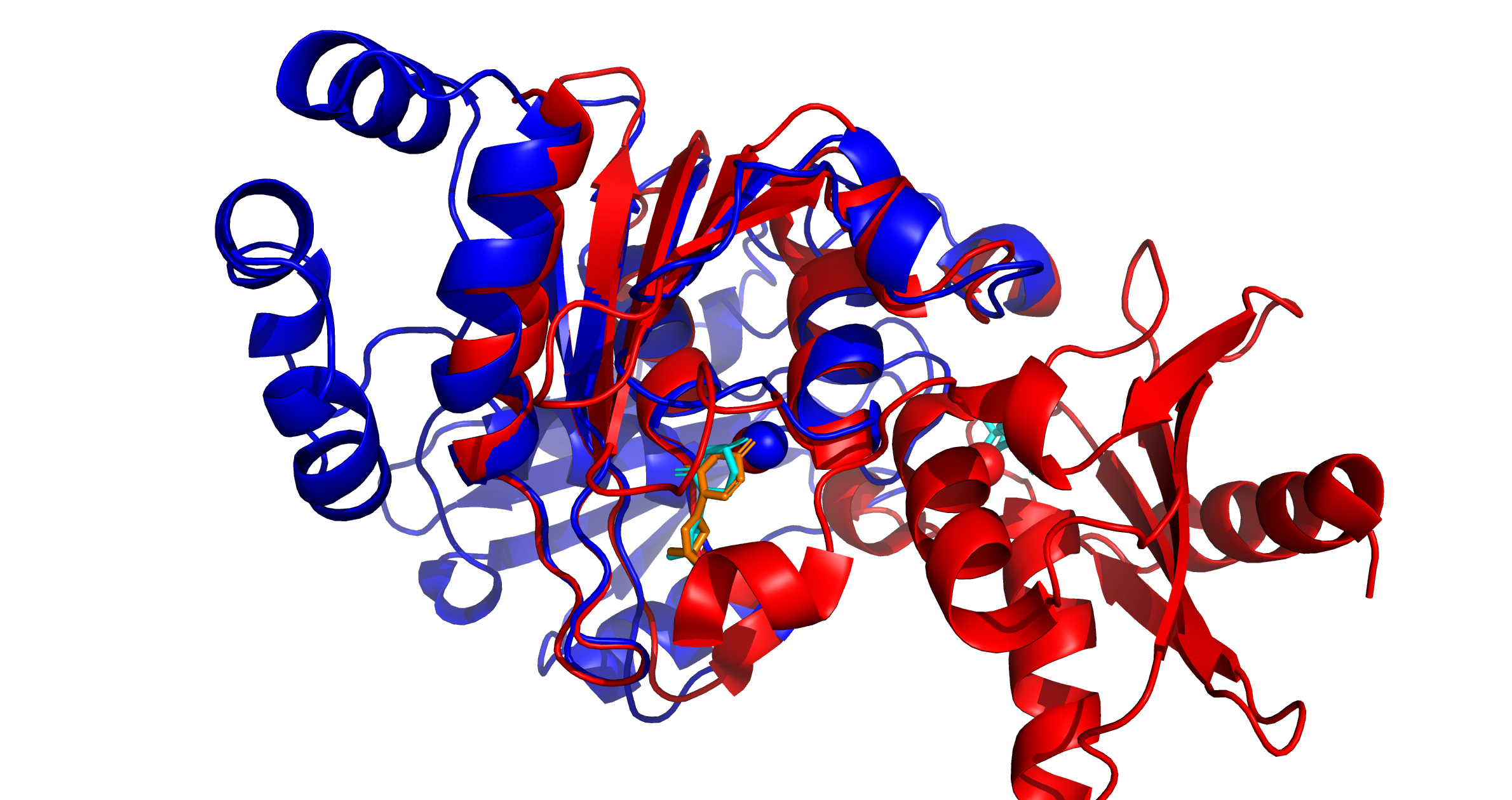
Рисунок 1. Сравнение пространственных структур белков пары CDD
Как мы можем заметить у данных белков присутсвуют схожие участки, которые совпадают при наложении. Расположение малых молекул также частично совпадает, так что можно сделать вывод, что каталитические сайты расположены одинаково. Из схожести структуры и более высокой идентичности при локальном выравнивание можно сделать вывод, что данные белки с большой долей вероятности имеют общность происхождения, однако, разошлись они достаточно давно и притерпели серьезные изменения.
Выравнивание случайных пар
Таблица 3. Выравнивание неродственных белков
| Program | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|
| Needle | DNAG_ECOLI | KHSE_BACSU | 30.0 | 3.3% | 6.2% | 666 | 8 | - | - |
| Water | DNAG_ECOLI | KHSE_BACSU | 42.0 | 28.3% | 48.2% | 6 | 2 | 9.5% | 19.1% |
Для выравнивания неродственных последовательностей, я случайно выбрал DNAG_ECOli - DNA primase и KHSE_BACSU - Homoserine kinase. Как и ожидалось для неродственных белков, выравнивания имели очень малую схожесть, и огромное количество гэпов. Хотя локальное выравнивание и показало более высокую схожесть, но процент покрытия выравниванием последовательностей оказался очень низок, и видимо, участки для выравнивания были выбраны случайно (у ECOLI 65-119 а. о., а у BACSU 248-306). Из этих данных, можем сделать точный вывод, что эти белки имеют схожее происхождение.
Множетсвенное выравнивание
Мной была выбрана мнемоника BIPA (Large ribosomal subunit assembly factor BipA). Данный белок участвует в сборке рибосом,
так что группе этих белков должна быть свойствена высокая консвервативность, что подтвержадется большим количеством записей о нем в UniProt.
Всего было найдено 40,436 записей.
Для множественного выравнивания, в дополнение к белкам Escherichia coli (strain K12) и Bacillus subtilis (strain 168),
были выбраны белки BIPA_SALTY (Salmonella typhimurium (strain LT2)), BIPA_ECO27 (Escherichia coli O127:H6 (strain E2348/69 / EPEC)),
BIPA_HELPY (Helicobacter pylori), BIPA_SHIFL (Shigella flexneri) и SVR3_ARATH (Arabidopsis thaliana).
Выравнивание проводилось, с помощью EMBOSS и программы muscle (подробные шаги изложены в подсказках к пр9 на kodomo).
В программе Jalview колонки были покрашены по идентичности, в связи с высокой консервативностью последовательностей, параметр conservation был выставлен 100%.
Множественное выравнивание
Выравнивание для всех белков прошло успешно, только у SVR3_ARATH (Arabidopsis thaliana) наблюдается отклонение от общего патерна, в виде вставки в последовательность дополнительных 79 а.о., что неудивительно, так как Arabidopsis thaliana
единственный представитель эукариот. Несмотря на это, в выравнивание встречается большое количество консервативных участков.
Приведу наиболее длинные из них:
Идентичные гомологичные участки находятся в позициях 89-100;
120-134 (за исключением позиции 127);
151-164 (с негомологичной заменой у Арабидопсиса в положение 166 A на S);
298-302 (замена глутамина на аланин в 299 положение у Арабидопсиса и в 300 у Хеликобактера)
347-350 (замена изолейцина на лейцин в позиции 350 у Bacillus subtilis)
524-528 (замена только у Арабидопсиса изолейцина на лейцин)
435-439; 451-454; 657-660 полностью идентичны
Участки со слабой гомологией: 250-262; 307-246; 356-380; 668-688
Обобщая, можно сказать, что данные белки гомологичны и имеют много консервативных участков, такое сходство объяснимо
функцией данных белков. Факторы, участвующие в сборке рибосом, должны действовать безошибочно и точно, иначе клетка окажется нежизнеспособной, в связи с этим мутации в данных последовательностях редко закрепляются.
Отдельно стоит сказать про Arabidopsis thaliana, он сильнее выбивается из общей канвы, так как является представителем эукариот, рибосомы которых отличаются от таковых в прокариотах.