Работа с алгоритмами поиска BLAST
Поиск ∆-субъединицы АТФсинтазы
Результатом текствого поиска по файлу с белковыми последовательностями организма Caenorhabditis elegans является
белок с accession NP_495286 и файл с последовательностью белка.
Этот белок закодирован на второй хромосоме, идентификатор которой NC_003280.
В геномном браузере на сайте NCBI был найден участок соответсвующий гену, кодирующему ∆-субъединицу АТФсинтазы:
Название гена: F58F12.1
Локализация гена: 6,383,645..6,384,692
Accession нуклеотидной последовательности: NM_062885.8
Файл с последовательностью ДНК, кодирующей этот белок и ее окрестности
Соседний ген: glam-3, кодирующий GLia Associated Membrane protein.
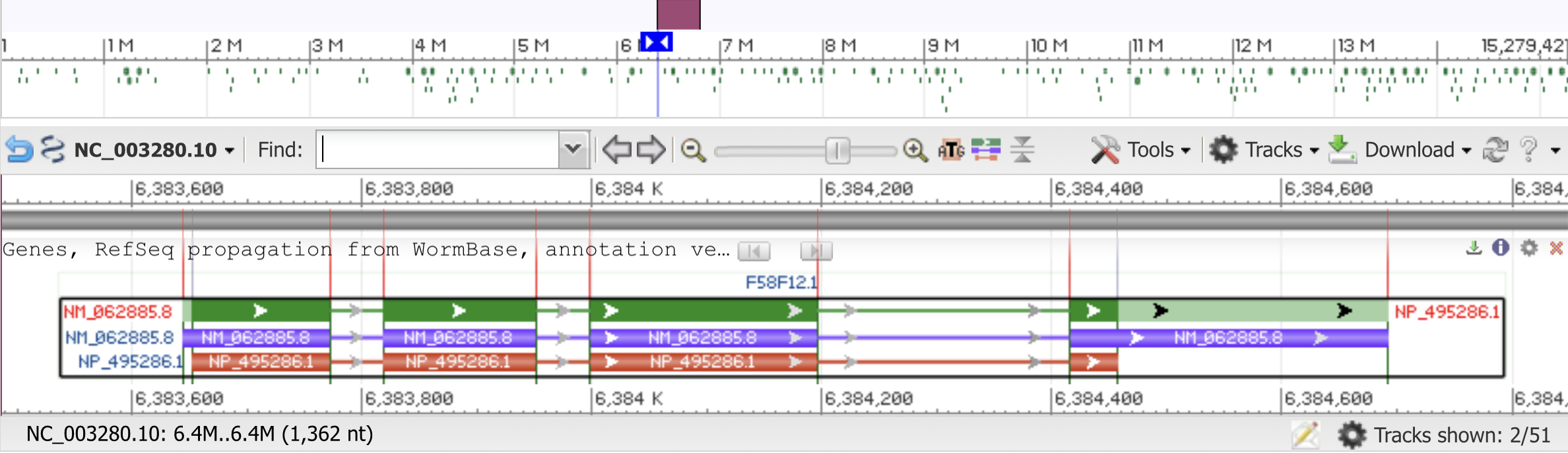
Рис. 1. Локализация гена F58F12.1 на хромосоме NC_003280 и коориднатами [6,383,645..6,384,692], кодирующего белок NP_495286.

Рис. 2. Окрестность гена F58F12.1, рядом расположен ген glam-3 и некодирующая РНК.
Проведение BLAST
Для найденной нуклеотидной последовательности проведен blastn и megablast для поиска гомологичных последовательностей ∆-субъединицы
АТФсинтазы из сем. Собачьи (Canidae) (намерено был выбран таксон достаточно отдаленный от изучаемого организма). Это семейство имеет в базе refseq genomes 7 сборок, с которыми и будет сравниваться наша исследуемая последовательность.
megablast и nblast
Проведение megablast со стандартными настройками (word size = 28) и с понижением параметра word size до 16 не позволило найти какие-либо схожие последовательности.
Это объясняется принципом работы megablast, который ищет сильно совпадающие последовательности, нахождение которых у отдоленных групп маловероятно.
Далее был произведен поиск с помощью blastn. Сперва со стандартными настройками (word size = 11), найти находки не удалось. Потом с понижением word size до 7, было найдено 4 находки, с максимальным весом 50.9 битов.

Рис. 3. Графическая интерпритация результата работы blastn. Зеленым обозначены совпадения с весом 50-80 битов, синим 40-50.
Проанализировав результат работы blastn можно сказать что он не подходит под данную задачу, так как находит мало хитов, с низким весом и очень маленькой длиной совпадающих участков.
Таблица с результатами
tblastn
После этого мы произвели поиск с помощью программы tblastn (word size = 5), было найдено 8 находок с максимальным весом 90.5 битов. Количество находок, превышающее количество сборок в refseq_genomes объясняется тем, что у одной и той же находки могут быть совпадающие последовательности к примеру на разных хромосомах.
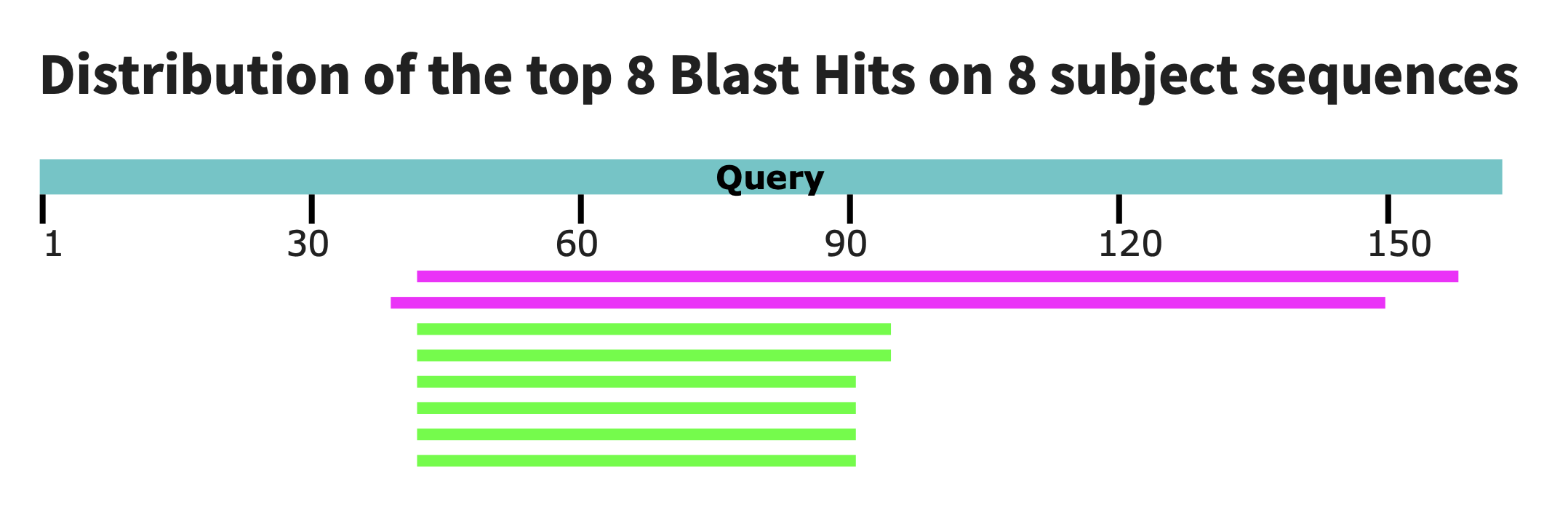
Рис. 4. Графическая интерпритация результата работы tblastn. Зеленым обозначены совпадения с весом 50-80 битов, розовым 80-200.
Таблица с результатами
Лучшая находка оказаль у вида Vulpes vulpes, ее вес 90.5 бита, а длина 120 аминокислот, что соответвует примерной длине этой белковой субъединицы, поэтому можно сделать вывод, что участки действительно гомологичны.
Оценив приведенные результы, можно сделать вывод, что tblastn работает эффективнее, выдает больше находок, с большим весом и сильно большей длиной, так как идет сравнение по аминокислотной (а не нуклеотидной) последовательности, которая более консервативна.
Поиск генов рРНК по далекому гомологу
Для поиска генов рРНК сперва необходимо проиндексировать последовательность генома Caenorhabditis elegans. Эту задачу мы волним с помощью следующего кода:
makeblastdb -in genom_WBcel -dbtype nucl
Для провидения blast были скачаны последовательности рРНК E. Coli, они будут подаваться на вход программе как query. Использовался blastn, так как он хорошо подходит для выравнивания высоко консервативных последовательностей, какими и являются гены рРНК.
blastn -task blastn -query rRNA_ecoli.txt -db genom_WBcel -out result_1
Текстовый результат бласт
blastn -task blastn -query rRNA_ecoli.txt -db genom_WBcel -out result_1_7 -outfmt 7
Табличный результат бласт
C помощью алгоритма было найдено 10 находок для 16S РНК, из приведенных концов выравненных участков можно сделать вывод, что всего найдено 5 разных гомологов.
Из которых достаточно большим весом и низким e-value обладают 2 гомолога.
Для последовательности NC_003279.8 с самым высоким score и низким e-value было найдено 6 совпадающих участков соответсвующих одному гомологу.
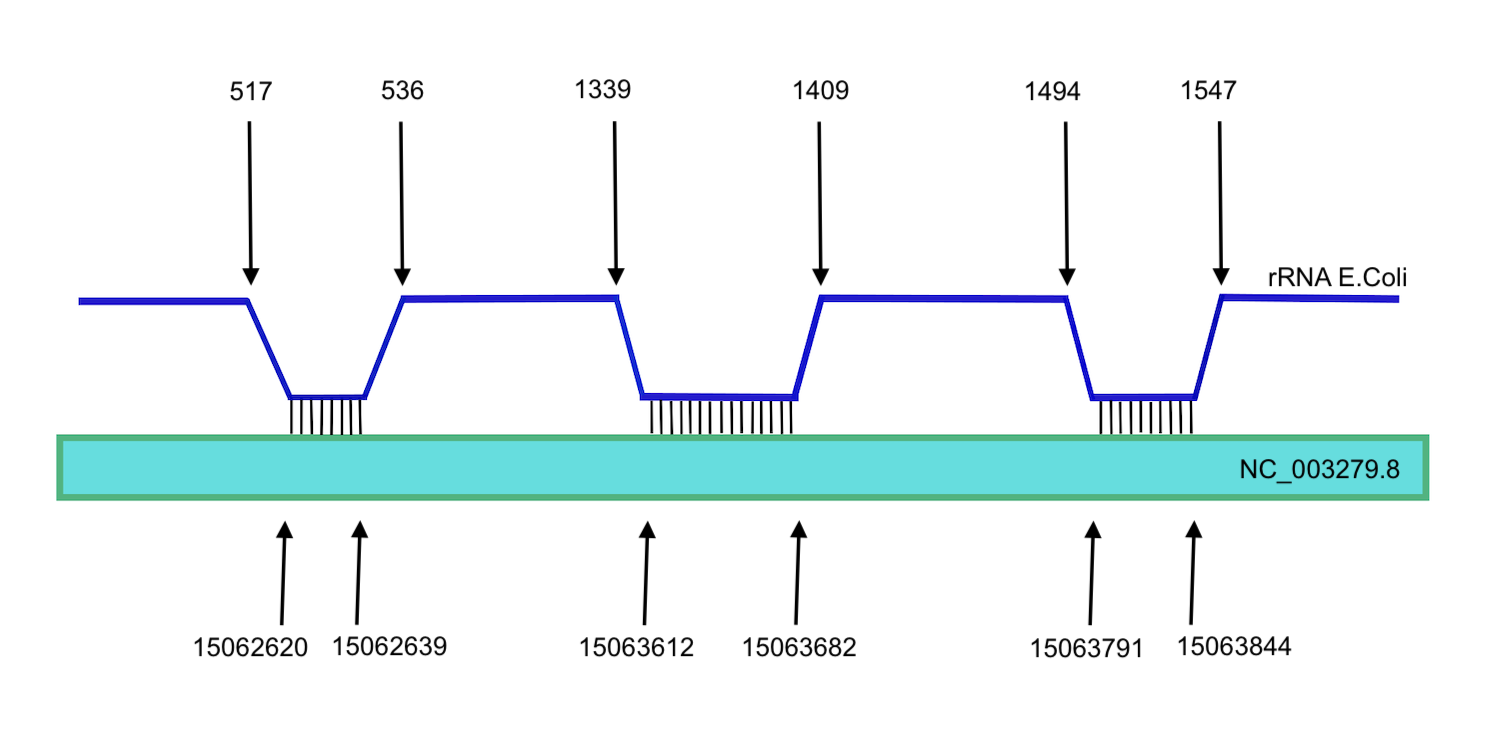
Рис. 5. Схема гомолога 16S РНК, цифрами обозначены концы участков выравнивания
Для 23S РНК было найдено 13 находок, из которых можно выделить 7 потеницальных гомологов, однако, обращая внимание на
вес и e-value находки, можно выделить только 2 гомолога.
Карты локальных сходств
Для данного блока практикума я выбрал две близкородственных археи Haloferax volcanii DS2 и Haloferax mediterranei ATCC 33500 их accession код хромосом в GenBank cоответсвенно CP001956.1 и CP039139.1. Эти археи
являются галофильными, то есть живут в средах с большим количеством солей, к пр. в Мертвом море. Эти организмы достаточно легко для экстремофилов культивируются в лабораторных условиях, а также способны из-за своей близости к генетическому обмену друг с другом.
Сперва мы провели blastn и megablast при стандартных параметрах на сайте NCBI для двух последовательностей.
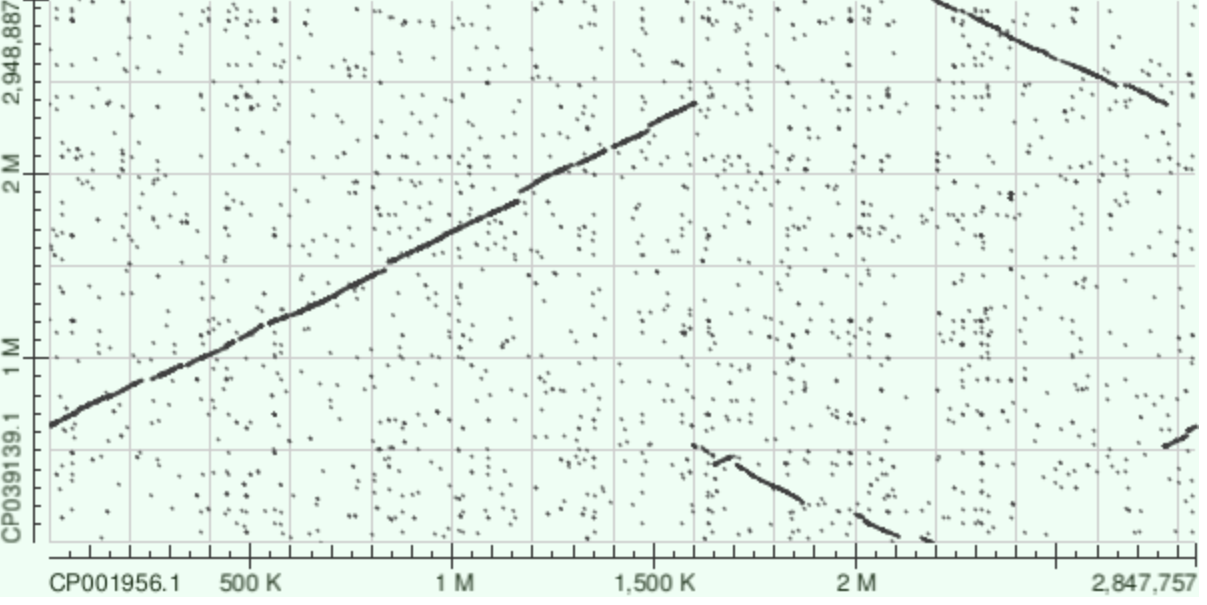
Рис. 6. Карта локальных сходств, построенная алгоритмом blastn

Рис. 7. Карта локальных сходств, построенная алгоритмом megablast
Оранжевые стрелочки - делеции, фиолетовые стрелочки - мисматчи, синии квадраты - инсерция + транслокаци, розовые - ранслокация, красный - инсерция, зеленый - разные точки начала секвенироваиня последовательностей.
В целом эти карты достаточно схожи, но карта blastn зашумленнее.
Для проведения tblastx пришлось взять последовательности двух плазмид (CP000451.1 и CP000452.1) из генома бактерии Nitrosomonas eutropha C91 (c которой я работал в предыдущих семестрах), так как на сайте NCBI не удается провести tblastx для больших последовательностей.
Принцип работы этого blast строится на транслировании последовательностей ДНК с 6 рамок считывания, и последующее сравнение их аминокислотных последовательностей, поэтому результат работы должен быть точнее, и в том числе карта локального сходства должна быть полнее, это мы и наблюдаем сравнивая карту, построенную с blastn/megablast и tblastx. Для работы программы использовался генетический код №11, характерный для бактерий.

Рис. 8. Карта локальных сходств плазмид, построенная с помощью blastn/megablast (одинаковый результат)
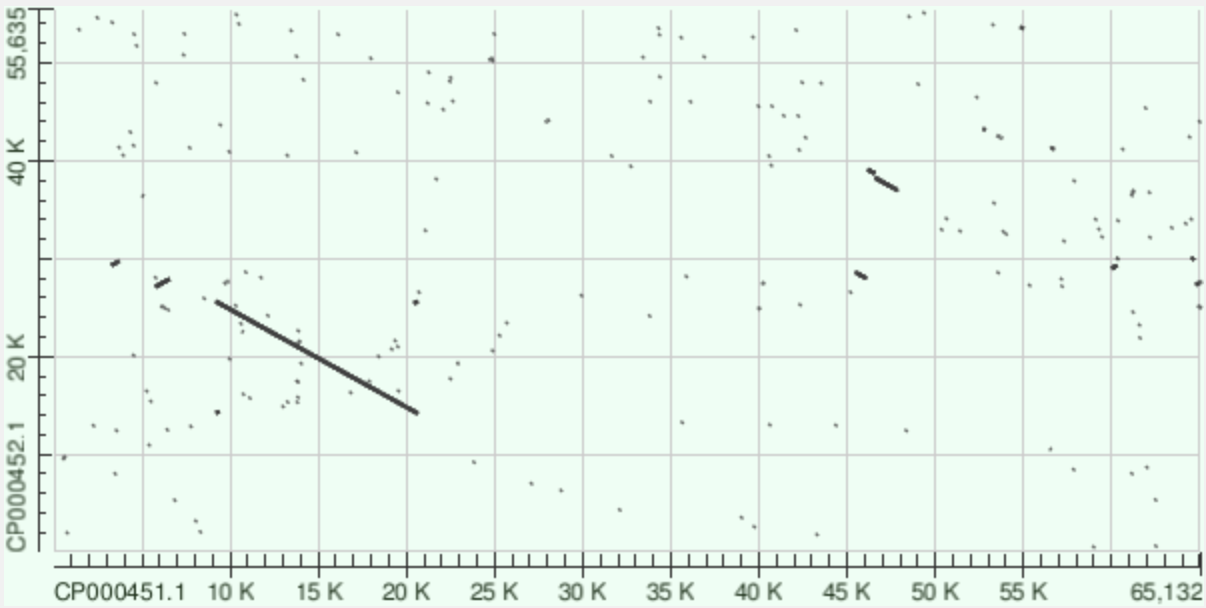
Рис. 9. Карта локальных сходств плазмиды, построенная с помощью tblastx