|
 |
 |
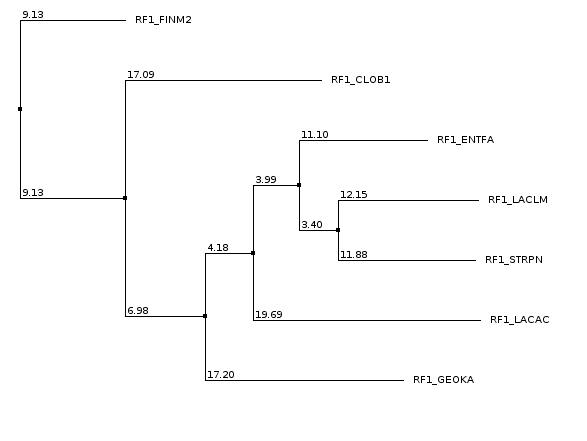
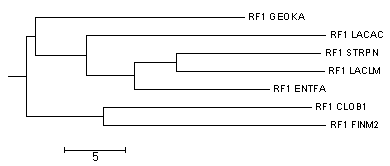
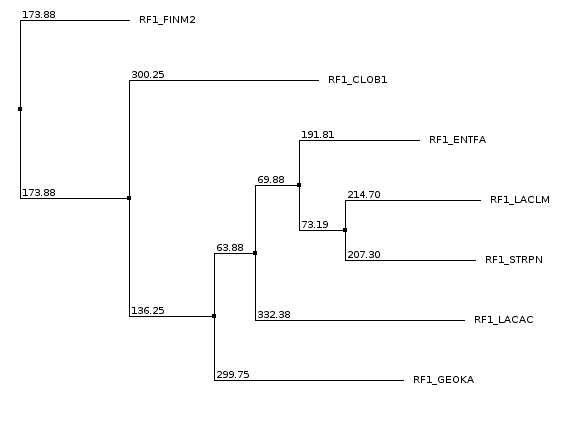
Укоренение в среднюю точку, посторенных методом neighbor-joining.
| Neigbour joining using % Identity | Neigbour joining using BLOSUM62 | ||
|---|---|---|---|
| Начальное дерево | После укоренения | Начальное дерево | После укоренения |
 |
 |
 |
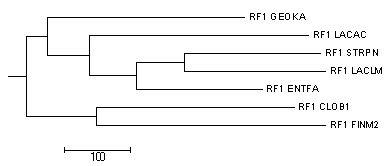 |
Деревья, построенные методом "Maximum parsimony" невозможно укоренить в среднюю точку, так как данный метод не реконструирует длины ветвей. Однако можно воспользоваться укорением с помощью внешней группы.
Реконструируем методом "Maximum parsimony" укорененное дерево выбранных бактерий, используя в качестве внешней группы белок rf1_ecoli.
Добавим в файл с предыдущими а/к последовательностями последовательность белка rf1_ecoli и получим файл. Данный файл откроем в JalView и выполним выравнивание Muscle with Defaults. В результате получим выравнивание. Этот файл(предварительно отредактировав) откроем в MEGA, укореним методом "Maximum parsimony" в ветвь, содержащую ECOLI (Substree → Root). Чтобы получить изображения укорененного дерева без ECOLI, воспользовались кнопкой "Show Subtree Separately". В итоге получили дерево:
Данное дерево совершенно не отличается от правильного дерева. Укоренение произошло в ветвь {RF1_CLOB1;RF1_FINM2}vs{RF1_GEOKA;RF1_ENTFA;RF1_STRPN;RF1_LACLM;RF1_LACAC}.
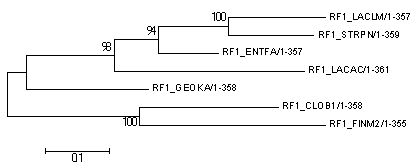
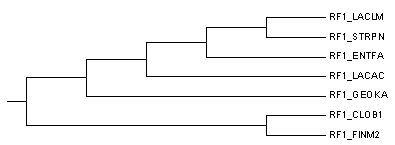
Провели бутстрэп-анализ филогении своих белков, используя метод "Maximum Likilihood" программы MEGA. Для этого в меню "test of Phylogeny" выбрали "Bootstap method" с числом реплик 100.
В итоге получили 2 идентичных дерева, которые не отличаются от правильного дерева. Ниже приведено "Original tree", так же можно посмотреть Bootstrap consensus tree":