|
 |
 |
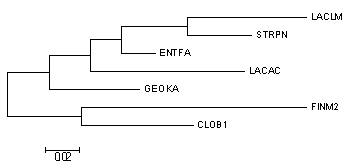
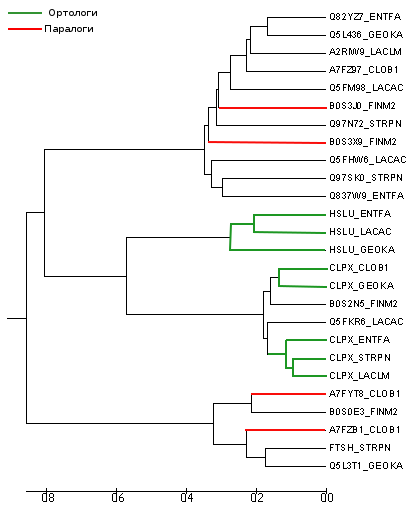
| Организм | Мнемоника | AC в EMBL | Координаты | Последовательность(+/-) |
|---|---|---|---|---|
| Finegoldia magna | FINM2 | AP008971 | 611796..613319 | + |
| Clostridium botulinum | CLOB1 | CP000726 | 63040..63541 | + |
| Geobacillus kaustophilus | GEOKA | BA000043 | 10421..11973 | + |
| Enterococcus faecalis | ENTFA | AE016830 | 248466..29987 | + |
| Lactobacillus acidophilus | LACAC | CP000033 | 59255..60826 | + |
| Lactococcus lactis | LACLM | AM406671 | 511423..512971 | + |
| Streptococcus pneumoniae | STRPN | CP001845* | 15355..16888 | + |