


 |
 |
 |
Задания
1.Для анализа была выбрана цепь A уже рассматриваемой структуры 2-кето-3-дезоксиглюконат альдолазы(1w37), а для аннтотации вторчной структры была использована программа Stride. В итоге запуска на сервисе был получен файл-аннотация.
В таблице.1 ниже представлено сравнение PDB-аннотации и Stride-аннтации α-спиралей.
Табл.1: Сравнением аннтотаций α-спиралей цепи А в PDB и Stride.
| PDB | Stride | Длина спирали | PDB | Stride | Длина спирали в pdb |
|---|---|---|---|---|---|
| 19-33 | 20-33 | 15 | 160-171 | 161-170 | 12 |
| 42-46 | 43-45 | 5 | 180-182 | — | 3 |
| 46-50 | 47-49 | 5 | 183-190 | 183-190 | 8 |
| 51-64 | 52-64 | 14 | 198-202 | 198-201 | 5 |
| 77-88 | 78-88 | 12 | 203-219 | 204-216 | 14 |
| 89-91 | — | 3 | 218-238 | 219-237 | 21 |
| 108-123 | 109-122 | 16 | 240-254 | 241-253 | 15 |
| 132-137 | 133-136 | 6 | 268-289 | 269-289 | 22 |
| 141-149 | 142-148 | 9 |
Как видно из таблицы, из 17 спиралей сошлись результаты только у спирали 183-190. Спирали меньше 3 аминокислотных остатков не находились, а остальные имеются неточное предсказание начала и конца. При более подробном рассмотрении случаев видно, что в 3х случаях (52, 133, 204 а.о.) аннотированная с помощью Stride спираль начинается с пролина, что наиболее предпочитительно, чем пролин внутри α-спирали. Например, спираль образованная с 51-64 а.о. в pdb имеет перед пролином остаток серина, у которого углы φ и ψ равны соответственно -66.12° и 163.64°. Если рассмотреть карту Рамачандрана для серина, то данная точка будет лежать на границе допустимой области, а также если рассмотреть ход цепи, то видно, то серин уже "вывернут" из хода спирали.(Рис.1)
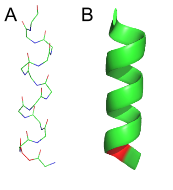
Рис.1: Изображением α-спирали с 51-64 а.о., красным обозначен Pro52. A: Остовная модель.B: модель cartoon.
В выбранной мной структуре нет явных β-листов, но есть бета бочонок. По результатам Stride были предсказаны все листы, аннотированные в PDB, однако в выходных данный программы также содержатся 2 листа, по 2 а.о. (12-13, 17-18).(РИс.3)
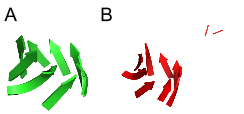
Рис.2: Изображением β-бочонка. A: аннотация PDB, B: аннотация Stride.
Программа SheeP однаружила β-баррель, содержащийся в цепи А структуры 1w37.
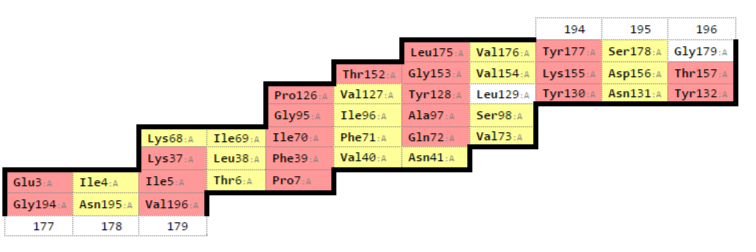
Рис.3: Карта β-листа, полученная программой SheeP.
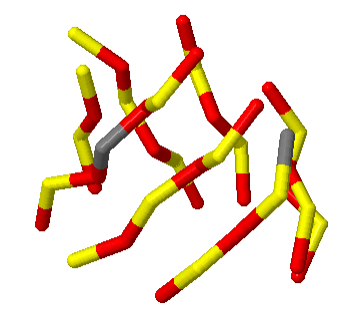
Рис.4: Изображение бочонка, с рацветкой соотвествующей карте.
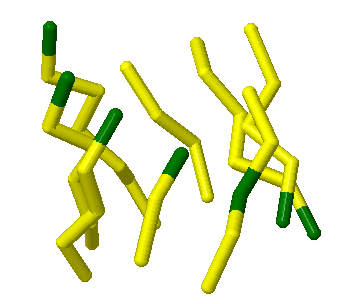
Рис.5: Изображение хребта(зеленым), образованного аминокислотами Leu175, Gly153, Tyr128, Ala 97, Gln72, Asn41, что соответствует пятому сторбцу справа в карте.
для определенения гидрофорбной стороны были изображены разным цветом гиброфобные и гидрофильные остатки, однако это не улучшило понимание.(Рис.6)
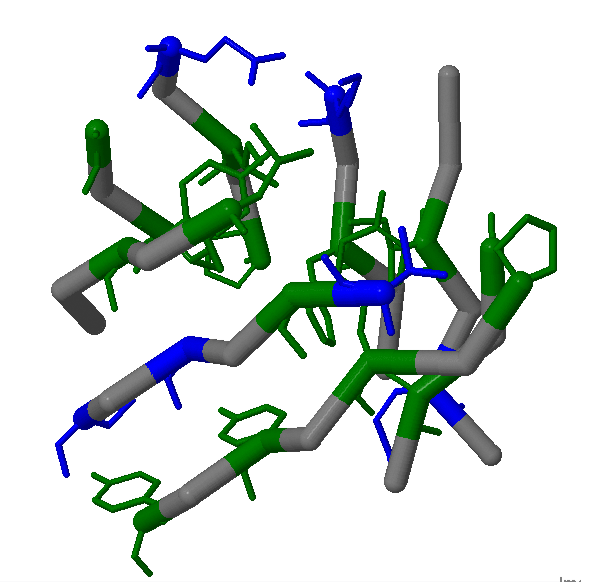
Рис.6: Изображение бочонка, где остатки гидрофобных а.к. - зеленые, гидрофильных - синие.
по полученным изображениям можно сделать вывод, что "канал", образованный бочонком более гидрофобен, однако встечаются и гидрофильные остатки. Следовательно, точно определить расположение гидрофильной стороны не удается.
Для совмещения были выбраны 5 из 6 гомологов. описанный в первом практикуме, а именно: 1nal, 3lcf, 3ird, 3e96, 3tuu. При визуализации совмещения структур оказалось, что действительно эти модели похожи.(Рис.7)
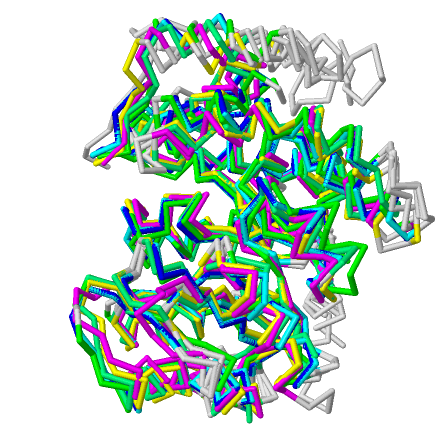
Рис.7: Совмещение выбранных гомологов и цепи А исходной структуры 1w37, изображенных разным цветом.
При рассмотрении выравниваний по структуре и последовательности нельзя сказать, что они идентичны. Например, разберу случай выравнивания участка 163-177 а.к. исходной структуры.(Рис.8)
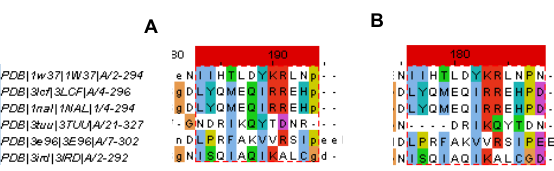
Рис.8: Выравнивания, размеченные по Crustalx, по структуре(A) и последовательности, полученное методом Muscle(B).
Выравнивание Ile и Leu не имеет никаких сомнений в правильности, но почему к ним выраниваниется Asn не совсем понятно, аналогичный случай,но уже в следующей колонке, с Pro. По моему мнению, выравнивание в третьей и четвертой позиции аминокислот достаточно достоверно. Если же посмотреть на все выравнивание участка в целом, кажется, что допущенные гэпы (в выравнивании по последовательности) ради хорошего выравнивания лизина в 8ой позиции являются нелогичными. И в целом складывается общее впечаление, что на данном участке выравнивание по структуре лучше, чем по последовательности. Однако, если посмотреть совмещение на этом участке, то видно, что структура 3tuu отстоит в пространестве от общего совмещения структур.(Рис.9) Хотя внешне она похожа на совместившиеся участки, отстоящие от нее. Поэтому, я считаю, что выравнивание по структуре в данном случае лучше, чем по последовательности.
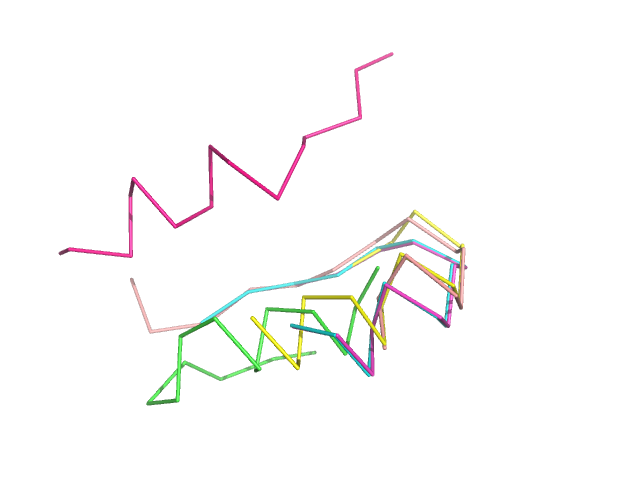
Рис.9: Совмещение стркуктур на участке с 163 по 177 аминокислоту представлено в виде остова и окрашено по цепям.
Для данной работы была выбрана структура 1qse, в SCOP обозначена α цепь, как участок с 188 по 206 а.о. цепи D, а β цепь - c 119 по 246 цепи E.С помощью серивиса SheeP было выделены β-листы в α и β цепях.(Рис.10)
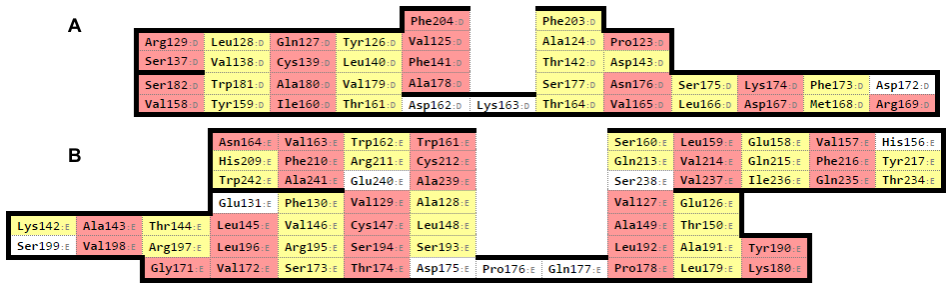
Рис.10: Карты β-листов в цепочке α(A) и β(B)
Консервативный остаток цистеина в α цепи - 139, β цепи - 147.
Для совмещения использовались команды:
select al, alpha and (resi 137-143 + resi 174-181) and name ca select be, beta and (resi 143-150 + resi 191-197) and name ca color red, al color blue, be pair_fit al, be
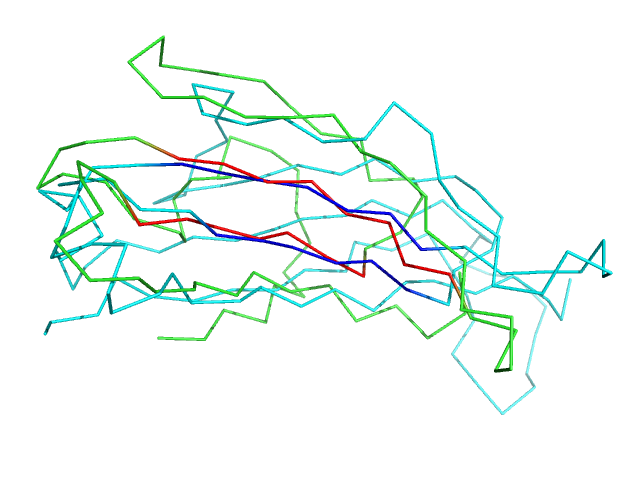
Рис.11: Совмешещение α(зеленый) и β(циановый) цепей по участкам, обозначенными красным и синим.
RSM difference данного совмещение равно 1.748 (15 из 15 атомов). Топология у данных структур похожи, однако есть и выпепливания и новые элементы вторичной структуры(с 134 по 140 и с 200 по 204 β цепь имеет спираль вместо несктруктурированного участка цепи α).
Файл с совмещением
Для нахождения гиброфодных кластеров была взята цепь А уже изучаемой структуры 1w37 и рассмотрены случаи поиска при разных парамертах:
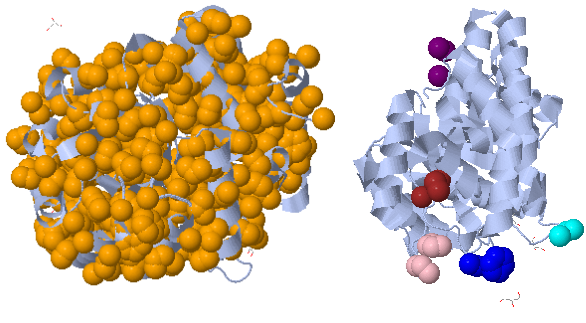
Рис.12: Выделенные гидрофобные кластеры в цепи А структуры 1w37, оранжевый - 644 атома, синий - 9, розовый - 7, бордовый, фиолетовый, циановый по 3 атома..
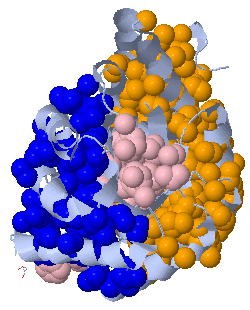
Рис.13: Выделенные гидрофобные кластеры в цепи А структуры 1w37, оранжевый - 279 атома, синий - 128, розовый - 94.
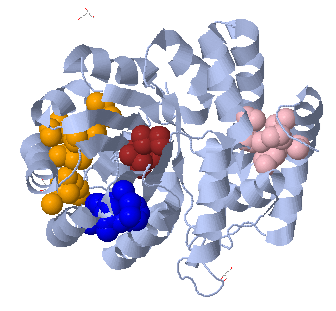
Рис.14: Выделенные гидрофобные кластеры в цепи А структуры 1w37, оранжевый - 18 атомов, синий - 23, розовый - 17 и бордовый - 16.
Для поиска гиброфобных кластеров на интерфейсах двух цепочек взяли цепи A и C структуры 1w37. При поиске с порогом 4.5 Å и минимальным количеством атомов 3 был найден всего один кластер, находящийся меджу двумя сбъединицами и состоящий из 12 атомов.(Рис.15) Также были найдены небольшие кластеры на границе каждого белка, поэтому было решено найти кластеры во всем гомотетрамере с теми же параметрами.
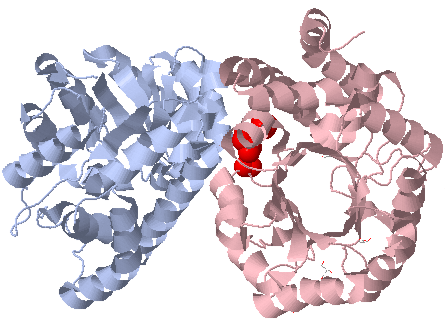
Рис.15: Выделенный гидрофобный кластер между цепями А и С структуры 1w37.
В результате среди 30 найденных класстеров всего 3 находились между субъединицами, а именно уже найденный на предыдущем шаге кластер между цепями A и C в 12 атомов, а также 2 кластера между цепями B и D по 4 атома.(Рис.15)
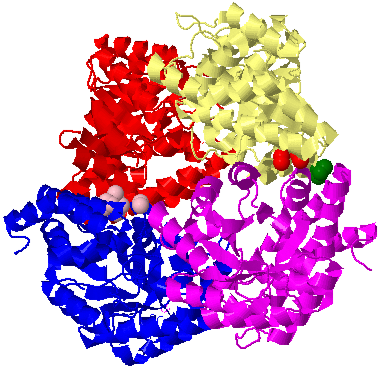
Рис.15: Выделенные гидрофобные кластеры между всему субъединицами структуры 1w37, розовый - 12 атомов, зеленый и красный по 4 атома.
Струкрура 2PUB состоит из молекулы транскрипционного фактора PurR и молекулы ДНК.(Рис.16)
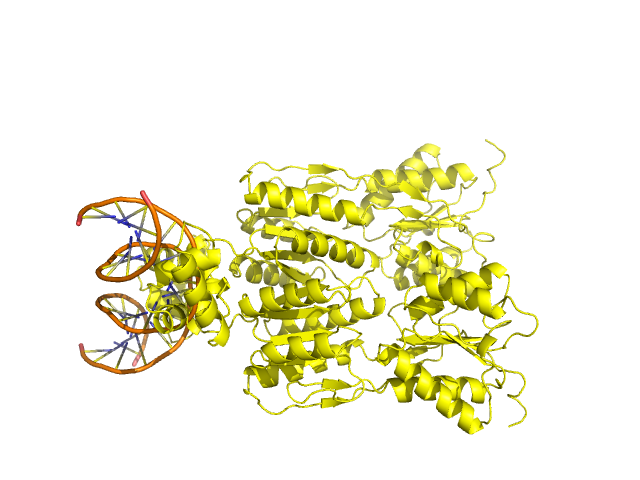
Рис.16: Изображение структуры 2PUB. Красным в виде cartoon показан транскрипционный фактор PurR.
Так как структрура содержит только мономерный белок и одноцепочечную молекулу ДНК, то есть всего две поверхности контакта: поверхность ДНК, контактирующая с белком и поверхность белка, находящаяся в контакте с ДНК.(Рис.17-18)
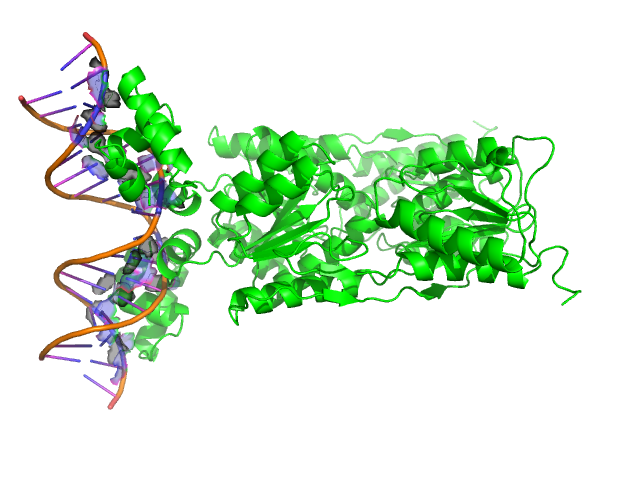
Рис.17: Изображение структруты 2PUB, где синим цветом показана поверхность ДНК, контактирующая с белком.
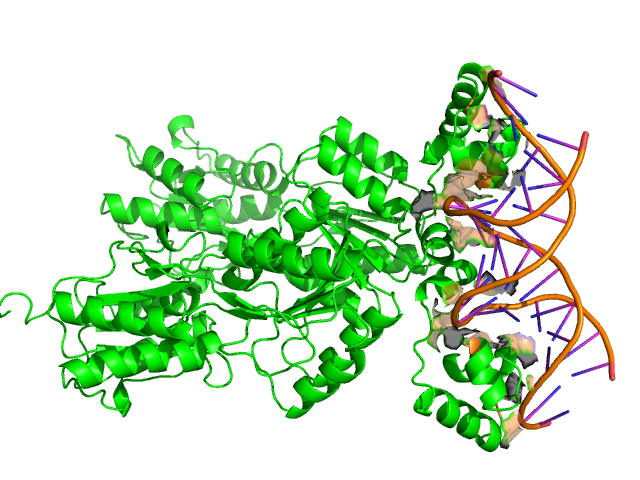
Рис.18: Изображение структруты 2PUB, где оранжевым цветом показана поверхность белка, контактирующая с ДНК.
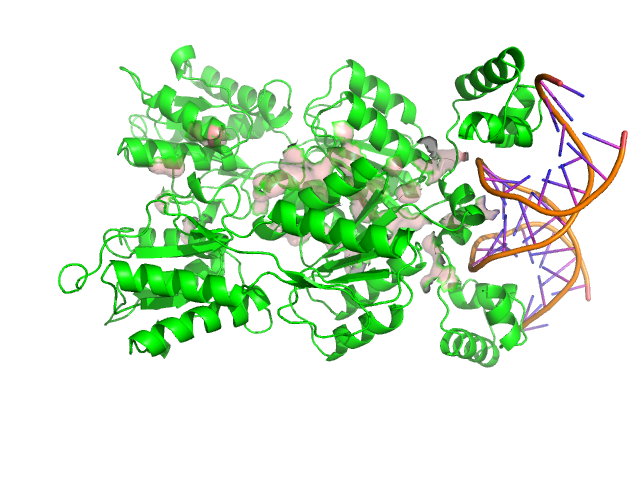
Рис.18: Изображение структруты 2PUB, где розовым цветом показана поверхность мономера, контактирующая с другим мономером.
Используя сервис CluD, были определены гиброфобные кластеры размером не менее 10 атомов на интерфейсе мономеров белка в рассматриваемом комплексе, а также порог расстояния - 5.0 Å. Было найдено 3 кластера.(Рис.19)
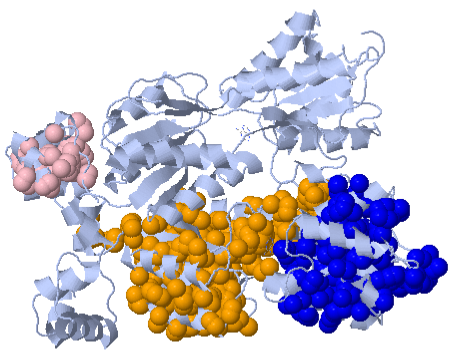
Рис.19: Изображение гиброфобных кластеров мономеров белка структруты 2PUB.
Так как не удалось отобразить атомы, полученный гидрофобных кластеров, то вместо этого изобразили поверхность контакта цветом так, что чем сильнее красный оттенок, тем гидрофобнее участок(Рис.20) с помощью скрипта color_h (Author(s): Eisenberg D., Schwarz E., Komarony M., Wall R., Reference: J. Mol. Biol. 179:125-142 (1984)).
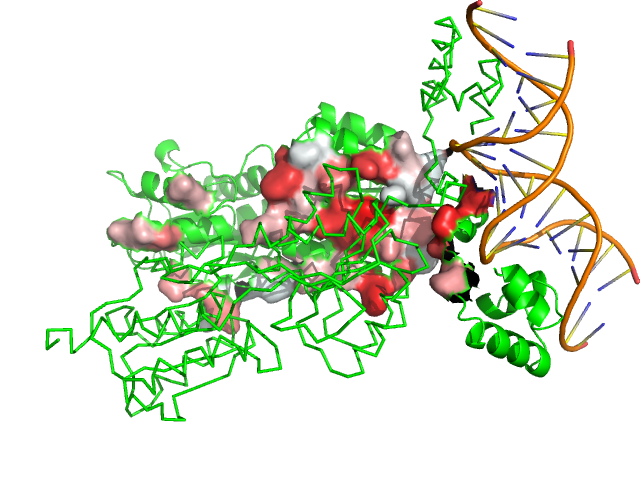
Рис.20: Изображение по гидрофобности а.о. поверхности контактов мономеров белка структруты 2PUB.
Для сравнения доменов была выбрана (оставлена из предыдущих заданий) структура 2-кето-3-дезокси глюконат альдолазы, 1w37. В SCOP данная структура обозначена, как один домен (110360) на каждую цепь-субъединицу белка, принадлежит к классу альдолаз первого типа и укладке TIM-баррель (Рис.21). В СATH также почти вся структура занесена в домен 1w37A00, имеющая границы с 2 по 294 а.о.(Рис.22) В Pfam тоже находится один домен (PF00701) с границами 1-282 а.к.(Рис.23)
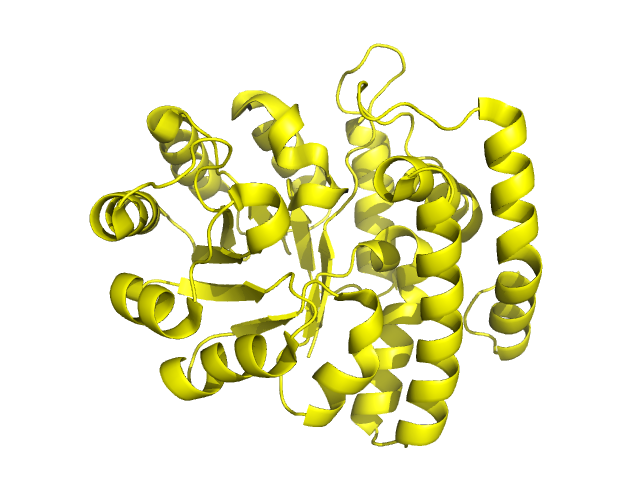
Рис.21: Изображение домена 110360 SCOP в структуре 1w37
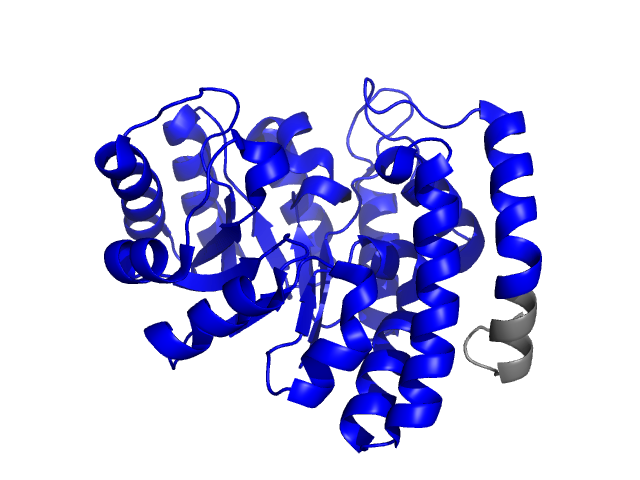
Рис.22:Изображение домена PF00701 Pfam в структуре 1w37
Рис.23: Изображение домена 1w37A00 CATH в структуре 1w37
Интересным фактом, по моему мнению, является то, что Pfam не включает участок последовательности из 12 аминокислот, когда большаяя его чать является α-спиралью.
© Butusova Anna,2014