Обзор особенностей генома бактерии Octadecabacter antarcticus 307
Гагарочкин Виталий Александрович
Факультет биоинженерии и биоинформатики
Московского государственного университета имени М.В. Ломоносова
АННОТАЦИЯ
Данный обзор посвящён изучению и анализу особенностей генома бактерии Octadecabacter antarcticus 307, выделенной из антарктического морского льда. Были изучены гены бактерии и их расположение на хромосоме и плазмиде, осуществлена попытка предсказания оперонов. Подробно рассмотрена асимметрия нуклеотидного состава репликонов и осуществлён поиск консервативных последовательностей.
ВВЕДЕНИЕ
Octadecabacter antarcticus 307 — грамотрицательная, гетеротрофная, психрофильная бактерия, выделенная из нижних 25-50 см антарктического морского льда пролива Мак-Мердо недалеко от острова Росса [1]. Как и для всего рода Octadecabacter, для Octadecabacter antarcticus 307 характерно высокое содержание октадеценовой кислоты (18:1) — более 70% от общего количества жирных кислот в клетке бактерии, а также формирование внутриклеточных газовых везикул [2].
Систематическое положение Octadecabacter antarcticus 307 [3]:
- Домен: Bacteria
- Филум (отдел): Pseudomonadota
- Класс: Alphaproteobacteria
- Порядок: Rhodobacterales
- Семейство: Roseobacteraceae (до 2022 года Rhodobacteraceae)
- Род: Octadecabacter
- Вид: Octadecabacter antarcticus
МАТЕРИАЛЫ И МЕТОДЫ
Для анализа были использованы таблица геномных особенностей, последовательности репликонов и CDS бактерии Octadecabacter antarcticus 307 геномной сборки GCF_000155675.2 (см. S1).
Распределение генов различных типов по репликонам бактерии Octadecabacter antarcticus 307
Для подсчёта количества генов каждого типа по репликонам были использованы методы электронных таблиц (см. S2).
Для генома бактерии ожидается наличие не менее трёх генов рРНК (5S-, 16S- и 23S-рРНК) и нескольких десятков генов тРНК. Однако количество генов тРНК, вероятно, будет меньшим, нежели количество кодонов, кодирующих аминокислоты (61 кодон), поскольку во взаимодействии 3-го нуклеотида кодона и 1-го нуклеотида антикодона наблюдается явление “вобблинга” (wobble base pairing), заключающееся в возможности формирования комплементарных пар, отличных от Уотсон-Криковских (Табл. 1) [4]. Также в первом положении антикодона может находиться инозин (дезаминированный аденозин), способный образовывать пары с адениловыми, уридиловыми и цитидиловыми нуклеотидами 3-й позиции кодона. За счёт этого одна молекула тРНК может соответствовать сразу нескольким кодонам, кодирующим одну аминокислоту.
| 5`-позиция антикодона | 3`-позиция кодона |
|---|---|
| A | U |
| C | G |
| G | C, U |
| U | A, G |
| I (инозин) | A, C, U |
Определение долей последовательностей репликонов бактерии Octadecabacter antarcticus 307, приходящихся на гены различных типов
Для подсчёта долей последовательностей репликонов, занимаемых генами каждого типа, были использованы методы электронных таблиц (см. S3).
Распределение длин возможных продуктов трансляции CDS бактерии Octadecabacter antarcticus 307
Для анализа были использованы методы электронных таблиц (см. S4).
Распределение CDS бактерии Octadecabacter antarcticus 307 по GC-составу
Для анализа были использованы методы электронных таблиц (см. S5).
Определение и анализ частот встречаемости старт- и стоп-кодонов в CDS бактерии Octadecabacter antarcticus 307
Для подсчёта числа старт-кодонов были использованы методы Bash (см. S6), стоп-кодонов — методы Python (см. S7). Старт-кодоны и стоп-кодоны были подсчитаны отдельно для CDS функциональных генов и для CDS псевдогенов.
Ожидается, что благодаря явлению “вобблинга” кроме каноничных старт-кодонов (ATG) и стоп-кодонов (TAA, TAG, TGA) в функциональных CDS могут быть обнаружены кодоны, отличающиеся от консенсуса на 1 нуклеотид в 1-й или 3-й позиции. Для CDS псевдогенов можно предположить большее разнообразие старт- и стоп-кодонов (точнее будет сказать, первых и последних триплетов), принимая во внимание их нефункциональность и предполагая для них нейтральную эволюцию [5].
Анализ межгенных промежутков и пересечений генов для хромосомы бактерии Octadecabacter antarcticus 307
Для анализа распределений длин межгенных промежутков и пересечений CDS были использованы методы электронных таблиц (см. S8). Для анализа участков последовательности хромосомы, содержащих пересечения CDS, были использованы средства Python (см. S9).
Ожидается обнаружение пересечений типа “голова к хвосту” (пересечение стоп-кодона одной CDS и стоп-кодона другой) длиной 1 или 4 п.н. для CDS, расположенных на одной цепи, а также пересечений типов “голова к голове” (пересечение старт-кодонов) длиной 2 п.н. и “хвост к хвосту” (пересечение стоп-кодонов) длиной 4 п.н. (Рис. 1) для CDS на разных цепях [6].
При этом для пересечений “голова к хвосту” ожидаются следующие варианты: TG[A]TG, TA[A]TG и [ATGA] (для пересечений на минус-цепи, соответственно CA[T]CA, CA[T]TA и [TCAT]); для пересечений “голова к голове”: C[AT]G; для пересечений “хвост к хвосту”: [CTAG], [TTAG], [CTAA] и [TTAA].
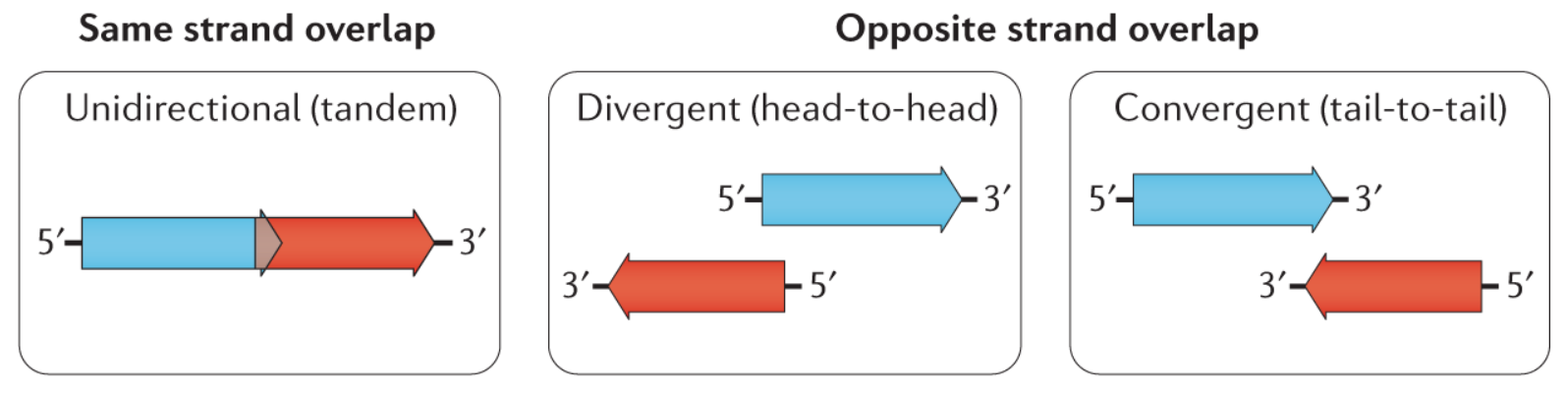
Стоит отметить, что перекрытия типа “голова к хвосту” часто встречаются в бактериальных оперонах, поскольку за счёт перекрытия достигается трансляционное сопряжение CDS оперона [6].
Предсказание оперонов в репликонах бактерии Octadecabacter antarcticus 307
Для предсказания оперонов в геноме бактерии были использованы методы Python (см. S10).
Группа CDS считалась потенциальным опероном, если соответствовала следующим критериям:
Все CDS группы расположены на одной цепи одного репликона.
Все CDS группы перекрываются со своими соседями по группе на 1 или 4 п.н.
Группа состоит из не менее, чем трёх CDS.
Поиск точек начала (origin) и терминации (termus) репликации в репликонах бактерии Octadecabacter antarcticus 307 путём анализа GC-перекоса и TA-перекоса (GC-skew и TA-skew)
Графики GC-перекоса и TA-перекоса были построены с применением методов Python (см. S11 и S12).
Перекос GC — явление, при котором в определённой области последовательности ДНК гуаниловые и цитидиловые нуклеотиды представлены не в равном количестве. Аналогичная ситуация для адениловых и тимидиловых нуклеотидов именуется перекосом TA.
Перекос GC в бактериальных хромосомах и плазмидах возникает вследствие особенностей работы репликативной вилки, в результате которой в последовательность ведущей цепи встраивается больше гуаниловых нуклеотидов, нежели цитидиловых (Рис. 2).
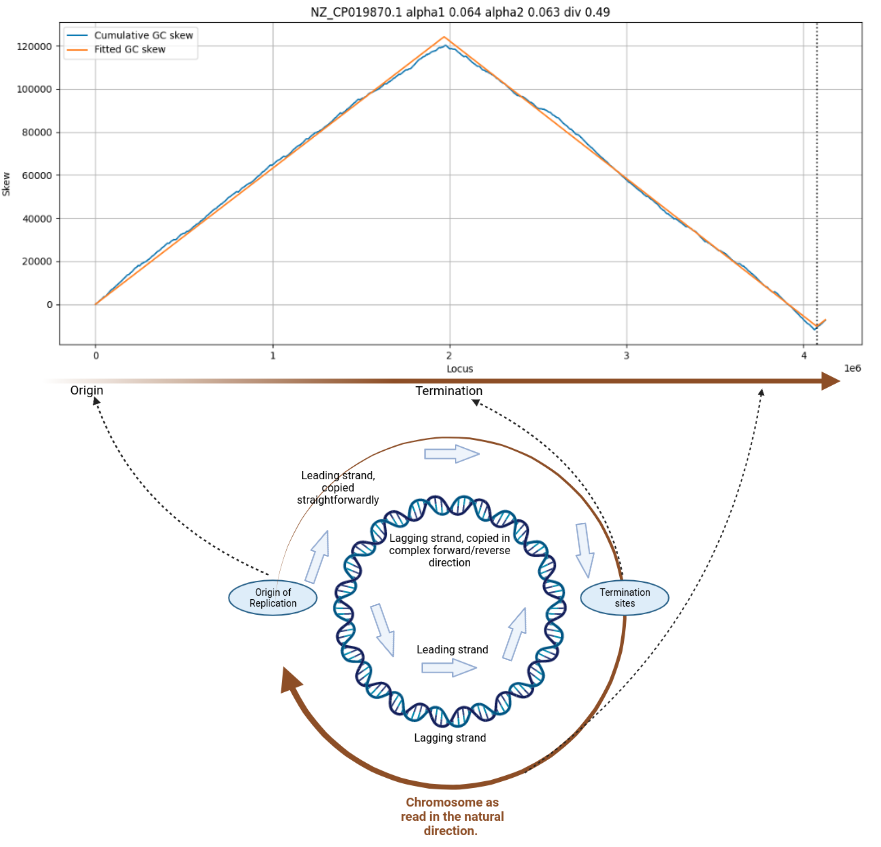
Но поскольку в точках начала и терминации репликации ведущая цепь становится отстающей и наоборот, в данных точках должна происходить инверсия GC-перекоса. Как следствие, анализ перекоса GC можно использовать для поиска точек начала и терминации репликации в бактериальных геномах [8].
Для анализа перекоса GC (или TA) можно использовать скользящее окно, тогда для него перекос GC будет рассчитываться по формуле:
\(\text{GC skew} = \frac{(G - C)}{(G + C)}\), где G и C — количества гуаниловых и цитидиновых нуклеотидов в окне. Для перекоса TA аналогично.
График, полученный таким способом, не всегда нагляден и сильно зависит от размера выбранного окна. Поэтому для поиска точек начала и терминации репликации чаще используются графики кумулятивного перекоса GC (TA).
Для построения графика кумулятивного перекоса GC также используется скользящее окно, но значение кумулятивного перекоса для n-го по счёту окна рассчитывается следующим образом:
\(\text{Cumulative GC skew} = \sum_{i = 1}^{n-1} \text{GC skew}_i\), где \(\text{GC skew}_i\) — это обычный перекос GC для i-го окна. Аналогично для перекоса TA.
График кумулятивного перекоса GC имеет два пика — максимальный и минимальный. Максимум соответствует точке терминации репликации, минимум — точке начала репликации.
Дальнейший анализ асимметрии в распределении нуклеотидов по последовательностям репликонов Octadecabacter antarcticus 307
Частоты встречаемости нуклеотидов были подсчитаны с применением методов Python (см. S13).
Частоты встречаемости нуклеотидов по группам подсчитывались следующим образом:
\(M = A + C\), \(K = G + T\)
\(R = A + G\), \(Y = C + T\)
\(S = G + C\), \(W = A + T\)
Для построения графиков несоответствия использовались методы Python (см. S14).
Несоответствия по группам нуклеотидов для позиции \(i\) рассчитывались по следующим формулам:
\(\text{GC disparity} = G_i - C_i\)
\(\text{TA disparity} = T_i - A_i\)
\(\text{MK disparity} = M_i - K_i\)
\(\text{RY disparity} = R_i - Y_i\)
\(\text{SW disparity} = S_i - W_i\)
Где \(N_i\) — количество нуклеотидов группы N в участке последовательности с первого нуклеотида по нуклеотид \(i\) включительно.
Z-кривая — способ визуализации последовательности ДНК в трёхмерном пространстве. При построении Z-кривой координаты позиции \(i\) определяются следующим образом: координата x — несоответствие RY, координата y — несоответствие MK, координата z — несоответствие SW [9].
Для построения Z-кривых использовались методы Python (см. S15).
График прохода по ДНК (DNA walk graph) также является способом визуализации последовательности, но в двухмерном пространстве. При построении графика прохода следующая точка смещается относительно предыдущей вверх, если следующий нуклеотид в последовательности — A, вниз — если T, вправо — если G и влево — если C [10]. Таким образом график прохода по последовательности можно построить, взяв по оси x несоответствие GC, а по оси y — несоответствие AT (несоответствие TA с обратным знаком).
Для построения графиков прохода по последовательностям репликонов бактерии использовались методы Python (см. S16).
Поиск консенсусных последовательностей в прилегающих к 5`-концу CDS областях в геноме Octadecabacter antarcticus 307
Поиск осуществлялся методом построения логотипа исследуемых последовательностей. Для построения логотипа использовались методы Python (см. S17).
Логотип последовательностей — метод графического представления информации о консервативности позиций в последовательностях.
Высота столбца для каждого нуклеотида в позиции \(i\) определяется следующими формулами:
\(r_{a,\text{ }i} = f_{a,\text{ }i} \cdot R_i\), где \(f_{a,\text{ }i}\) — частота данного нуклеотида в данной позиции, \(R_i\) — полная информация позиции.
\(R_i = \log_2 (4) - (H_i + e(n))\), где \(H_i\) — информационная энтропия Шеннона для данной позиции, \(e(n)\) — поправка, \(n\) — количество анализируемых последовательностей.
\(H_i = -\sum_{i = 1}^{n} f_{a,\text{ }i} \cdot \log_2 (f_{a,\text{ }i})\)
\(e(n) = \frac{1}{\ln (2)} \cdot \frac{4 - 1}{2n}\)
Таким образом по высоте столбцов можно судить о консервативности позиции в последовательности: чем выше столбец определённого нуклеотида, тем более консервативным является его наличие в данной позиции [11].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Распределение генов различных типов по репликонам бактерии Octadecabacter antarcticus 307
Были подсчитаны количества генов, кодирующих белки и РНК разных типов, а также псевдогенов для каждого репликона бактерии Octadecabacter antarcticus 307 (Табл. 2).
Можно заметить, что все гены, кодирующие различные нематричные РНК локализованы на хромосоме бактерии, в плазмиде же закодированы лишь 68 CDS, из которых белковый продукт имеют 61 CDS.
В геноме бактерии, как и ожидалось (см. “Материалы и методы”), имеется 45 генов тРНК, что меньше, чем количество кодонов, кодирующих аминокислоты (61 кодон). Этот факт объясняется тем, что за счёт явления “вобблинга” в 3-м положении кодона один антикодон может соответствовать нескольким различающимся по 3-му положению кодонам.
| Тип гена | Хромосома | Плазмида | Всего |
|---|---|---|---|
| Функциональные CDS | 4 444 | 61 | 4 505 |
| CDS псевдогенов | 513 | 7 | 520 |
| tRNA | 45 | 0 | 45 |
| rRNA | 6 | 0 | 6 |
| tmRNA | 1 | 0 | 1 |
| ncRNA | 2 | 0 | 2 |
| Всего | 5 011 | 68 | 5 079 |
Определение долей последовательностей репликонов бактерии Octadecabacter antarcticus 307, приходящихся на гены различных типов
Были рассчитаны доли последовательности, занимаемые генами разных типов для репликонов бактерии Octadecabacter antarcticus 307 (Табл. 3).
Как видно из полученных данных, межгенные промежутки и в хромосоме, и в плазмиде составляют менее 20% от последовательности репликона, что ожидаемо для генома бактерии. Также стоит отметить, что хотя количество генов тРНК значительно превышает количество генов рРНК (45 против 6), суммарная длина последовательностей генов рРНК больше, чем таковая для тРНК (0,184% длины репликона против 0,074%). Это наблюдение объясняется тем, что транспортные РНК Octadecabacter antarcticus 307 имеют длину от 74 до 91 п.н., а рибосомальные: 5S рРНК — 115 п.н., 16S рРНК — 1467 п.н., 23S рРНК — 2851 п.н. (см. S18) Причём ген каждой рРНК представлен в геноме бактерии двумя копиями, что может свидетельствовать о произошедшей в прошлом дупликации. Аналогичная картина наблюдается для генов некоторых тРНК.
| Тип гена | Хромосома | Плазмида |
|---|---|---|
| Функциональные CDS | 79,357% | 78,720% |
| CDS псевдогенов | 7,424% | 3,558% |
| tRNA | 0,074% | 0,000% |
| rRNA | 0,184% | 0,000% |
| tmRNA | 0,008% | 0,000% |
| ncRNA | 0,011% | 0,000% |
| Межгенные промежутки | 12,943% | 17,722% |
| Длина репликона (п.н.) | 5 011 | 62 881 |
Распределение длин возможных продуктов трансляции CDS бактерии Octadecabacter antarcticus 307
Были проанализированы длины 5025 возможных продуктов трансляции CDS. Полученная диаграмма (Рис. 3) отражает распределение длин белковых продуктов.
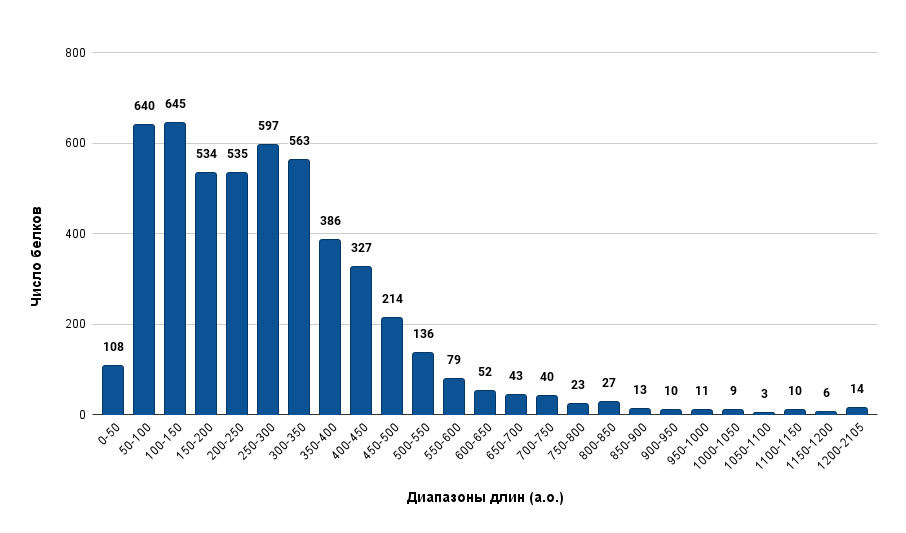
Данное распределение имеет 2 максимума — в промежутках 100–150 и 250–300 аминокислотных остатков, при этом медианное значение равно 253 а.о. Большая часть белковых продуктов имеет длину в промежутке 50–500 а.о. — 4441 продукт из 5025 (88,4%).
Распределение CDS бактерии Octadecabacter antarcticus 307 по GC-составу
Была построена столбчатая диаграмма, отражающая распределение CDS бактерии Octadecabacter antarcticus 307 по GC-составу (Рис. 4).
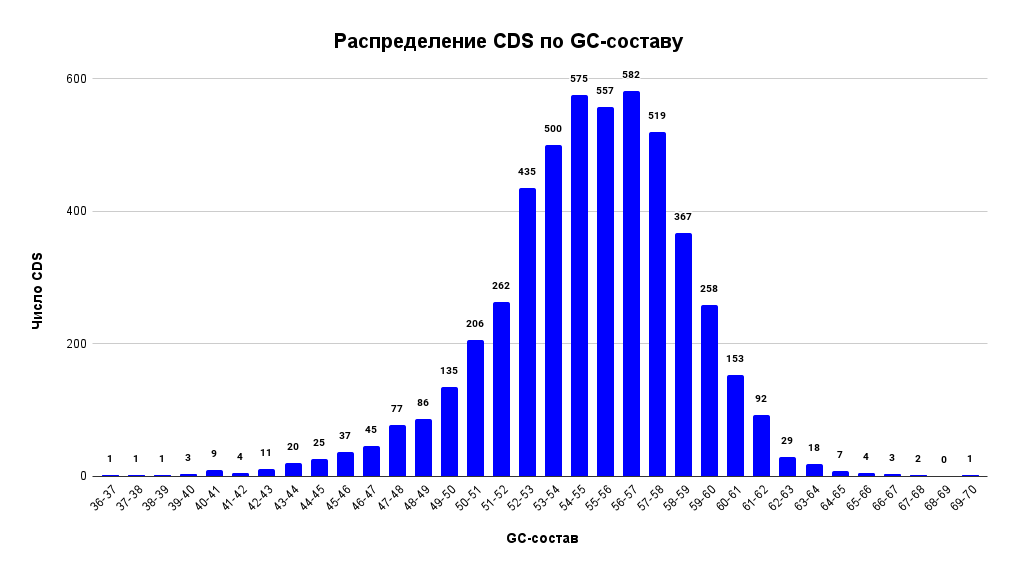
Полученное распределение имеет максимум в районе 54-57% и медианное значение 55,1%, что несколько выше среднего GC-состава по геному (54,6%). При этом GC-состав более 50% имеют 4570 CDS из 5025 (90,9%).
Определение и анализ частот встречаемости старт- и стоп-кодонов в CDS бактерии Octadecabacter antarcticus 307
Были подсчитаны частоты встречаемости различных старт-кодонов в CDS бактерии. Подсчёт был проведен отдельно для всех CDS, для функциональных CDS и для CDS псевдогенов.
Результаты определения частот старт-кодонов (Табл. 4) свидетельствуют о значительно большем разнообразии старт-кодонов в CDS псевдогенов, в сравнении с CDS функциональных генов.
| Старт-кодоны | Функциональные CDS | CDS псевдогенов | Все CDS |
|---|---|---|---|
| ATG | 3 850 | 287 | 4 137 |
| GTG | 346 | 10 | 356 |
| TTG | 262 | 12 | 274 |
| ATT | 22 | 6 | 28 |
| ATC | 14 | 10 | 24 |
| CTG | 7 | 12 | 19 |
| ATA | 4 | 7 | 11 |
| GGC | 0 | 20 | 20 |
| CCC | 0 | 12 | 12 |
| AAC | 0 | 11 | 11 |
| GGG | 0 | 8 | 8 |
| AAA, CGC, CTT | 0 | 7 | 7 |
| GAG, GAT, CAA, CAG | 0 | 5 | 5 |
| AGC, GAA, GAC, GTT, TAT, TTT, CAT, CGA | 0 | 4 | 4 |
| AAG, AAT, ACA, ACG, ACC, CGA, GGT, GCT, CGT, CTA | 0 | 3 | 3 |
| GCC, TAC, CTC, CCT | 0 | 2 | 2 |
| AGA, AGG, AGT, ACT, GTC, GCA, TGG, TGT, TGC, TTC, TCG, CAC, CGG, CCG | 0 | 1 | 1 |
Всего в CDS Octadecabacter antarcticus 307 встречаются 54 различных старт-кодона, столько же старт-кодонов можно обнаружить в псевдогенах. В функциональных генах старт-кодонами могут являться только 7 различных нуклеотидных триплетов, отличающихся от каноничного (ATG) не более, чем на один нуклеотид в 1-й или 3-й позиции.
Эти наблюдения согласуются с моделью, предполагающей нейтральную эволюцию псевдогенов [5]. В соответствие с этой моделью на мутации в псевдогенах не действует отрицательный отбор, поэтому даже мутации, затрагивающие старт-кодон могут закрепиться в популяции в результате дрейфа генов. В случае функциональных генов большинство подобных мутаций, вероятно, приводит к потере функции гена и либо элиминируется отбором, либо становится причиной превращения гена в псевдоген.
Аналогично выглядит распределение частот встречаемости стоп-кодонов (Табл. 5). В CDS Octadecabacter antarcticus 307 на месте стоп-кодона могут находится 56 различных триплетов. Однако, в данном случае, в функциональных CDS обнаруживаются только 3 каноничных стоп-кодона (TAG, TGA, TAA). Остальные же 53 встречаются в CDS псевдогенов, что, вероятно, объясняется теми же причинами, что и высокое разнообразие старт-кодонов в этих CDS.
| Старт-кодоны | Функциональные CDS | CDS псевдогенов | Все CDS |
|---|---|---|---|
| TAG | 1 970 | 151 | 2 121 |
| TGA | 1 598 | 119 | 1 717 |
| TAA | 936 | 81 | 1 017 |
| AAA | 0 | 14 | 14 |
| AAC | 0 | 13 | 13 |
| GAT | 0 | 11 | 11 |
| TCA | 0 | 9 | 9 |
| ACG, TGG, CGC | 0 | 6 | 6 |
| ATC, GCC, GCG | 0 | 5 | 5 |
| GGC, CTG, GTC, GCA, TAC | 0 | 4 | 4 |
| GTT, TTG, TTC, ACC, TCT, CAC, GAC, CTT, ATT, ATG | 0 | 3 | 3 |
| TAT, GGA, CGT, GGT, CGG, GAA, CAT, GCT, CCC, GGG, CCG | 0 | 2 | 2 |
| ACA, TCG, AAT, AAG, CTC, CCT, ACT, ATA, AGT, GTG, GAG, CCA, AGA, CAG, TCC, TGC, CAA | 0 | 1 | 1 |
Анализ межгенных промежутков и пересечений генов для хромосомы бактерии Octadecabacter antarcticus 307
Были проанализированы промежутки между CDS отдельно на плюс- и на минус-цепи хромосомы бактерии Octadecabacter antarcticus 307 (Рис. 5).
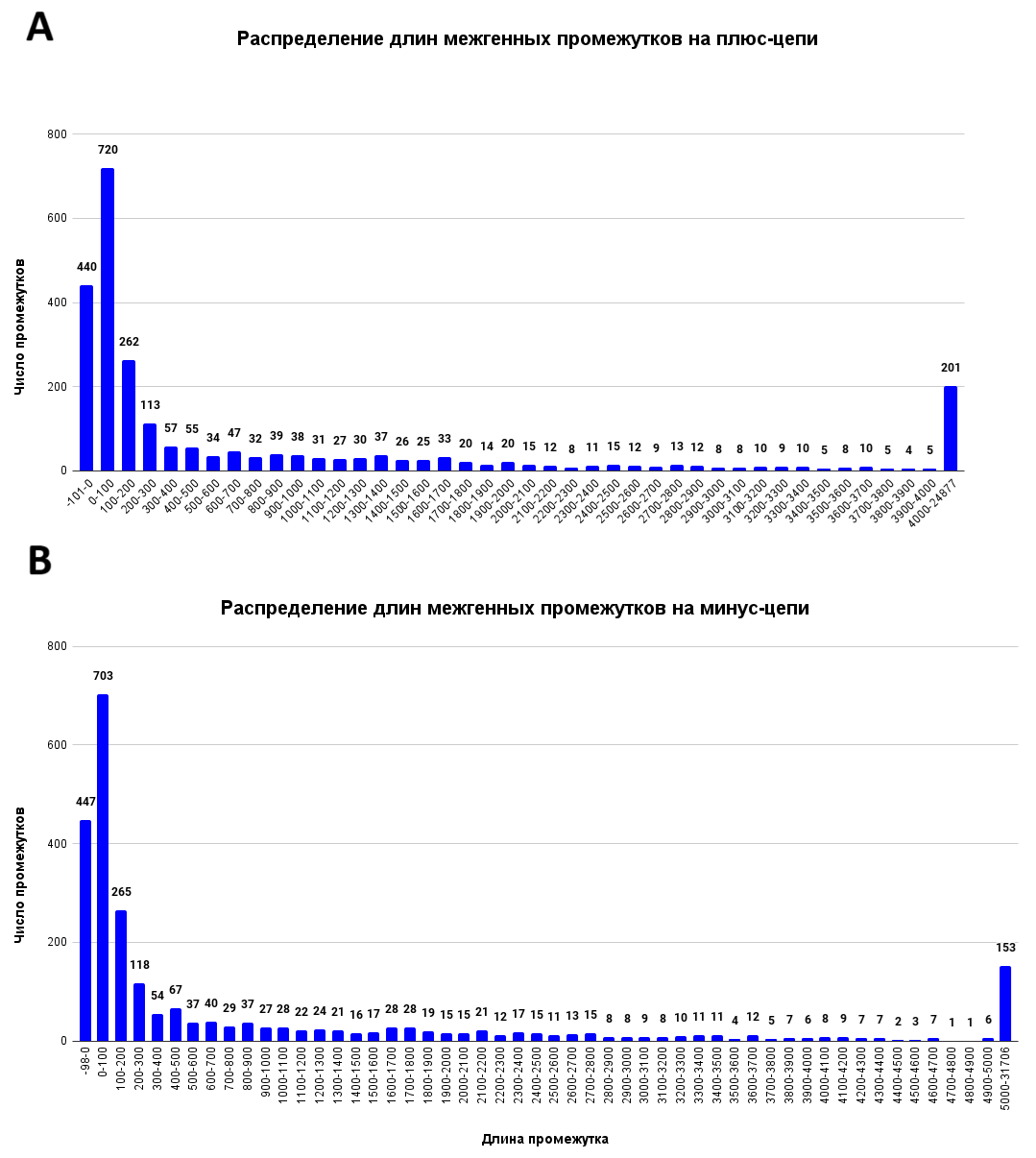
Из полученных диаграмм видно, что наиболее распространены межгенные промежутки длиной от 0 до 100 п.н. Также имеется большое количество промежутков длиной менее 0 п.н., это означает, что в геноме бактерии распространены пересечения CDS.
Для пересечений CDS, расположенных на одной цепи, также были построены столбчатые диаграммы, отражающие распределения их длин (Рис. 6).
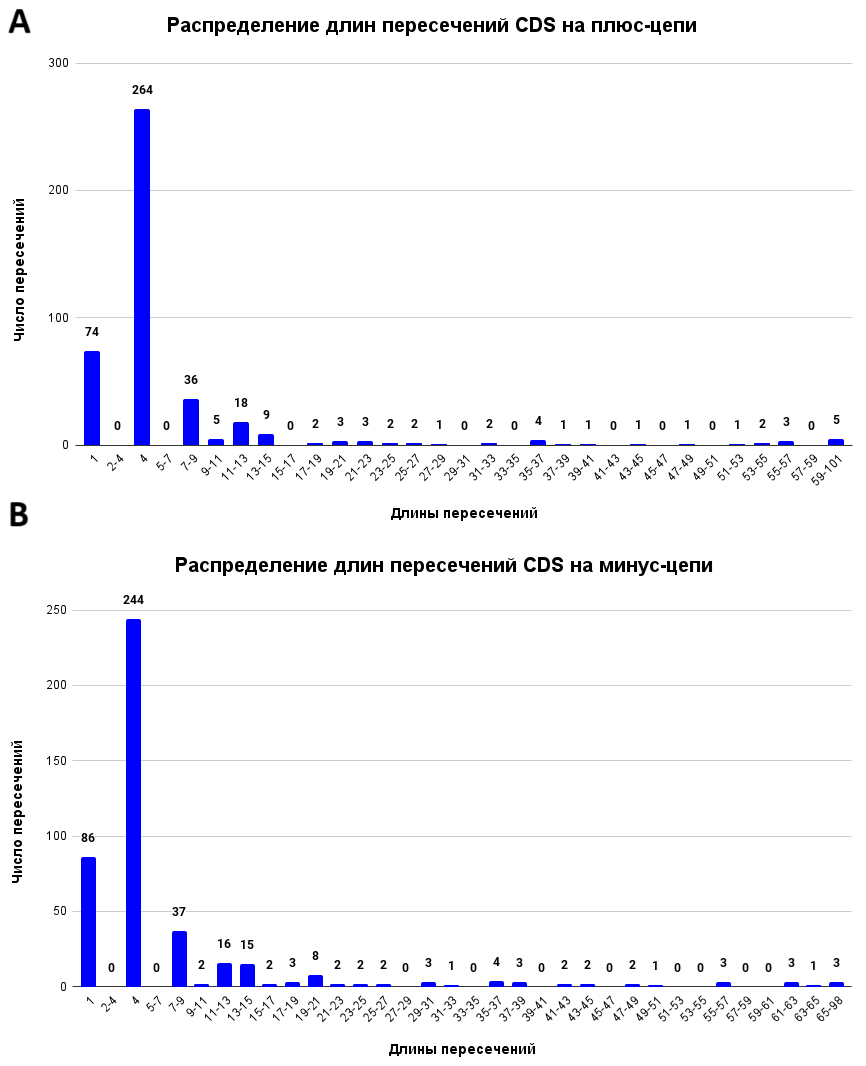
Как видно из диаграмм, большая часть пересечений имеет длины 1 или 4 п.н., причём пересечения длины 2 и 3 п.н. отсутствуют, что согласуется с предположением (см. “Материалы и методы”).
Для подтверждения предположения о пересечениях CDS малой длины, пересечения с длинами 1 и 4 п.н. были подсчитаны для плюс- и минус-цепей хромосомы бактерии Octadecabacter antarcticus 307 (Табл. 6).
| Пересечения длиной 1 п.н. | ||
|---|---|---|
| Пересечение | Количество | Цепь |
| TGATG | 46 | + |
| TAATG | 27 | + |
| TGATC | 1 | + |
| CATCA | 47 | - |
| CATTA | 37 | - |
| CACTA | 2 | - |
| Пересечения длиной 4 п.н. | ||
|---|---|---|
| Пересечение | Количество | Цепь |
| ATGA | 224 | + |
| GTGA | 31 | + |
| TTGA | 7 | + |
| ATAA | 2 | + |
| TCAT | 195 | - |
| TCAC | 31 | - |
| TCAA | 17 | - |
| TCAG | 1 | - |
Последовательности этих пересечений являются типичными для пересечений CDS, расположенных на одной цепи (пересечения “голова к хвосту” — пересекаются старт-кодон одной последовательности и стоп-кодон другой). Однако стоит отметить, что в некоторых пересечениях принимают участие старт-кодоны, отличные от каноничного (ATG): среди пересечений длиной 1 п.н. — ATC на плюс-цепи и GTG на минус-цепи, длиной 4 п.н. — GTG, TTG и ATA на плюс-цепи и GTG, TTG и CTG на минус-цепи.
Пересечения CDS, находящихся на разных цепях также были проанализированы, и для них была построена столбчатая диаграмма распределения длин (Рис. 7).
Из диаграммы можно понять, что наиболее распространены пересечения длинами 2 и 4 п.н., пересечения же длинами 1 и 3 п.н. отсутствуют, что также соответствует предположению (см. “Материалы и методы”).
Пересечения CDS, расположенных на разных цепях, длинами 2 и 4 п.н. были подсчитаны (Табл. 7).
Эти пересечения также являются типичными пересечениями между CDS, расположенными на разных цепях. Причём пересечения длиной 2 п.н. являются пересечениями “голова к голове” (то есть пересекаются старт-кодоны двух последовательностей), а длиной 4 п.н. — пересечениями “хвост к хвосту” (пересекаются стоп-кодоны двух последовательностей). Стоит отметить, что пересечений первого типа значительно меньше, чем пересечений второго типа.
| Пересечения длиной 2 п.н. | |
|---|---|
| Пересечение | Количество |
| CATG | 5 |
| Пересечения длиной 4 п.н. | |
|---|---|
| Пересечение | Количество |
| CTAG | 224 |
| TTAG | 31 |
| CTAA | 7 |
| TTAA | 2 |
Предсказание оперонов в репликонах бактерии Octadecabacter antarcticus 307
На основании данных о координатах начала и конца CDS, имеющих белковый продукт (подробнее см. “Материалы и методы”), была создана таблица из 113 предполагаемых оперонов в геноме бактерии Octadecabacter antarcticus 307 (см. S19). Таблица была проанализирована, и группы CDS, кодирующих белки, связанные физиологически или биохимически, были выбраны в качестве претендентов на статус оперонов (Табл. 8).
| Оперон | Начало | Конец | Функция или процесс |
|---|---|---|---|
| 1 | 246 244 | 251 575 | Метаболизм азота |
| 2 | 381 654 | 386 189 | Синтез фенилаланил-тРНК |
| 3 | 1 402 727 | 1 405 795 | Метаболизм ионов железа, цинка и марганца |
| 4 | 2 278 103 | 2 280 416 | Биосинтез тиаминпирофосфата |
| 5 | 2 718 449 | 2 722 805 | Формирование липополисахаридного слоя |
| 6 | 2 948 806 | 2 952 073 | Сборка и функционирование жгутика |
| 7 | 3 031 635 | 3 032 973 | Формирование газовых везикул |
| 8 | 3 773 073 | 3 775 515 | Транспорт соединений молибдена |
| 9 | 4 093 217 | 4 094 384 | Антипорт одновалентных катионов и протонов |
| 10 | 4 118 132 | 4 123 327 | Катаболизм гистидина |
Стоит отметить, что значительная часть претендентов представляет собой группы CDS, кодирующих субъединицы ABC-транспортёров, эти потенциальные опероны дальше рассмотрены не будут.
Предполагаемые опероны в геноме бактерии Octadecabacter antarcticus 307:
Оперон, состоящий из CDS, кодирующих субъединицы нитритредуктазы B и D и нитратредуктазу. Данный оперон играет роль в метаболизме азота.
Оперон, содержащий CDS, кодирующие субъединицы фенилаланил-тРНК-лигазы альфа и бета, белок, содержащий РНК-связывающий домен ASCH, и один гипотетический белок. Вероятно, отвечает за синтез фенилаланил-тРНК.
Оперон, состоящий из трёх CDS, кодирующих ABC-переносчики ионов металлов; и CDS, кодирующей фактор-регулятор транскрипции семейства FUR (Ferric uptake regulator). Скорее всего отвечает за метаболизм ионов таких металлов, как железо, цинк и марганец [12].
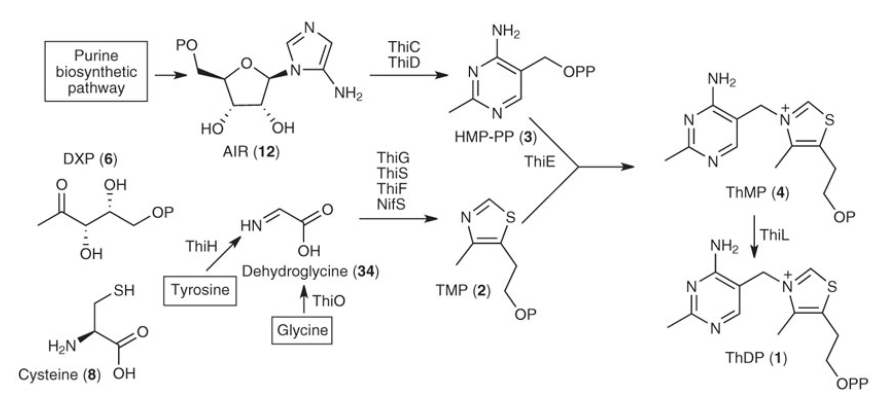
Оперон из CDS, кодирующих белок семейства ThiF, тиамифосфатсинтазу и тиаминсинтазу. Скорее всего играет роль в биосинтезе тиаминпирофосфата (Рис. 8).
Оперон, состоящий из CDS, кодирующих ABC-транспортёры липополисахаридов и белок, отвечающий за сборку липополисахаридов. Данный оперон участвует в синтезе липополисахаридного слоя клеточной стенки бактерии.
Оперон, кодирующий белки семейств FliI и FliH - субъединиц цитоплазматического АТФазного комплекса, являющегося частью системы секреции жгутика III типа [13], а также белка-переключателя направления вращения жгутика FliG. Отвечает за сборку и функционирование бактериального жгутика [13, 14].
Оперон, кодирующий три белка газовых везикул, что коррелирует с наличием газовых везикул у Octadecabacter antarcticus 307 [2].
Оперон, состоящий из CDS, кодирующих переносчики соединений молибдена.
Оперон из CDS, кодирующих субъединицы белков-антипортеров протонов и других одновалентных катионов (например, натрия или калия).
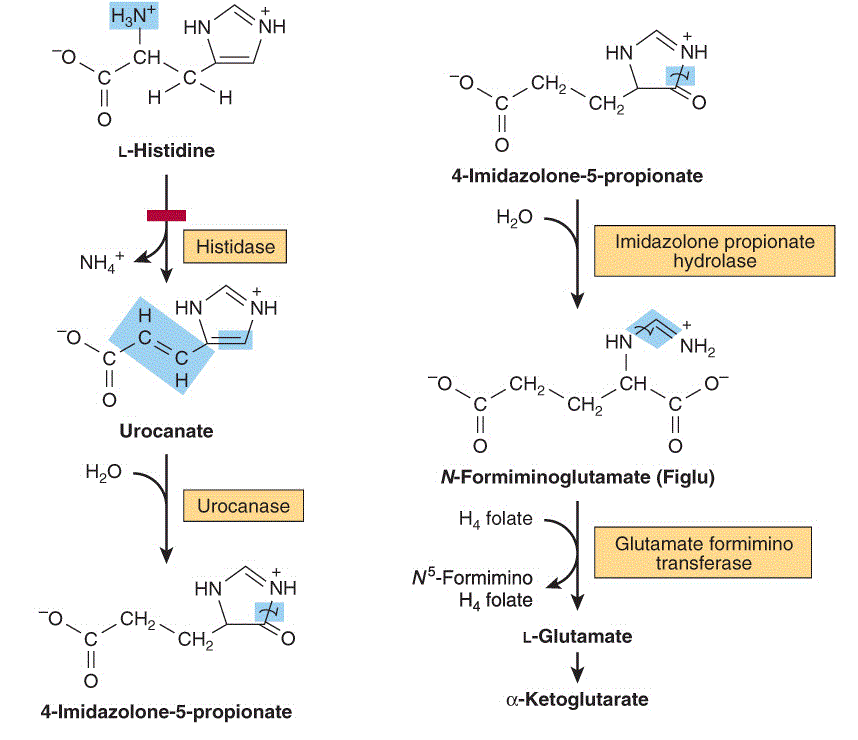
Оперон, содержащий CDS, кодирующие имидазолонпропионазу, гистидин-аммиак-лиазу, N-формилглутаматдеформилазу и уроканатгидратазу. Скорее всего играет роль в катаболизме гистидина (Рис. 9).
Поиск точек начала (origin) и терминации (termus) репликации в репликонах бактерии Octadecabacter antarcticus 307 путём анализа GC-перекоса и TA-перекоса (GC-skew и TA-skew)
Был проанализирован перекос GC и TA вдоль последовательностей хромосомы и плазмиды бактерии Octadecabacter antarcticus 307.
Из графиков кумулятивного перекоса GC (Рис. 10) можно предположить местоположение точек начала репликации в хромосоме и плазмиде бактерии: около 4,6 млн п.н. для хромосомы (для большей наглядности графика в качестве начальной точки была выбрана позиция 1 млн п.н.) и около 49 тыс. п.н. для плазмиды; а также точек терминации репликации: около 2,4 млн п.н. для хромосомы и около 30 тыс. п.н. для плазмиды.
Можно заметить, что расстояние между точками начала и терминации репликации составляет примерно 19 тыс. п.н., что значительно меньше, чем половина длины плазмиды (длина плазмиды равна 62,9 тыс. п.н.). Данный факт можно объяснить тем, что при репликации этой плазмиды одна репликационная вилка движется значительно быстрее другой (в среднем в \(\frac{62,9 - 19}{19} = 2,31\) раза), что может быть связано, например, со взаимодействием определённых белков, замедляющих продвижение вилки, с ДНК в районе с 30 до 49 тыс. п.н.
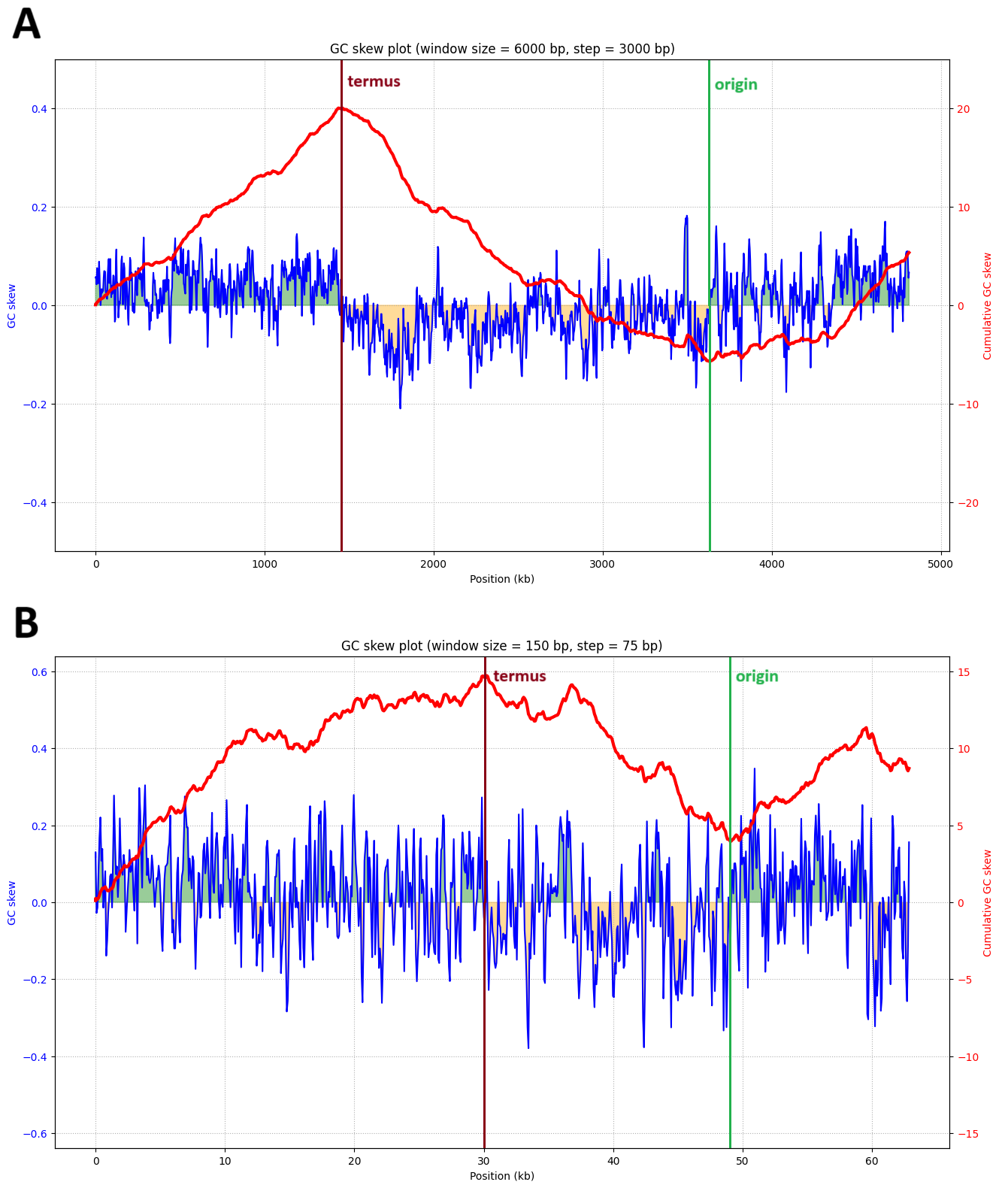
Также были построены графики кумулятивного TA-перекоса для хромосомы и плазмиды Octadecabacter antarcticus 307 (Рис. 11).

Как можно заметить из графиков, изменение перекоса TA для хромосомы бактерии коррелирует с местами нахождения точек начала и терминации репликации так же, как и перекос GC. Для плазмиды же корреляция не столь выражена, и пики кумулятивного перекоса AT не всегда совпадают с таковыми для GC.
Дальнейший анализ асимметрии в распределении нуклеотидов по последовательностям репликонов Octadecabacter antarcticus 307
Был проведён анализ асимметрии в распределениях нуклеотидов по группам (амино(M)/кето(K), пурины(R)/пиримидины(Y), сильные(S)/слабые(W)) в хромосоме и плазмиде бактерии Octadecabacter antarcticus 307.
Расчёт частот встречаемости нуклеотидов по репликонам (Табл. 9) показывает, что и в хромосоме, и в плазмиде наблюдается превышение K (тимин и гуанин) над M (цитозин и аденин), R (аденин и гуанин) над Y (тимин и цитозин) и S (гуанин и цитозин) над W (аденин и тимин).
| Нуклеотиды | Хромосома | Плазмида |
|---|---|---|
| A | 22,68% | 23,55% |
| T | 22,68% | 23,66% |
| G | 27,41% | 26,68% |
| C | 27,23% | 26,11% |
| M | 49,91% | 49,66% |
| K | 50,09% | 50,34% |
| R | 50,09% | 50,23% |
| Y | 49,91% | 49,77% |
| S | 54,64% | 52,79% |
| W | 45,36% | 47,21% |
Графики несоответствия количеств нуклеотидов разных групп (Рис. 12) показывают, что для хромосомы асимметрия по всем рассмотренным группам (M/K и R/Y) связана с точками начала и терминации репликации. Для плазмиды же распределение пуринов и пиримидинов по последовательности напрямую не коррелирует с этими точками, то есть наблюдается ситуация, аналогичная TA-перекосу. Однако, корреляция с точками origin и termus наблюдается в распределении амино и кето-нуклеотидов.

Отдельно были рассмотрены графики несоответствия для групп нуклеотидов S и W (Рис. 13), эти графики имеют иную форму, поскольку напрямую отражают распределение GC-состава по последовательности.
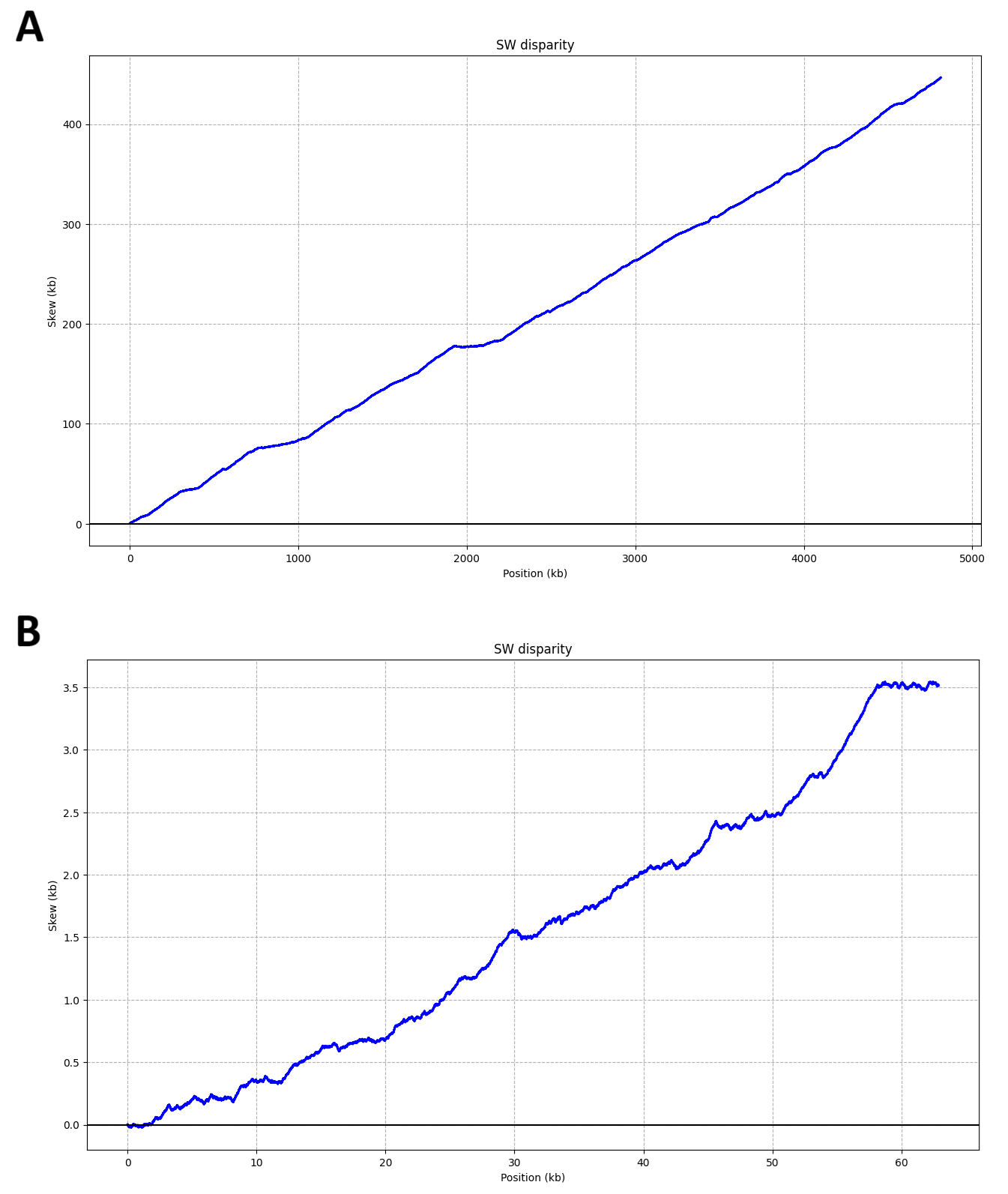
Можно видеть, что распределение GC-состава по хромосоме более равномерное, нежели по плазмиде. Так, в плазмиде имеются множество участков с GC-составом, отличающимся от среднего по последовательности, самые яркие из них: GC-богатый участок с 54 по 58 тыс. п.н. и участок с 58 до 62,9 тыс. п.н. c GC-составом около 50% (среднее для плазмиды - 52,79%).
Также для демонстрации закономерностей в распределении нуклеотидов по последовательностям репликонов были построены Z-кривые (Рис. 14) и графики прохода по ДНК (DNA walk graph) (Рис. 15).
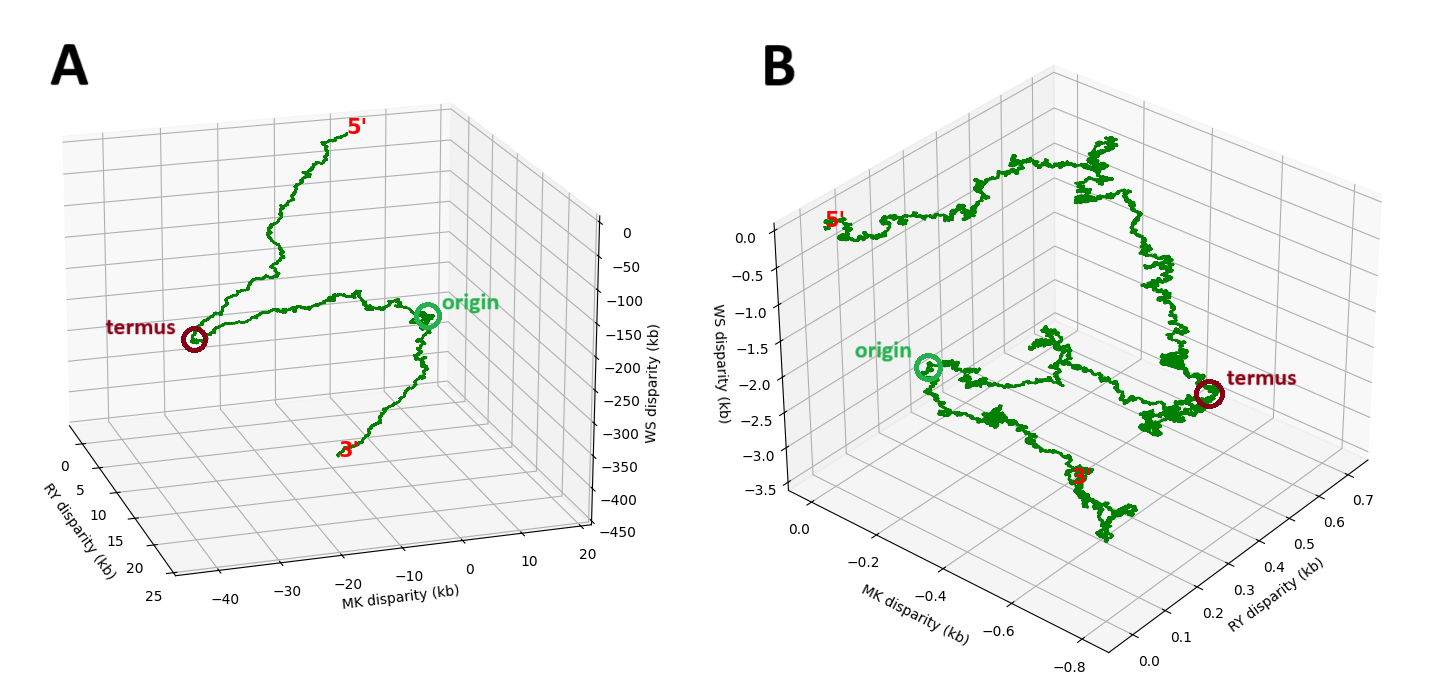
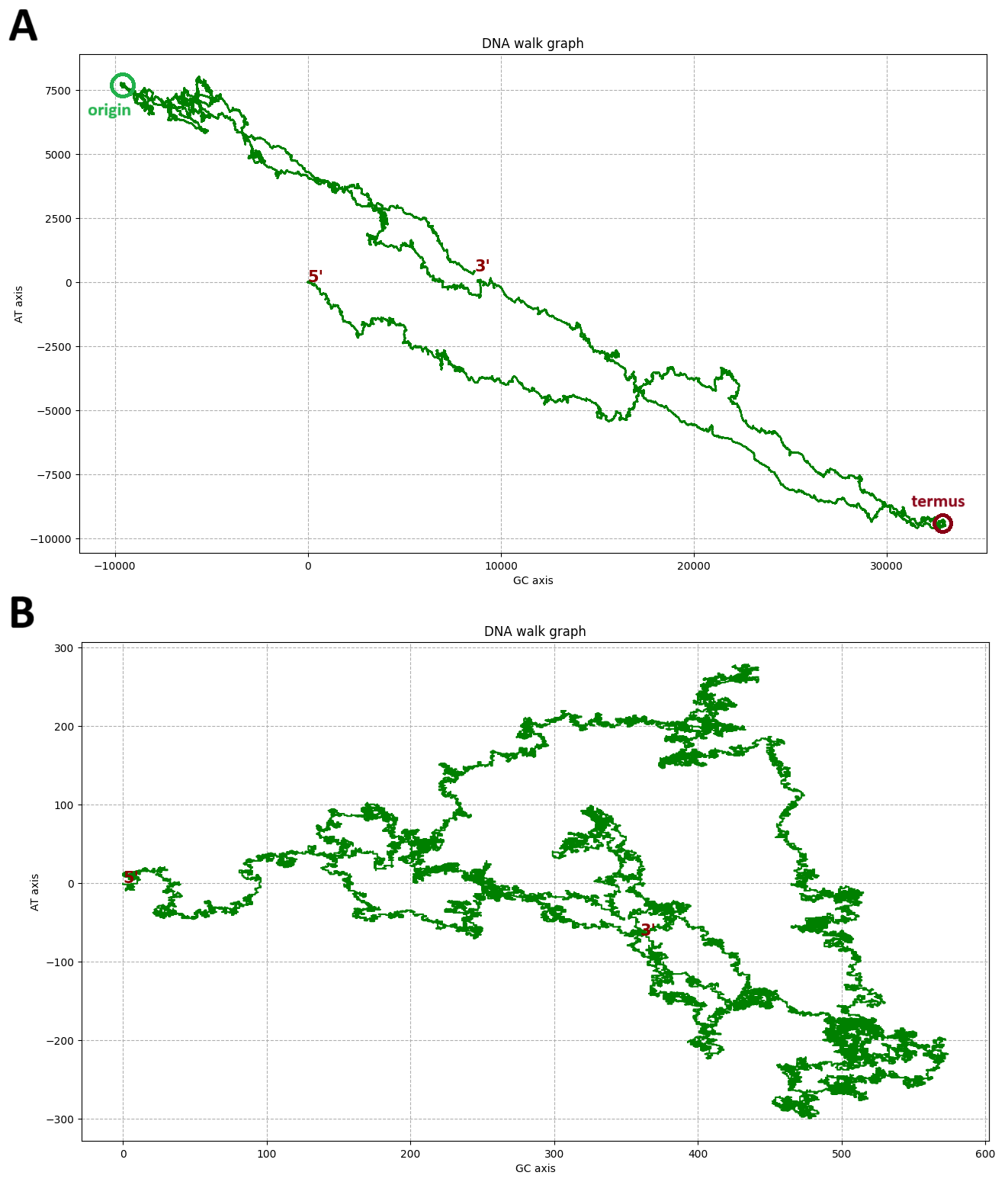
Можно заметить, что Z-кривая и график прохода по последовательности для хромосомы имеют характерную форму, что отражает связь между несоотвествиями нуклеотидного состава по разным группам нуклеотидов и точками начала и терминации репликации. Для плазмиды подобная картина не наблюдается и оба графика имеют множество “петель” и “поворотов”.
Из графиков также видно, что при движении от 5`-конца к 3`-концу последовательности репликона Z-кривая и для хромосомы, и для плазмиды “движется вниз”, что связано с тем, что оба репликона имеют GC-состав более 50%.
Поиск консенсусных последовательностей в прилегающих к 5`-концу CDS областях в геноме Octadecabacter antarcticus 307
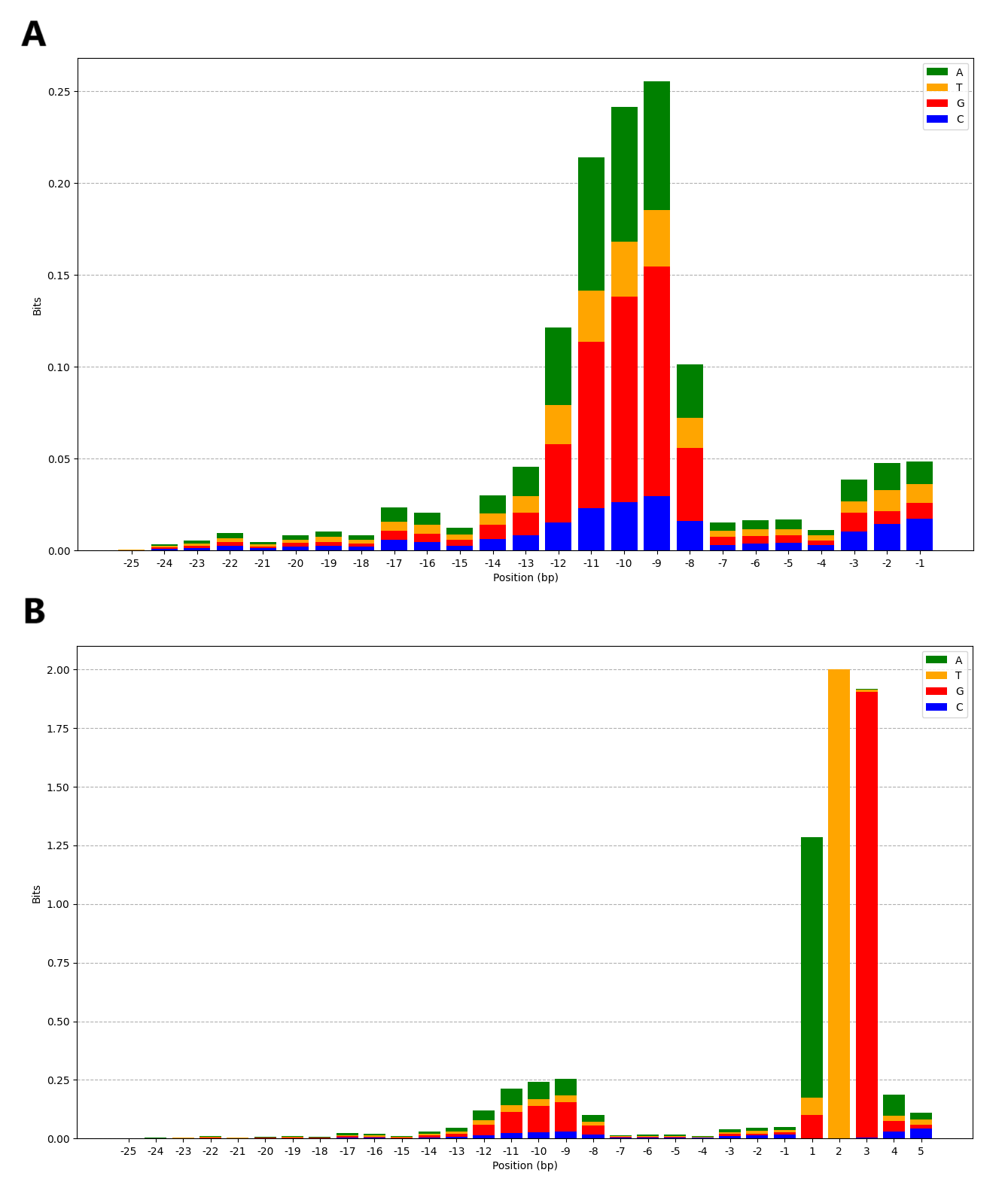
Для регионов, прилегающих к 5`-концу CDS, имеющих белковые продукты, были построены логотипы последовательностей (Рис. 16).
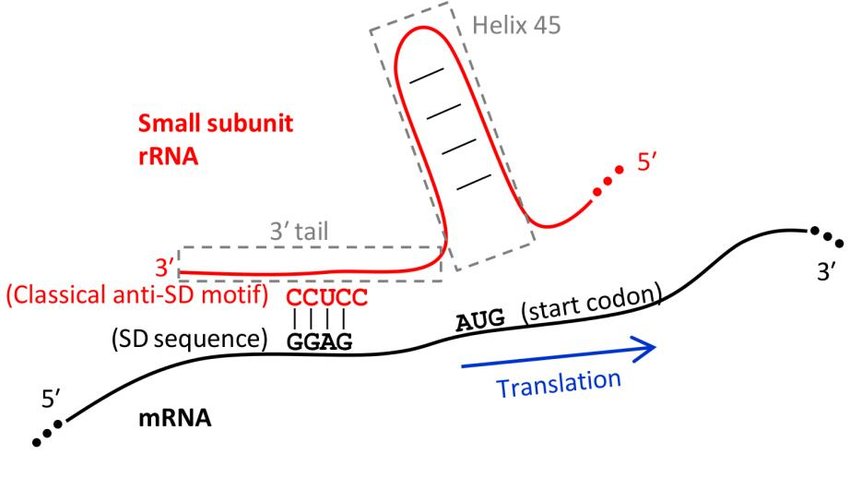
Как видно из логотипа (Рис. 16A), в районе с -12 по -8 п.н. преобладают гуаниловые нуклеотиды, что соответствует последовательности Шайна-Дальгарно, необходимой для связывания малой субъединицы рибосомы с мРНК при инициации трансляции (Рис. 17). Однако, стоит отметить, что консервативность данной последовательности значительно ниже, чем у старт-кодона (Рис. 16B).
ЗАКЛЮЧЕНИЕ
В ходе анализа генома бактерии Octadecabacter antarcticus 307 были выявлены различные особенности его состава, такие как асимметричный нуклеотидный состав плазмиды и низкая консервативность последовательности Шайна-Дальгарно. В целом, такие бактерии, как Octadecabacter antarcticus, могут представлять интерес для исследователей ввиду их быстрой эволюции, что связано с экстремальными условиями обитания.
БЛАГОДАРНОСТИ
Автор выражает благодарность Масленикову Всеволоду и Куликову Антону за помощь и поддержку в процессе подготовки обзора.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
Каталог Google Drive с файлом Scripts for minireview.docx и всеми файлами формата .py
Таблица “Genomic features of Octadecabacter antarcticus 307”
Таблица “CDS from genome of Octadecabacter antarcticus 307”
Таблица “Operones prediction for Octadecabacter antarcticus 307”
Каталог геномной сборки GCF_000155675.2
Каталог геномной сборки GCF_000155675.2, файлы GCF_000155675.2_ASM15567v2_genomic.fna.gz - последовательности репликонов, GCF_000155675.2_ASM15567v2_cds_from_genomic.fna.gz - последовательности CDS, GCF_000155675.2_ASM15567v2_feature_table.txt.gz - таблица геномных особенностей.
Таблица “Genomic features of Octadecabacter antarcticus 307”, лист “per-replicons”.
Таблица “Genomic features of Octadecabacter antarcticus 307”, лист “percentage_of_length”.
Таблица “CDS from genome of Octadecabacter antarcticus 307”, листы “prot_lengths” и “prot_len_hist”.
Таблица “CDS from genome of Octadecabacter antarcticus 307”, листы “cds_gc” и “gc_hist”.
Файл Scripts for minireview.docx, пункт 1.
Файл Stop_codons.py; файл Scripts for minireview.docx, пункт 2.
Таблица “Genomic features of Octadecabacter antarcticus 307”, листы “inter_cds_intervals”, “inter_cds_intervals-hist”, “intersecting_cds”, “intersecting_cds-hist”, “intervals_dif_strands” и “intervals_dif_strands_hist”.
Файлы Overlaps.py и Overlaps_dif_strands.py; файл Scripts for minireview.docx, пункт 3.
Файл Operones.py; файл Scripts for minireview.docx, пункт 4.
Файл GC_skew.py; файл Scripts for minireview.docx, пункт 5.
Файл CGC_CAT_skew.py; файл Scripts for minireview.docx, пункт 6.
Файл Nucleotide_count.py; файл Scripts for minireview.docx, пункт 7.
Файлы All_disparities.py и SW_disparity.py; файл Scripts for minireview.docx, пункт 8.
Файл Z-curve.py; файл Scripts for minireview.docx, пункт 9.
Файл GC_on_AT.py; файл Scripts for minireview.docx, пункт 10.
Файл Seq_logo.py; файл Scripts for minireview.docx, пункт 11.
Таблица “Genomic features of Octadecabacter antarcticus 307”, лист “tRNAs”.
Таблица “Operones prediction for Octadecabacter antarcticus 307”.
ЛИТЕРАТУРА
BacDive: Octadecabacter antarcticus 307 is a Gram-negative bacterium that was isolated from polar sea ice, 25 - 50 cm from the bottom of the ice. BacDive ID: 132057 https://bacdive.dsmz.de/strain/132057
Gosink, J.J., Herwig, R.P., Staley, J.T. Octadecabacter arcticus gen. nov., sp. nov., and O. antarcticus, sp. nov., Nonpigmented, Psychrophilic Gas Vacuolate Bacteria from Polar Sea Ice and Water. Systematic and Applied Microbiology, Volume 20, Issue 3, August 1997, Pages 356-365.
NCBI Taxonomy: Octadecabacter antarcticus 307. NCBI:txid391626 https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=391626
Agris, Paul & Eruysal, Emily & Narendran, Amithi & Väre, Ville & Vangaveti, Sweta & Ranganathan, Srivathsan. (2017). Celebrating Wobble Decoding: Half a century and still much is new. RNA Biology. 15. 00-00. 10.1080/15476286.2017.1356562.
Douglas, G.M., Shapiro, B.J. Pseudogenes act as a neutral reference for detecting selection in prokaryotic pangenomes. Nat Ecol Evol 8, 304–314 (2024). https://doi.org/10.1038/s41559-023-02268-6
Wright, B.W., Molloy, M.P. & Jaschke, P.R. Overlapping genes in natural and engineered genomes. Nat Rev Genet 23, 154–168 (2022). https://doi.org/10.1038/s41576-021-00417-w
Hubert, B. SkewDB, a comprehensive database of GC and 10 other skews for over 30,000 chromosomes and plasmids. Sci Data 9, 92 (2022). https://doi.org/10.1038/s41597-022-01179-8
Andrei Grigoriev, Analyzing genomes with cumulative skew diagrams. Nucleic Acids Research, Volume 26, Issue 10, 1 May 1998, Pages 2286–2290. https://doi.org/10.1093/nar/26.10.2286
Zhang R, Zhang CT. A Brief Review, The Z-curve Theory and its Application in Genome Analysis. Curr Genomics, 2014 Apr, 15(2):78-94. doi: 10.2174/1389202915999140328162433. PMID: 24822026; PMCID: PMC4009844.
Arakawa, Kazuharu & Tamaki, Satoshi & Kono, Nobuaki & Kido, Nobuhiro & Ikegami, Keita & Ogawa, Ryu & Tomita, Masaru. (2009). Genome Projector: zoomable genome map with multiple views. BMC bioinformatics. 10. 31. 10.1186/1471-2105-10-31.
Thomas D. Schneider, R.Michael Stephens. Sequence logos: a new way to display consensus sequences. Nucleic Acids Research, Volume 18, Issue 20, 25 October 1990, Pages 6097–6100. https://doi.org/10.1093/nar/18.20.6097
Fillat MF. The FUR (ferric uptake regulator) superfamily: diversity and versatility of key transcriptional regulators. Arch Biochem Biophys. 2014 Mar 15;546:41-52. doi: 10.1016/j.abb.2014.01.029. Epub 2014 Feb 7. PMID: 24513162.
Kinoshita, M., Minamino, T., Uchihashi, T. et al. FliH and FliI help FlhA bring strict order to flagellar protein export in Salmonella. Commun Biol 7, 366 (2024). https://doi.org/10.1038/s42003-024-06081-0
Marykwas DL, Berg HC. A mutational analysis of the interaction between FliG and FliM, two components of the flagellar motor of Escherichia coli. J Bacteriol. 1996 Mar; 178(5):1289-94. doi: 10.1128/jb.178.5.1289-1294.1996. PMID: 8631704; PMCID: PMC177801.
Kong, D., Zhu, Y., Wu, H. et al. AtTHIC, a gene involved in thiamine biosynthesis in Arabidopsis thaliana. Cell Res 18, 566–576 (2008). https://doi.org/10.1038/cr.2008.35
Victor W. Rodwell, Catabolism of the Carbon Skeletons of Amino Acids. Basicmedical key. https://basicmedicalkey.com/catabolism-of-the-carbon-skeletons-of-amino-acids/
Lim, Kyungtaek & Kobayashi, Ichizo & Nakai, Kenta. (2014). Alterations in rRNA-mRNA Interaction during Plastid Evolution. Molecular biology and evolution. 31. 10.1093/molbev/msu120.