Анализ семейства белковых доменов пиоцина S-типа
Для анализа было выбрано семейство доменов пиоцина S-типа (информация о семействе в Табл. 1), бактериального пептидного токсина, вырабатываемого бактериями из рода Pseudomonas. Был осуществлен поиск максимальных достоверных блоков, а также абсолютно консервативных позиций в полученном множественном выравнивании (см. S1 и S2).
| Информация | |
|---|---|
| AC pfam | PF06958 |
| ID pfam | S-type Pyocin |
| #SEED | 36 |
| #All | 119 |
| #SW | 3 |
| #architectures | 50 |
| #3D | 2 |
| Taxonomy | |
| #eukaryota | 0 |
| #archaea | 1 |
| #bacteria | 2200 |
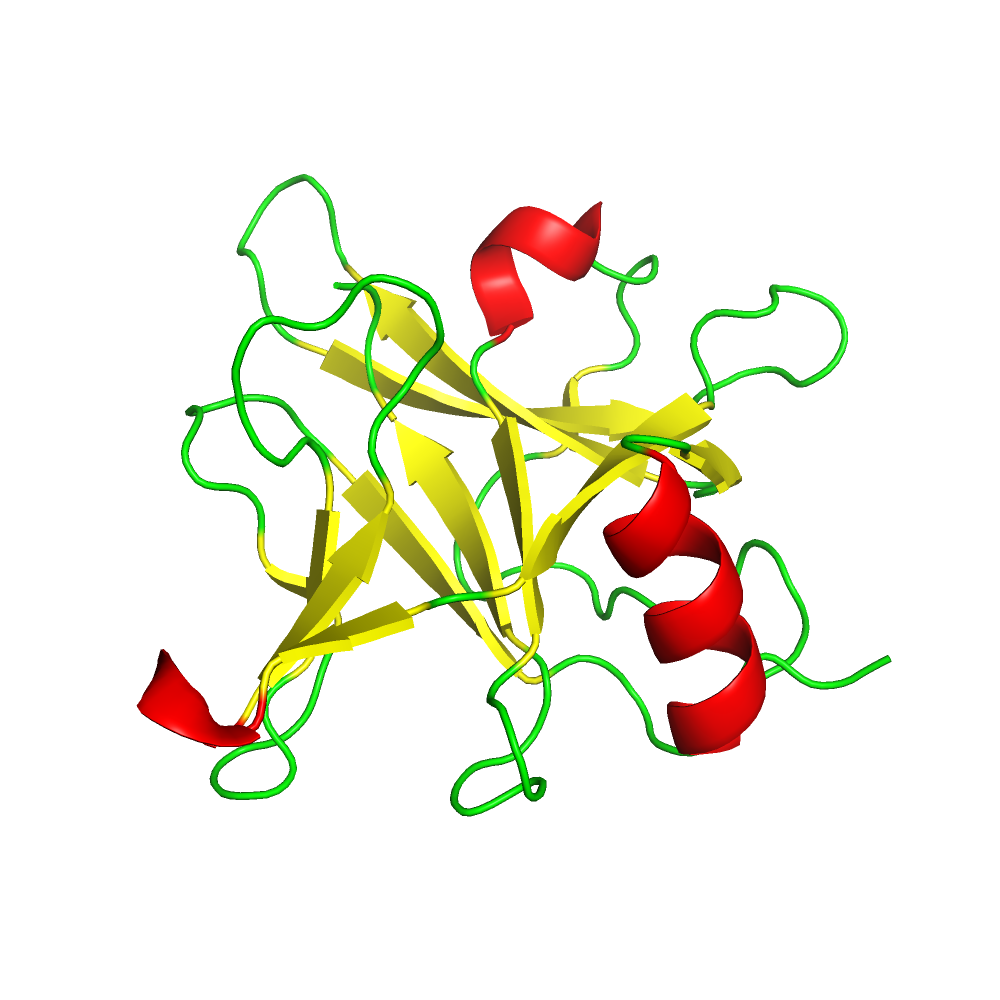
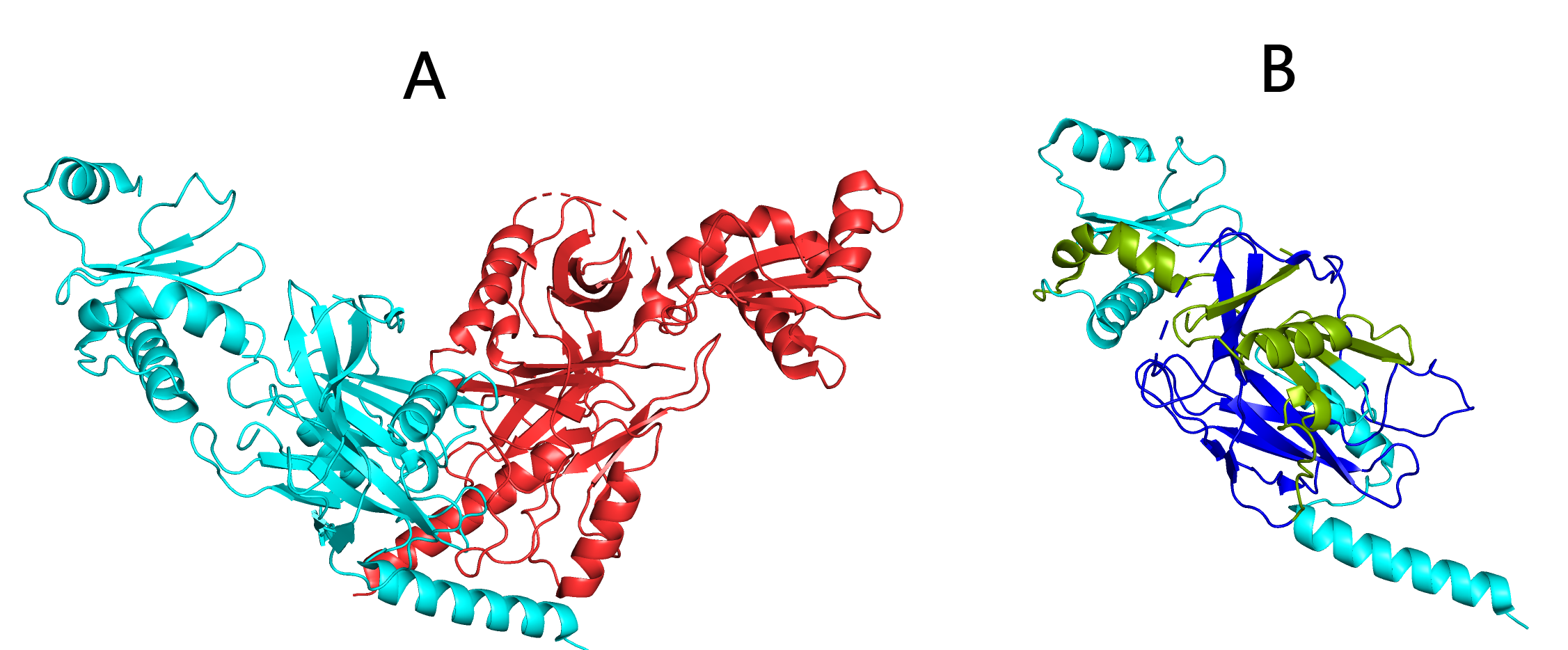
Для 3D-структуры данного домена характерно преобладание бета-слоев (Рис. 1), причем в представленной на сайте InterPro структуре центрального и C-концевого доменов колицина D последовательность, относящаяся к семейству доменов пиоцина S, находится внутри домена транслокации клоацина (Cloacin translocation domain, Рис. 2). В статье, посвященной исследованию структуры данных участков колицина D, говорится, что участок последовательности колицина D, гомологичный участкам последовательностей пиоцинов (этот участок и относится к семейству доменов пиоцина S), участвует во взаимодействии с сигнальной пептидазой LepB, что необходимо для дальнейшего FtsH-зависимого расщепления токсина и проникновения токсичного домена (например, тРНКазного) в клетку [1].
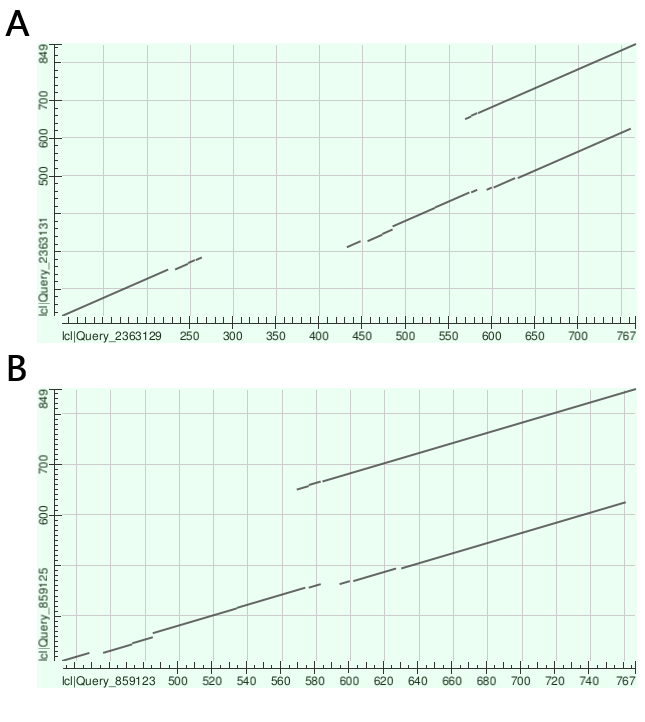
Карта локального сходства последовательностей с разной доменной архитектурой
Для построения карты локального сходства были выбраны два белка из разных доменных архитектур, содержащих домен из семейства пиоцина S (Табл. 2).
| Информация | |
|---|---|
| Доменная архитектура 1 | PF06958 - PF21431 |
| Белок с архитектурой 1 | Q51502 |
| Доменная архитектура 2 | PF06958 - PF21431 - PF21431 |
| Белок с архитектурой 2 | J2MQ52 |
Построенная карта локального сходства (Рис. 3) показала наличие сопоставимых участков, которые соотвествуют доменам из семейств PF06958 и PF21431. Причем, как и ожидалось, части последовательности белка Q51502, соотвествующей сесмейству доменов PF21431, сопоставляются 2 участка последовательности белка J2MQ52, что согласуется с их доменными архитектурами. Однако, помимо участков, сходство которых можно предположить на основании доменных архитектур белков, dot plot при пороговом значении e-value 0.05 (Рис.3A) показал схожесть N-концевых сегментов данных последовательностей. Но при уменьшении порога e-value до 10-11 данный участок на графике пропадает (Рис. 3B), что говорит о более слабом сходстве N-концевых фрагментов по сравнению с участками из одного семейства доменов.
Эволюция доменов в белках с разной доменной архитектурой
Для анализа были выбраны доменные архитектуры PF05488 - PF06958 - PF21431 (33 последовательности, далее - архитектура 1) и PF05488 - PF06958 - PF09000 (21 последовательность, далее - архитектура 2). Все выравнивания были получены пр помощи алгоритма MAFFT, для множественного выравнивания белков с первой доменной архитектурой были удалены избыточные последовательности с идентичностью более 95% (из 33 последовательностей остались 23). Для доменов пиоцина S из данных доменных архитектур было построено множественное выравнивание (см. S3 и Рис. 4).
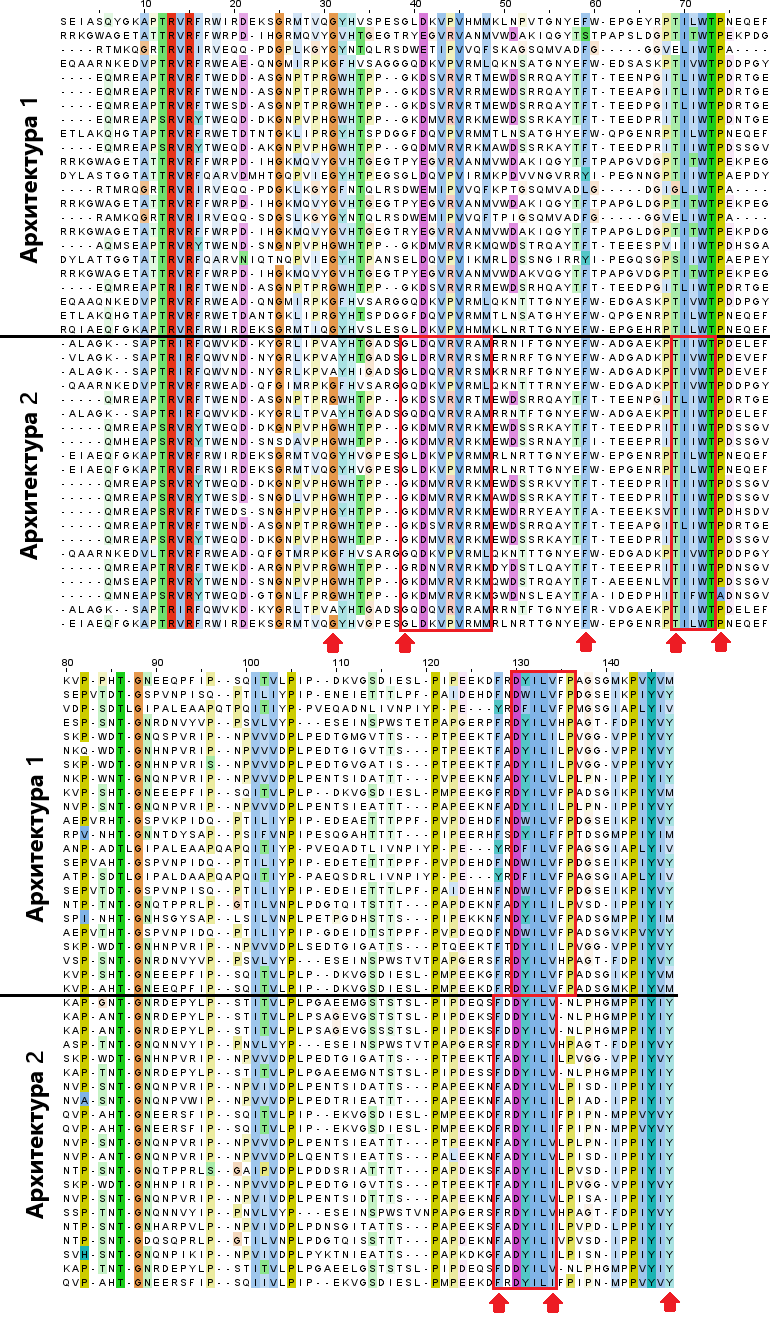
В полученном множественном выравнивании можно отметить различия между доменами из разных доменных архитектур. Так, например, для архитектуры 2 в колонках 39-48 имеется максимальный достоверный блок, в то время как для архитектуры 1 такого достоверного блока не существует, так как позиция 39 выравнивания для данной архитектуры не является абсолютно консервативной. Аналогично для второй доменной архитектуры существуют максимальные достоверные блоки в колонках 69-73 и 128-134, отсутствующие в выравнивании для первой архитектуры, а для первой архитектуры такой блок существует в колонках 130-136. К тому же есть отдельные колонки, абсолютно консервативные только для одной из архитектур (для 1-й: 31 и 74; для 2-й: 59 и 147).
Однако, стоит отметить, что в большей части выравнивания абсолютно консерватиная колонка для одной архитектуры является таковой и для другой, а также аминокислотные полиморфизмы, встречающиеся в некоторых менее консервативных позициях, часто совпадают для двух архитектур (колонки 34, 35, 44, 68, 102 и 124). Эти факты не позволяют отвергнуть вариант включения в рассматриваемые многодоменные белки общего предка данных доменов.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
ЛИТЕРАТУРА И ИСТОЧНИКИ
Chang JW, Sato Y, Ogawa T, Arakawa T, Fukai S, Fushinobu S, Masaki H. Crystal structure of the central and the C-terminal RNase domains of colicin D implicated its translocation pathway through inner membrane of target cell. J Biochem. 2018 Nov 1;164(5):329-339. doi: 10.1093/jb/mvy056. PMID: 29905832.