Анализ структуры белка
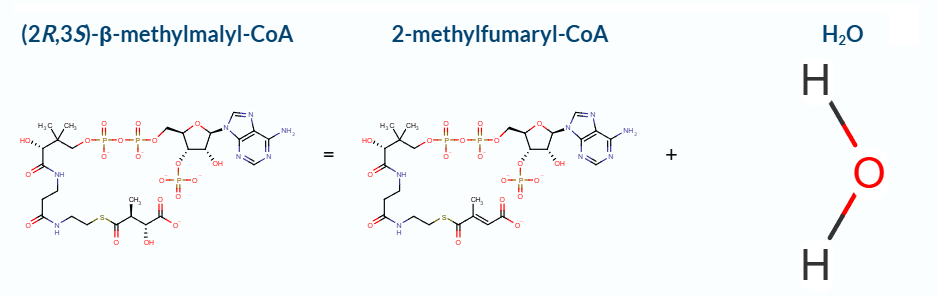
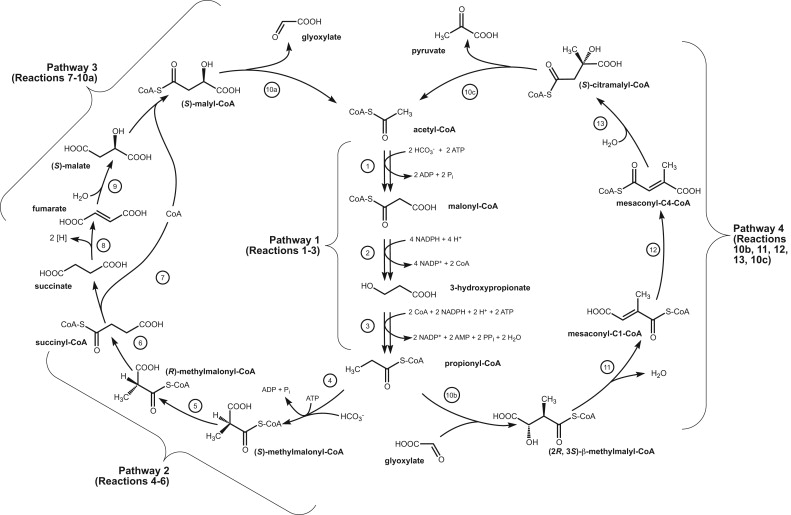
В базе данных PDB идентификатор 4E3E имеет структура белкового комплекса, выделенного из зелёной несерной бактерии Chloroflexus aurantiacus J-10-fl [1]. Данный комплекс является бета-метилмалонил-КоА дегидратазой (Beta-methylmalyl-CoA dehydratase) — ферментом, катализирующим одно из превращений (Рис. 1) в особом пути фиксации CO2, характерном для фотосинтезирующих бактерий филума Chloroflexota — 3-гидроксипропионатном бицикле (пути Фукса-Холо, Рис. 2).
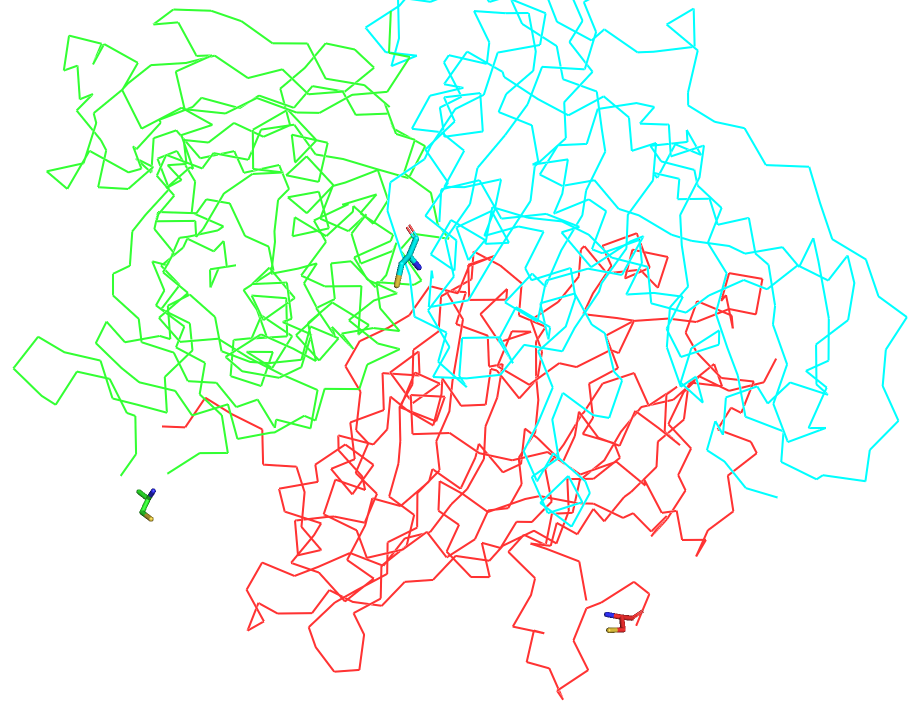
Биологическая единица представляет собой гомотример, состоящий из трёх цепей A (Рис. 3). При этом для данного белка кроме цепи A описана также цепь B, однако они идентичны по аминокислотному составу и структуре: имеют длину 352 а.к. и не имеют мутаций по сравнению с референсной последовательностью из базы данных UniProt (ID A9WC34).
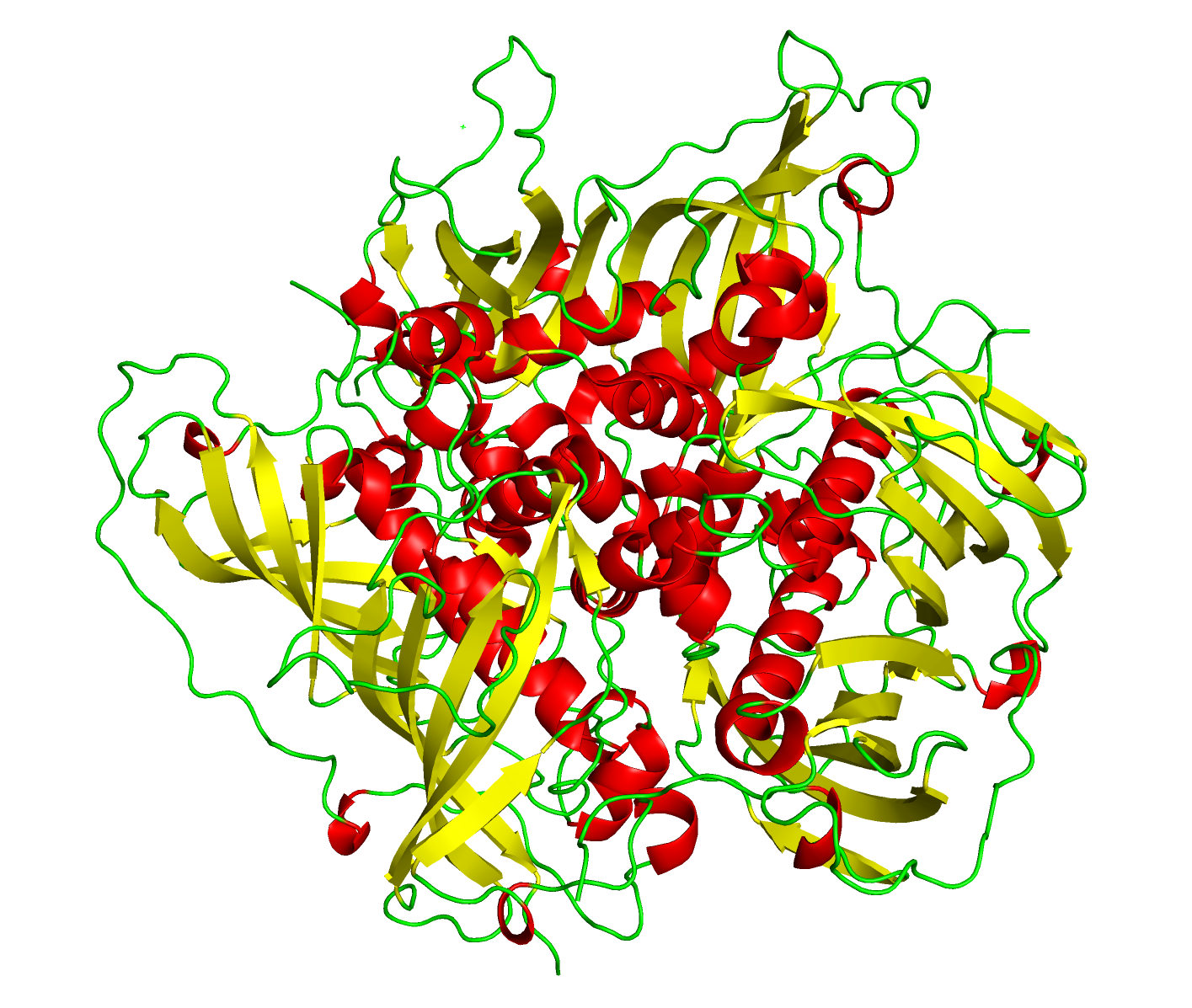
Для вторичной структуры бета-метилмалонил-КоА дегидратазы характерно наличие как альфа-спиралей, так и бета-листов (Рис. 4). При этом альфа-спирали располагаются во внутренней части гомотримера, тогда как бета-листы — во внешней.
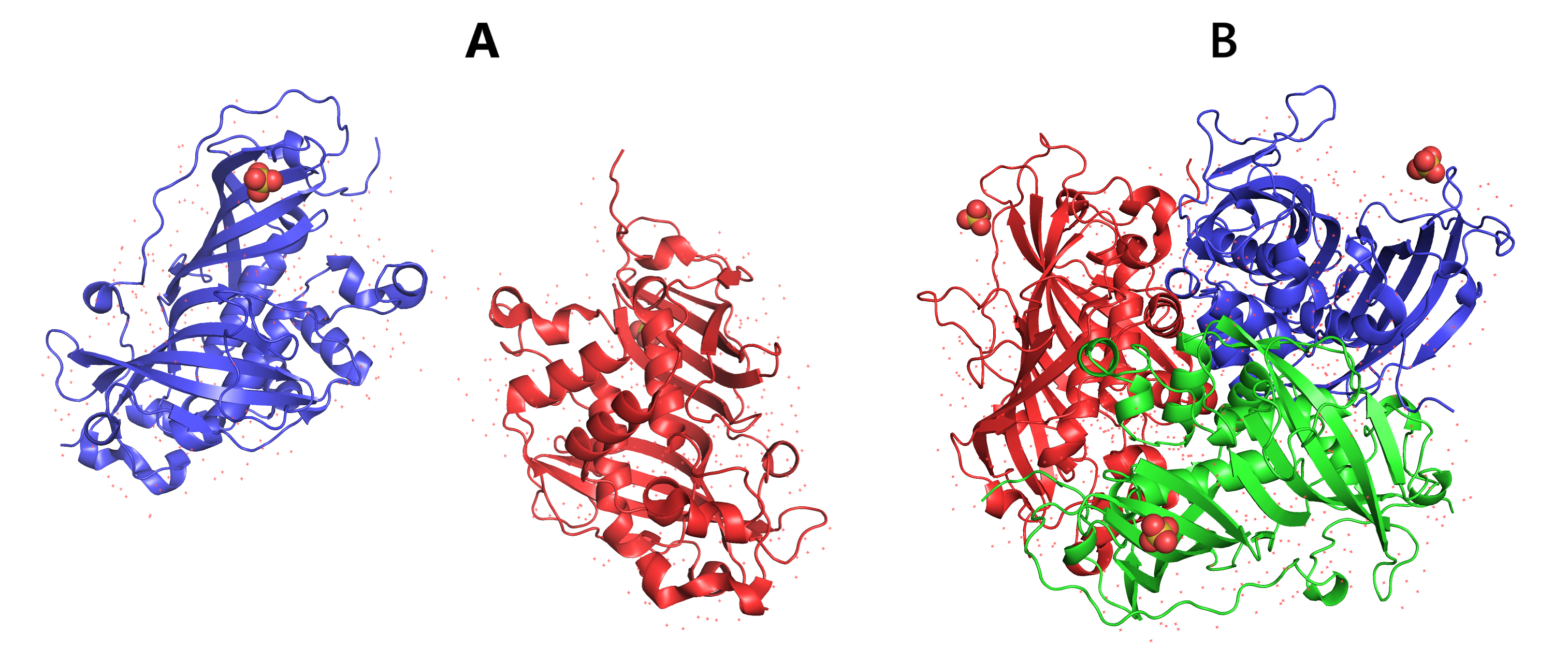
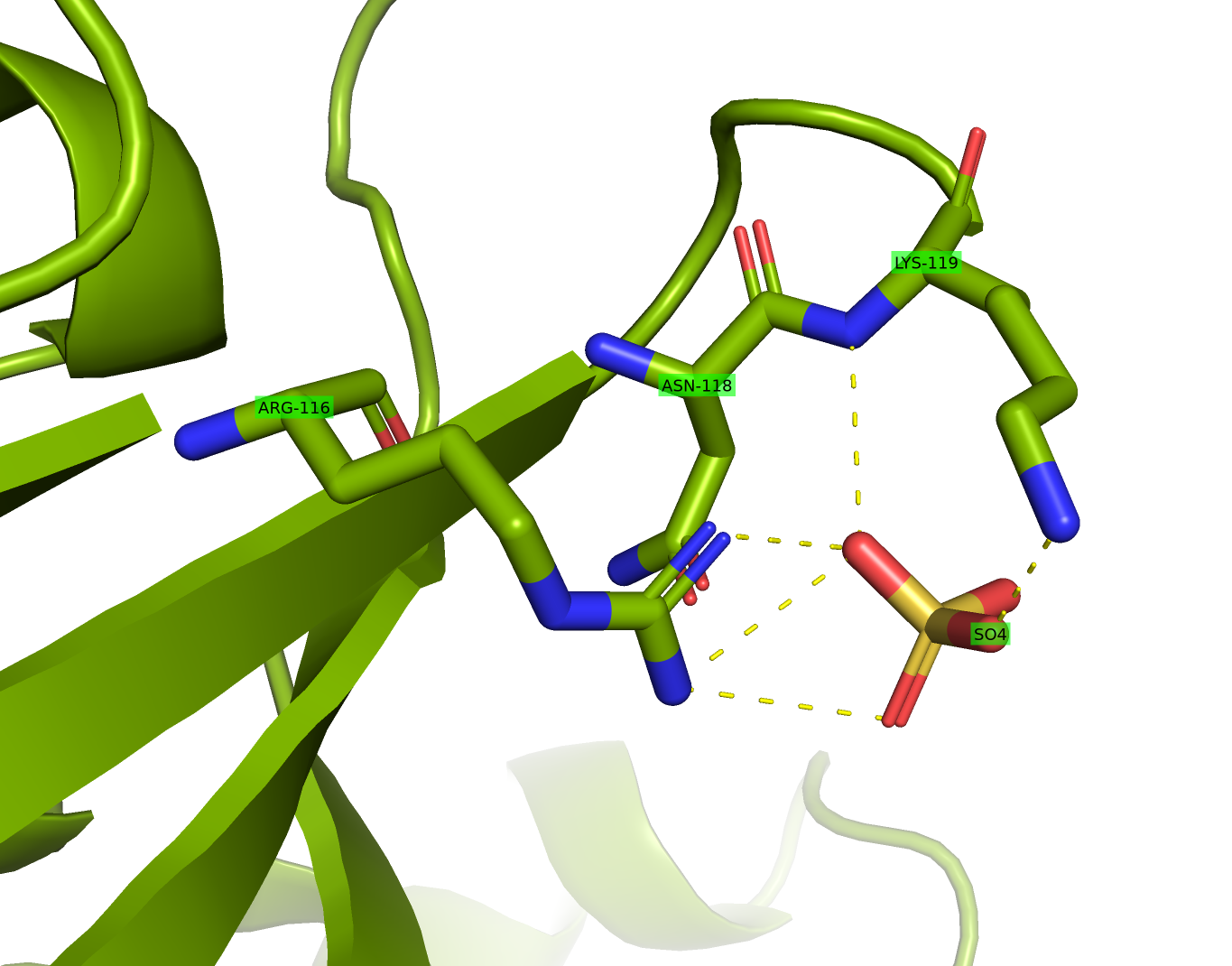
В представленной структуре комплекса все остатки метионина заменены на остатки селенометионина. Скорее всего, в данном случае селенометионин был включен в последовательность в процессе наработки белка с целью увеличения эффективности рентгеновской кристаллографии. Также в записи присутствуют два типа малых молекул: сульфат-анион (SO4, Sulfate ion, см. S1), являющийся компонентом кристаллизационного буфера, а также молекулы воды (HOH, Water, см. S2). Каждая субъединица в тримере связана с сульфат-ионом (Рис. 5), в координации которого принимают участие остатки аргинина-116 и аспарагина-118, образующие водородные связи с анионом, а также лизина-119, связанного с остатком серной кислоты посредством ионной связи. Причем стоит отметить, что в связывании принимает участие не боковая цепь аспарагина-118, а атом водорода, связанный с азотом пептидной связи между аспарагином-118 и лизином-119.
Анализ нековалентных взаимодействий в структуре белка
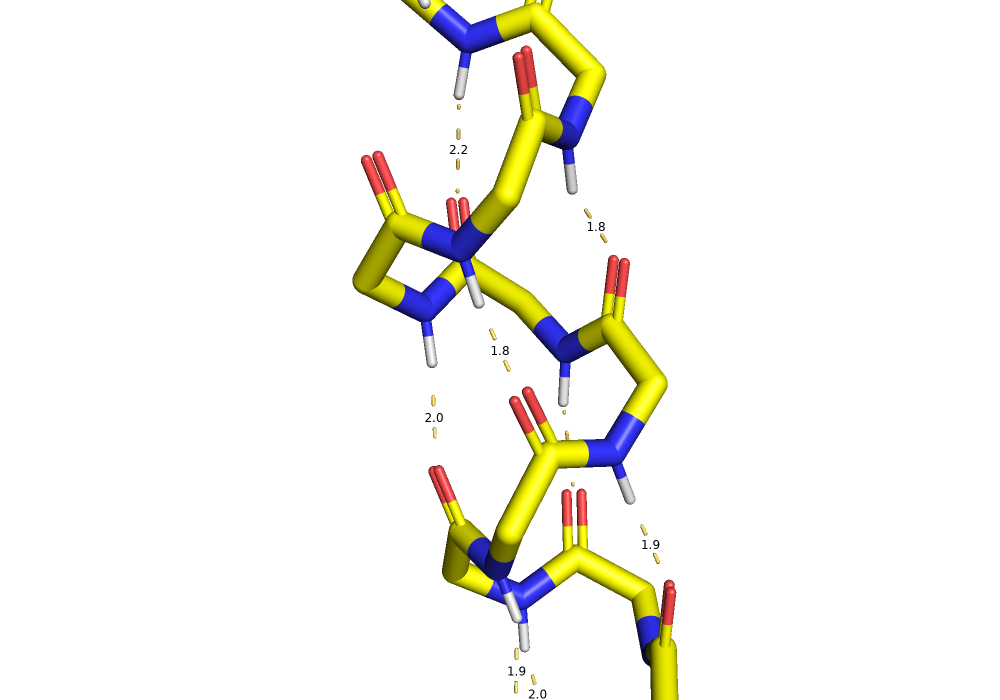
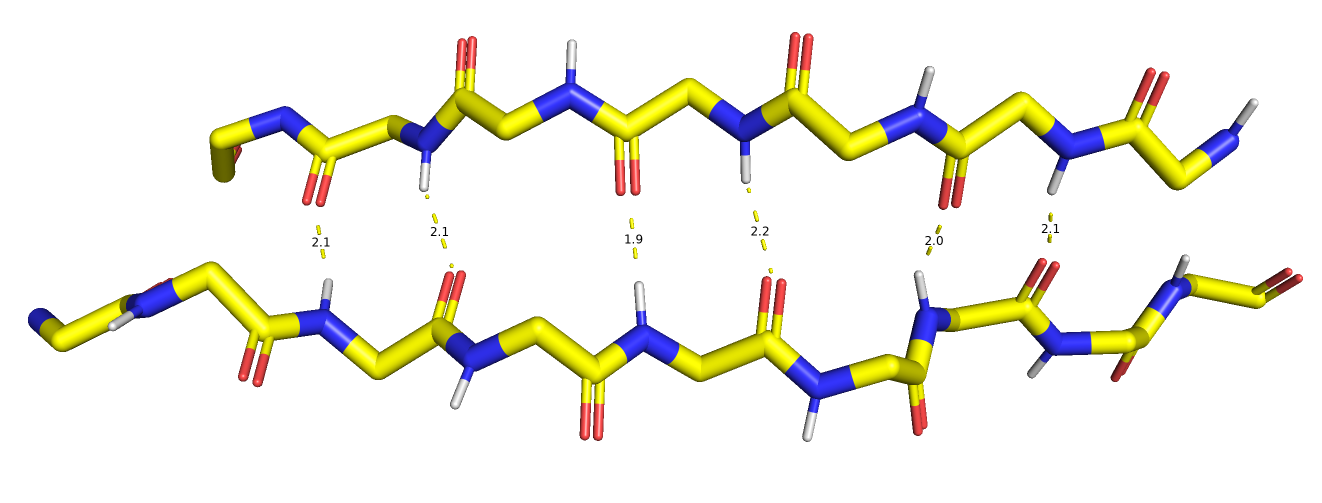
В анализируемом белке представлены альфа-спирали (Рис. 6) и бета-листы (Рис. 7), имеющие типичное строение.
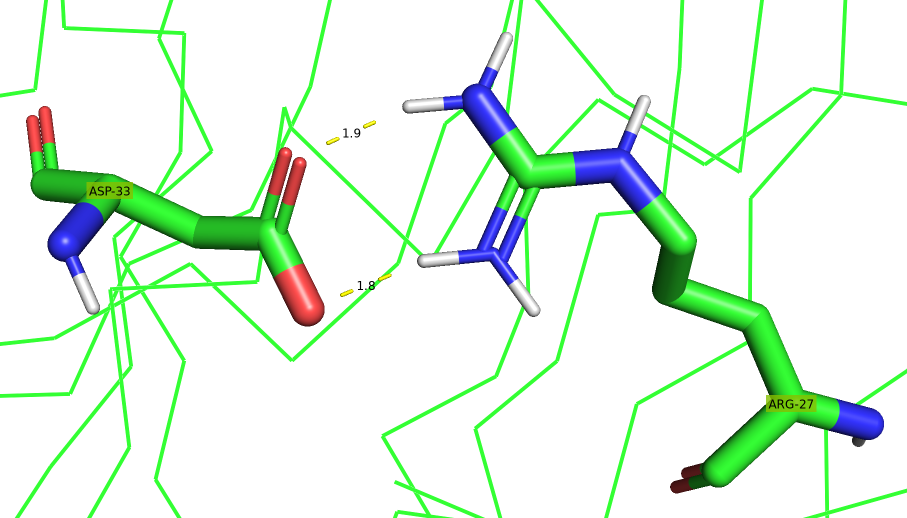
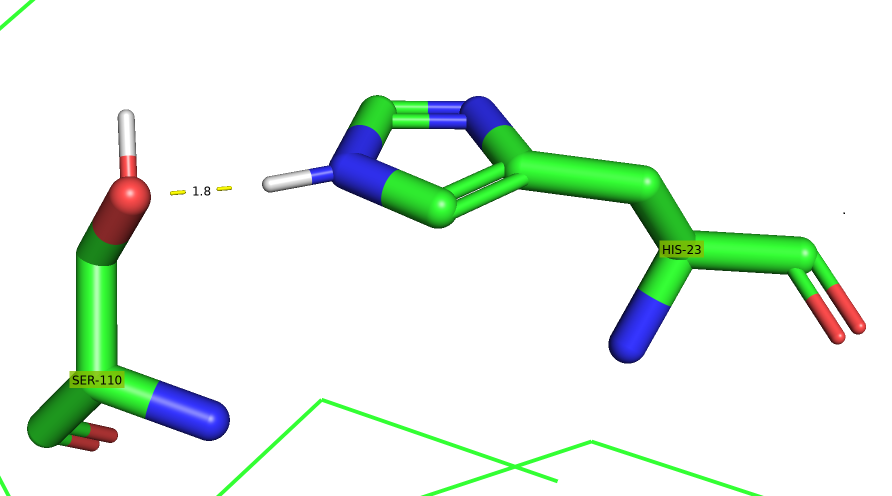
В структуре бета-метилмалонил-КоА дегидратазы также присутствуют солевые мостики (Рис. 8) и водородные связи между боковыми радикалами аминокислотных остатков.
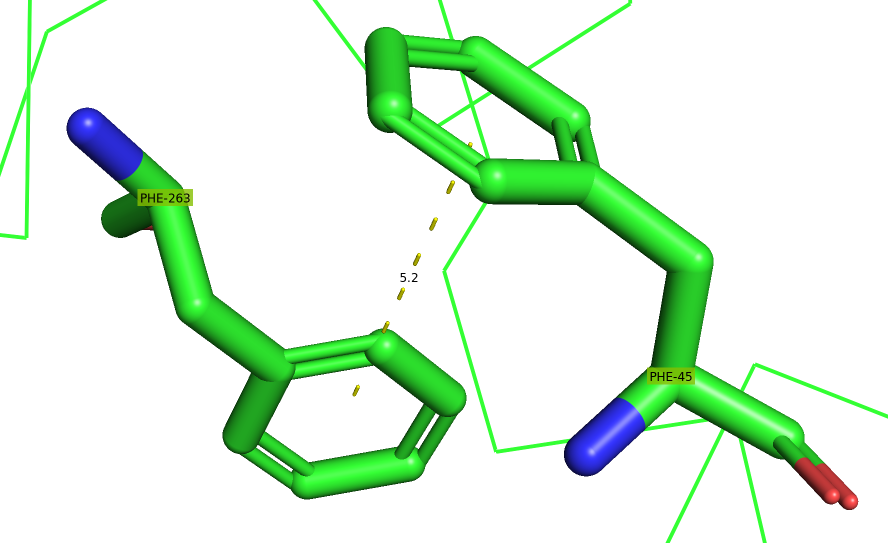
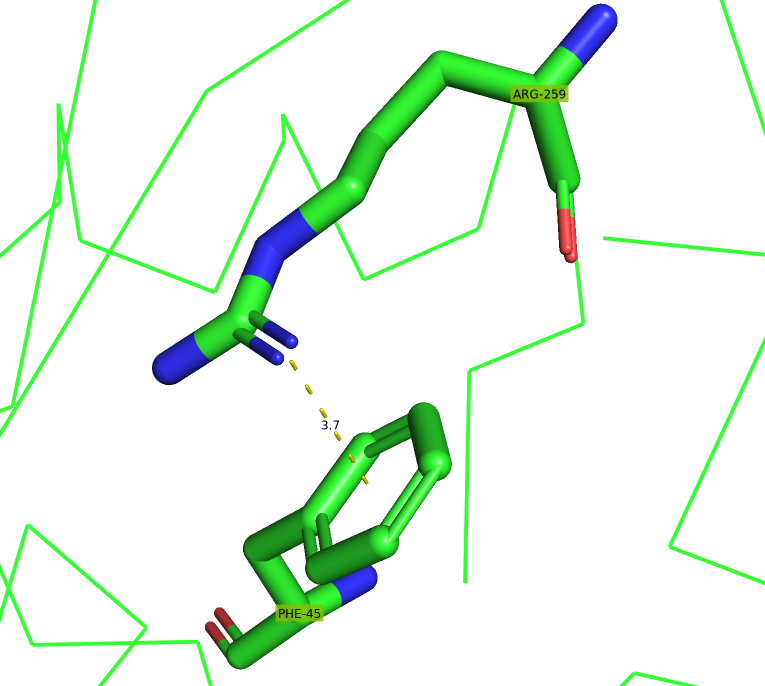
В исследуемой структуре присутствуют стекинг-взаимодействия, включающие π-π (Рис. 10) и π-катионные (Рис. 11) взаимодействия.
В структуре каждой субъединицы присутствует только один остаток цистеина (Рис. 12), вследствие чего образование дисульфидных связей не происходит, что в целом типично для цитозольных белков.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
- Файл, содержащий информацию о сульфат-ионах в структуре белка
- Файл, содержащий информацию о молекулах воды в структуре белка
ЛИТЕРАТУРА И ИСТОЧНИКИ
The Protein Data Bank Malashkevich, V.N., Bhosle, R., Toro, R., Hillerich, B., Gizzi, A., Garforth, S., Kar, A., Chan, M.K., Lafluer, J., Patel, H., Matikainen, B., Chamala, S., Lim, S., Celikgil, A., Villegas, G., Evans, B., Zenchek, W., Love, J., Fiser, A., Khafizov, K., Seidel, R., Bonanno, J.B., Almo, S.C., New York Structural Genomics Research Consortium (NYSGRC) CRYSTAL STRUCTURE OF putative MaoC domain protein dehydratase from Chloroflexus aurantiacus J-10-fl https://doi.org/10.2210/pdb4e3e/pdb
UniProt: A9WC34 MCH_CHLAA https://www.uniprot.org/uniprotkb/A9WC34
Matthew d. Mattozzi, Marika Ziesack, Mathias J. Voges, Pamela A. Silver, Jeffrey C. Way, Expression of the sub-pathways of the Chloroflexus aurantiacus 3-hydroxypropionate carbon fixation bicycle in E. coli: Toward horizontal transfer of autotrophic growth, Metabolic Engineering, Volume 16, 2013, Pages 130-139, ISSN 1096-7176, https://doi.org/10.1016/j.ymben.2013.01.005.