Анализ референсного протеома Octadecabacter ascidiaceicola
В предыдущих работах использовалась геномная сборка бактерии Octadecabacter antarcticus 307, имеющая ID GCF_000155675.2 в базе данных RefSeq. На странице данной геномной сборки в NCBI Datasets Genome указан идентификатор сборки INSDC: GCA_000155675.2. Данный идентификатор был использован в поисковом запросе (genome_assembly:GCA_000155675.2) для поиска протеома бактерии в базе данных UniProt Proteomes. Найденный протеом имеет идентификатор UP000005307 и статус "Other proteome".
Далее был осуществлен поиск референсных протеомов, принадлежащих бактериями из рода Octadecabacter, в базе данных UniProt Proteomes, для этого использовался запрос (taxonomy_id:53945). По данному запросу были найдены 3 протеома: Octadecabacter sp. SW4, Octadecabacter arcticus 238 и Octadecabacter ascidiaceicola. Для дальнейшего анализа был выбран протеом Octadecabacter ascidiaceicola (ID: UP000203464), поскольку для него указан CPD Close to standard (low value), и из данных трех протеомов он характеризуется наибольшей долей генов, определенной алгоритмом BUSCO как "Single" (99,3%).
Для скачивания этого протеома в формате .gz был использован конвейер: curl 'https://rest.uniprot.org/uniprotkb/stream?compressed=true&format=txt&query=(xref:proteomes-UP000203464)' > UP000203464.swiss.gz.
Для оценки количества обладающих ферментативной активностью белков из протеома Octadecabacter ascidiaceicola были использованы следующие запросы в базе данных UniProtKB: (proteome:UP000203464) AND (cc_catalytic_activity:*), (proteome:UP000203464) AND (ec:*), (proteome:UP000203464) AND ((ec:*) OR (cc_catalytic_activity:*)). По первому запросу было найдено 579 записей, тогда как по второму 1204 результата. Такая большая разница может объясняться тем, что для многих белков, потенциально обладающих каталитической активностью, последняя должным образом не охарактеризована. По третьему запросу нашлось 1208 белков, то есть для 4 белков, имеющих в комментариях раздел "CATALYTIC ACTIVITY", не указан номер EC.
Также была осуществлена попытка оценки данного количества методами командной строки Linux, для этого был составлен конвейер: zcat UP000203464.swiss.gz | grep '\-!\-' | uniq | grep -c 'CATALYTIC ACTIVITY'. Предполагается, что вероятность того, что последний пункт комментариев к одной записи и первый пункт комментариев к следующей будут "CATALYTIC ACTIVITY", довольно мала. Действительно, подсчитанное таким образом число потенциальных ферментов составляет 575, что мало отличается от 579, полученных при поиске по запросу (proteome:UP000203464) AND (cc_catalytic_activity:*). Однако стоит отметить, что как было показано ранее, количество ферментов в протеоме данной бактерии скорее всего значительно больше.
Как было сказано ранее, в рассматриваемом протеоме значительная часть белков, имеющая номер EC, не имеет записи "CATALYTIC ACTIVITY" в комментариях. Был проведен анализ номеров EC для таких ферментов. С этой целью был использован скрипт на языке Python (см. S1), создающий файл формата CSV, в который записаны EC для рассматриваемых белков и указано их количество (см. S2).
Полученные результаты были отсортированы (см. S3), из них видно что значительная часть белков в данной группе имеет неполный номер EC. Такие EC были отобраны отдельно (см. S4) и записаны в файл CSV (см. S5), который также был отсортирован (см. S3). В результате выяснилось что 181 EC из полученных 629 неполные.
Аналогичные операции были проделаны с белками для которых в комментариях указана их каталитическая активность (см. S6 - S10). Для них из указанных 1232 номеров EC (их значительно больше, чем белков, поскольку для одного белка зачастую указано несколько номеров) неполными являются только 32.
Наблюдаемую ситуацию можно объснить тем, что все белки в исследуемом протеоме были аннотированы автоматически, и, по-видимому, для многих алгоритм HAMAP не предсказал каталитическую активность. Белки же, имеющие неполный автоматически аннотированный номер EC чаще находятся именно в этой группе.
Анализ референсного протеома
Была проведена симуляция двумерного гель-электрофореза белков из протеома Octadecabacter ascidiaceicola. Двумерный гель-электрофорез - метод разделения белков, включающий собственно электрофорез (разделение по молекулярной массе) и изоэлектрическое фокусирование (разделение по изоэлектрической точке, pI).
При помощи инструмента pepstats из пакета EMBOSS (конвейер: zless UP000203464.swiss.gz | pepstats -filter | grep -e '^PEPSTATS' -e '^Molecular weight' -e '^Isoelectric Point' > stat.out) был получен файл (см. S11), содержащий ID белковых последовательностей из протеома бактерии, а также информацию о молекулярной массе и изоэлектрической точке для каждого из белков. Также был написан скрипт на языке Python (см. S12), позволяющий построить точечный график (dot plot, Рис. 1), отражающий ожидаемое распределение белков при двумерном гель-электрофорезе всех белков из протеома Octadecabacter ascidiaceicola.
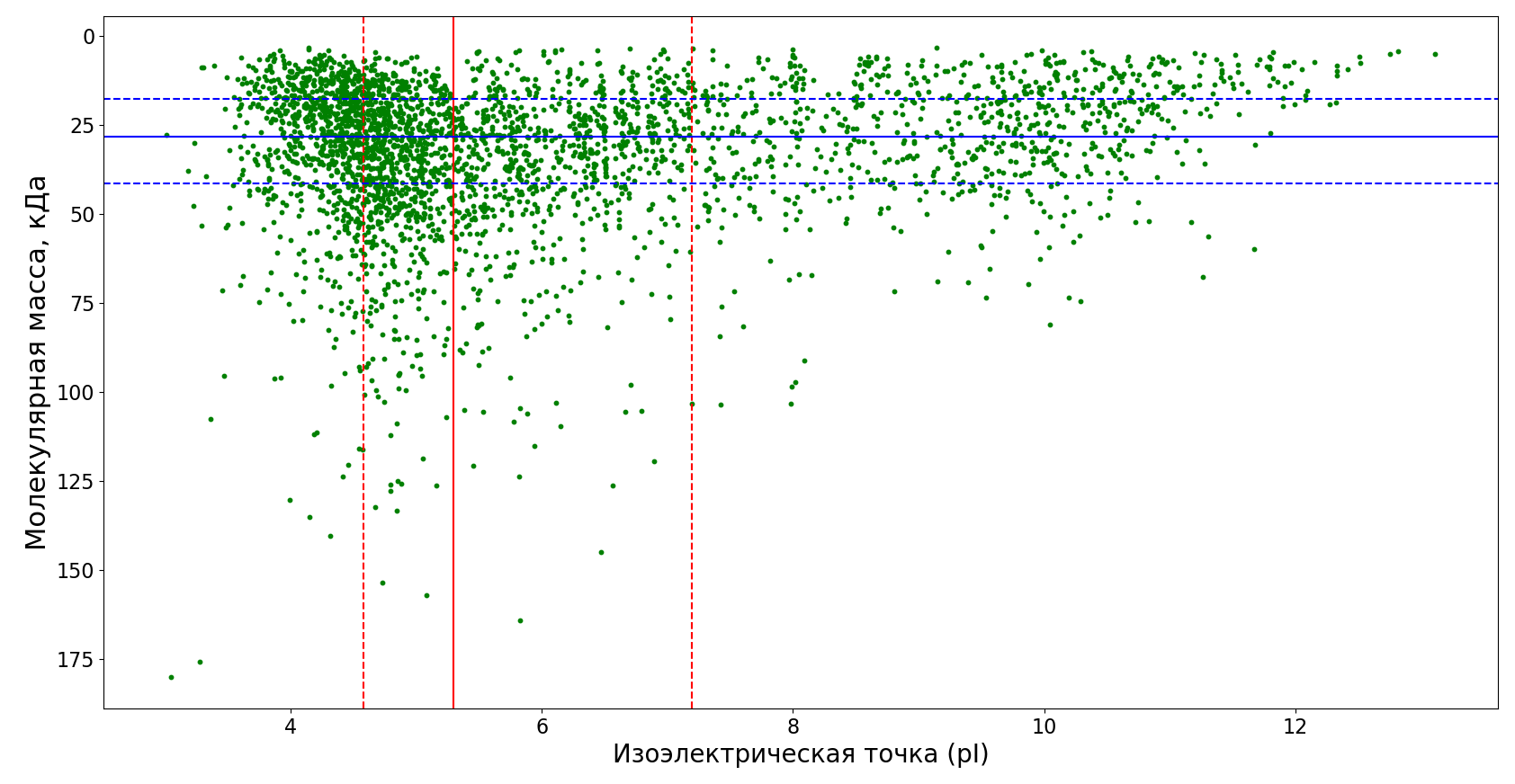
Медианное значение pI белков протеома бектерии составляет 5.3, молекулярной массы - 28.2 кДа. Из графика можно видеть, что значительная часть белков имеют pI от 3.5 до 6 и молекулярную массу не более 75 кДа (1894 белка из 3287, данные получены при помощи скрипта, описанного далее). Затем были отдельно рассмотрены белки, имеющие харатеристики, значительно отклоняющиеся от медианных: белки, имеющие молекулярную массу 150 кДа и более, pI 12 и более, pI 3.5 и менее. Для этого был написан скрипт на языке Python (см. S13), фильтрующий белки из ранее полученного файла stat.out и создающий таблицу формата TSV с ID данных белков и значениями pI и молекулярной массы для каждого из них (см. S14).
Среди белков с большим молекулярным весом значительную часть составили гемолизин и бифункциональные гемолизин/аденилатциклазы - бактериальные токсины, вызывающие разрушение эритроцитов, а бифункциональные гемолизин/аденилатциклазы также способны нарушать нормальное функционирование клеток за счет некотролируемого синтеза цАМФ [1].
Белки, имеющие низкие значения pI весьма разнообразны, например, среди них есть белок содержащий домен EF-руки, функцией которого является связывание ионов кальция, а также несколько гемолизинов.
Среди представителей с высоким значением pI, как и можно было ожидать, большую часть составляют белки, по-видимому вступающие во взаимодействие с нуклеиновыми кислотами или нуклеотидами, например, белки рибосомных субъединиц, ДНК-связывающий активатор транскрипции GcvA и пептидил-тРНК-гидролаза ArfB, отвечающая за один из путей высвобождения рибосом, по каким-либо причинам остановившихся посреди мРНК или продолжающих безостановочный (non-stop) синтез (например, при потере стоп-кодона) [2].
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
ЛИТЕРАТУРА И ИСТОЧНИКИ
Szabo G, Gray MC, Hewlett EL. Adenylate cyclase toxin from Bordetella pertussis produces ion conductance across artificial lipid bilayers in a calcium- and polarity-dependent manner. J Biol Chem. 1994 Sep 9;269(36):22496-9. PMID: 8077197.
Chadani Y, Ono K, Kutsukake K, Abo T. Escherichia coli YaeJ protein mediates a novel ribosome-rescue pathway distinct from SsrA- and ArfA-mediated pathways. Mol Microbiol. 2011 May;80(3):772-85. doi: 10.1111/j.1365-2958.2011.07607.x. Epub 2011 Mar 21. PMID: 21418110.