Резюме
В данной работе представлен анализ протеома бактерии Helicobacter canadensis . Данные были получены с сайта NCBI. Таблица характеристик была обработана при помощи Excel. Полученные данные были оформленны в виде таблицы; данные по распределению длин белков представлены в виде гистограммы.
Ключевые слова
Протеом, Helicobacter camadensis
1. Введение
Бактерии рода Helicobacter обычно имеют форму изогнутого или спирального стержня, за что и получили такое название. Это грамотрицательные, микроаэрофильные, оксидаза-положительные бактерии с температурным оптимумом от 37 до 42 градусов.(1) Кардинальная характеристика этого таксона - это способность гидролизовать индоксил ацетат. (2)
Геном Helicobacter canadensis состоит из 1 кольцевой хромосомы длиной в 1,623,845 bp.(3)
2. Методы
При написании этой работы я активно использовал Microsoft Excel и Microsoft Office Word. Данные о протеоме и многие статьи были взяты с сайта NCBI (ссылка выше).
2.1 Получение информации
1) Скачал файл с сайта
2) Разархивировал его. Я использовал программу Winrar Archive.
3) Открыл текстовый файл с помощью Microsoft Excel.
2.2 Построение гистограммы распределений длин белков
1) Создал новый лист в Excel и скопировал в него колонку product_length из листа chr_table.
2) В первую ячейку колонки Шаг записал значение 0, во вторую – 25 и заполнил весь столбец до значения 1500, так как дальше количество белков определённой длины было незначительно, что мешало увидеть всю картину распределения белков по длинам.
3) В соседнюю колонку записал функцию СЧЁТЕСЛИМН/COUNTIFS. Итоговая формула выглядела так: “=СЧЁТЕСЛИМН([столбец product_length]; ”>=”&[ячейка из колонки Шаг в той жестроке];[столбец product_length]; "<" &[ячейка из колонки Шаг ниже на 1 строку])”.
2.3 Построение таблицы числа генов по цепям ДНК
1) Создал новый лист в Excel.
2) Назвал в нем 4 колонки (слева направо): Цепь ДНК/Тип генов, Белки, Псевдогены, Гены РНК.
3) Добавил в первую колонку ещё 2 строки (сверху вниз): Прямая цепь ДНК, Обратная цепь ДНК.
4) Для подсчёта количества генов на прямой цепи ДНК, кодирующих белки использовал формулу: “СЧЁТЕСЛИМН([столбец strand из genes]; ”+”;[столбец # feature из genes];”CDS”)”. Для подсчёта количества генов на обратной цепи ДНК, кодирующих белки использовал формулу: “СЧЁТЕСЛИМН([столбец strand из genes]; ”-”;[столбец # feature из genes];”CDS”)”.
5) Для подсчёта количества генов на прямой цепи ДНК, кодирующих псевдогены использовал формулу: “СЧЁТЕСЛИМН([столбец strand из chr_table]; ”+”;[столбец class из chr_table];”pseudogene”)”. Для подсчёта количества генов на обратной цепи ДНК, кодирующих псевдогены использовал формулу: “СЧЁТЕСЛИМН([столбец strand из chr_table]; ”- ”;[столбец class из chr_table];”pseudogene”)”.
6) Для подсчёта количества генов на прямой цепи ДНК, кодирующих РНК использовал формулу: “СЧЁТЕСЛИМН([столбец strand из chr_table]; ”+”;[столбец # feature из chr_table];”ncRNA”) + СЧЁТЕСЛИМН([столбец strand из chr_table]; ”+”;[столбец # feature из chr_table];”tRNA”) + СЧЁТЕСЛИМН([столбец strand из chr_table]; ”+”;[столбец # feature из chr_table];”rRNA”)”. Для подсчёта количества генов на обратной цепи ДНК, кодирующих РНК использовал формулу: “СЧЁТЕСЛИМН([столбец strand из chr_table]; ”- ”;[столбец # feature из chr_table];”ncRNA”) + СЧЁТЕСЛИМН([столбец strand из chr_table]; ”- ”;[столбец # feature из chr_table];”tRNA”) + СЧЁТЕСЛИМН([столбец strand из chr_table]; ”-”;[столбец # feature из chr_table];”rRNA”)”.
2.4 Построение круговой диаграммы
1) Аналогично предыдущему пункту подсчитал общее количество псевдогенов, кодирующих генов и генов РНК. (“СЧЁТЕСЛИМН([столбец 3 feature из genes];”что необходимо найти”)”)
2) Создал на основе этих данных круговую диаграмму для достижения большей наглядности.
2.5 Гипотетические белки
1) С помощью функции СЧЁТЗ/COUNTA подсчитал количество всех белков
2) С помощью функции СЧЁТЕСЛИМН подсчитал ко-личество гипотетических белков.
3) На основе полученных данных построил круговую диаграмму.
2.6 Рибосомальные белки
1) При помощи фильтра отсортировал только рибосомальные белки.
2) Создал новый лист Excel и скопировал данные туда.
3) Удалил все дубликаты.
4) С помощью функции СЧЁТЕСЛИМН подсчитал количество генов определённого типа.
5) Создал гистограмму для наглядного отображения.
3. Результаты
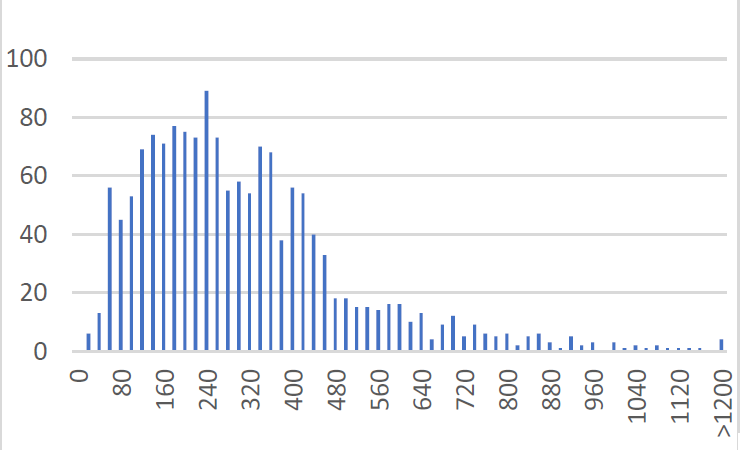
3.1 Изучение длин белков
На данной гистограмме (рис 1) хорошо видно, что наибольшее количество белков Helicobacter Canadensis имеют длину от 50 до 450. При данном шаге хорошо заметен изгиб, который характерен многим бактериям (в интервале 240 – 360). Также предоставляю варианты гистограмм с другими шагами.
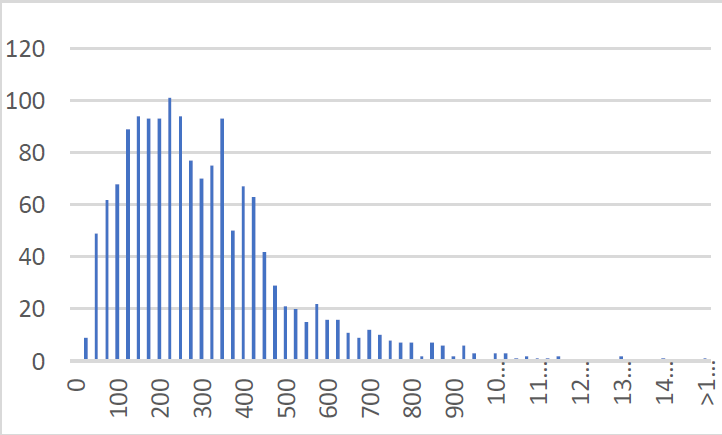
3.2 Изучение генов на прямой и обратных цепях ДНК
3.2.1 Таблица распределения генов на прямой и обратной цепях ДНК протеома.
На данной таблице видно, что распределение генов на прямой и обратной цепи ДНК равномерно, за исключением небольшого преобладания генов белков на прямой цепи ДНК по сравнению с обратной цепью ДНК. Но стоит учесть, что протеом данной бактерии не был полностью расшифрован.
| Цепь ДНК | Гены Белков | Псевдогены | Гены РНК |
|---|---|---|---|
| Прямая Цепь ДНК | 860 | 8 | 23 |
| Обратная Цепь ДНК | 675 | 11 | 26 |
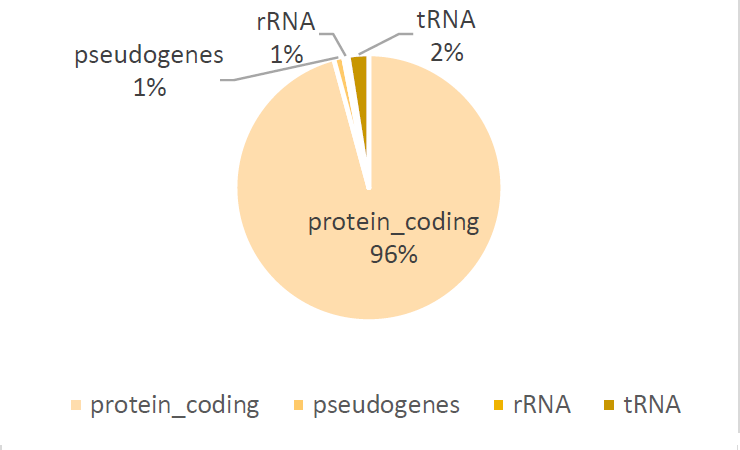
3.2.2 Соотношение псевдогенов, генов кодирующих белок, генов РНК и hypothetical genes
Интересно заметить, что у данной бактерии сравнительно небольшое число псевдогенов. Основную часть генов занимают гены кодирующие белки, что логично.
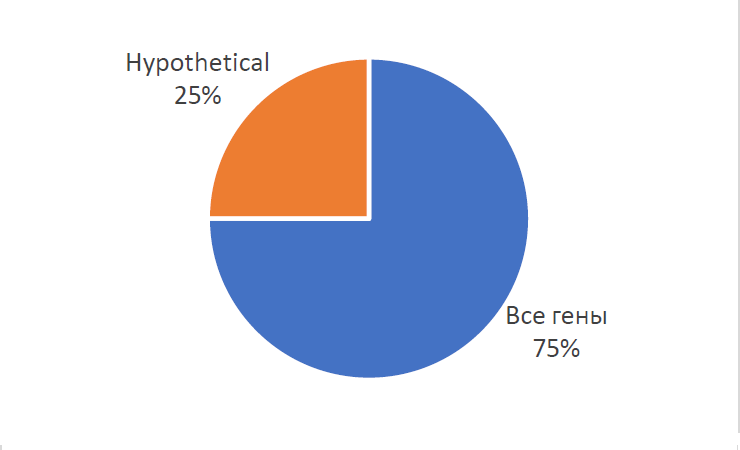
3.3 Гипотетические белки
В биохимии, гипотетический белок представляет собой белок, существование которого было предсказано, но для которых существует недостаток экспериментальных доказательств того, что она выражается в естественных условиях. В протеоме Helicobacter Canadensis насчитывается 528 таких белков, что составляет 25% ото всех белков.
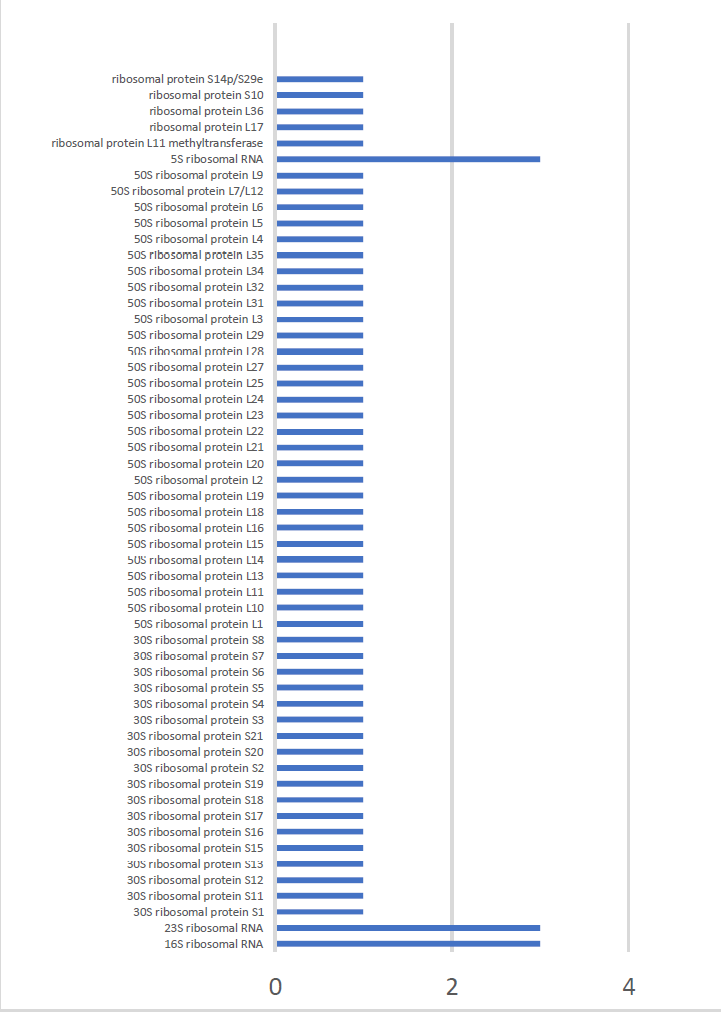
3.4 Рибосомальные белки
Данная гистограмма показвает, что в основном все рибосомаль-ные белки содержатся в единственном экземппляре. Вот те, что встречаются более одного раза:
16S ribosomal RNA
23S ribosomal RNA
5S ribosomal RNA
Скорее всего это связано с повышенной экспрессией генов или же это спонтанная мутация.
Заключение
Был проведён анализ протеома бактерии Helicobacter Canadensis . В ходе работы были рассмотрены интересные факты о данной бактерии, распределение белков по длинам и соотношение различных белков в протеоме этого организма. Было выяснено, что протеом не был до конца расшифрован, из-за чего данные могут не обладать 100% точностью.
Благодарности: Хочу выразить благодарность следующим моим однокурсникам: Татьяна Миньковская, Татьяна Романова, Владимир Ноздрин. Так же спасибо преподавателям информатики за столь интересное задание!
Список литературы
1. Campylobacter and Helicobacter // Medical Microbiology / edited by Samuel Baron. — 4th edition. — Galveston: The University of Texas Medical Branch, 1996. — 1273 p. — ISBN 0-9631172-1-1.
2. Inglis; Douglas, G.; McConville, Malcolm; de Jong, Anno (2006). "Atypical Helicobacter canadensis strains associated with swine". Applied and Environmental Microbiology. 72 (6): 4464–4471. doi:10.1128/aem.02843-05. PMC 1489614. PMID 16751570
3. Loman, N. J.; et al. (2009). "Genome sequence of the emerging pathogen Helicobacter canadensis". Journal of Bacteriology. 191 (17): 5566–5567. doi:10.1128/jb.00729-09. PMC 2725615. PMID 19542273.