
1. Описание домена в Pfam
В этой части практикума мы должны были описать белковый домен в базе данных Pfam, используя данный веб-сервис (ссылка). Некоторая информация приведена в Таблице 1 (ниже).
| ID | P53_HUMAN |
|---|---|
| AC | P04637 |
| Имена (белка) | У этого белка много имён (что не удивительно в связи с его популярностью). Вот некоторые из них: Клеточный опухолевый антиген p53, опухолевый супрессор p53, Фосфопротеин p53, Антиген NY-CO-13, Хранитель генома (the Guardian of the Genome), TRP53 (transformation related protein). |
| Функция (белка) | P53 является супрессором опухелового роста, мутации в этом белке связаны с большой долей всех видов рака человека. |
| Имя (домена) | P53 DNA-binding domain |
| Функция (домена) | Этот домен находится в транскрипционных факторах P53, где он отвечает за ДНК-связывание. ДНК-связывающий домен состоит из β-сэндвича, образованного из 9 нитей в 2 листах с топологией греческого ключа (меня это очень порадовало). |
| Количество последовательностей (full) | 872 |
| Число последовательностей в выравнивании seed | 51 |
| Число доменных архитектур с данным доменом | 26 |
| Домены приятели | P53_tetramer - P53 tetramerisation motif (тетрамеризация жизненно важна для активности p53 in vivo), SAM-2 - sterile α-motif (важен для стабильности домена) |
| Число 3D структур доменов из разных последовательностей | 4 |
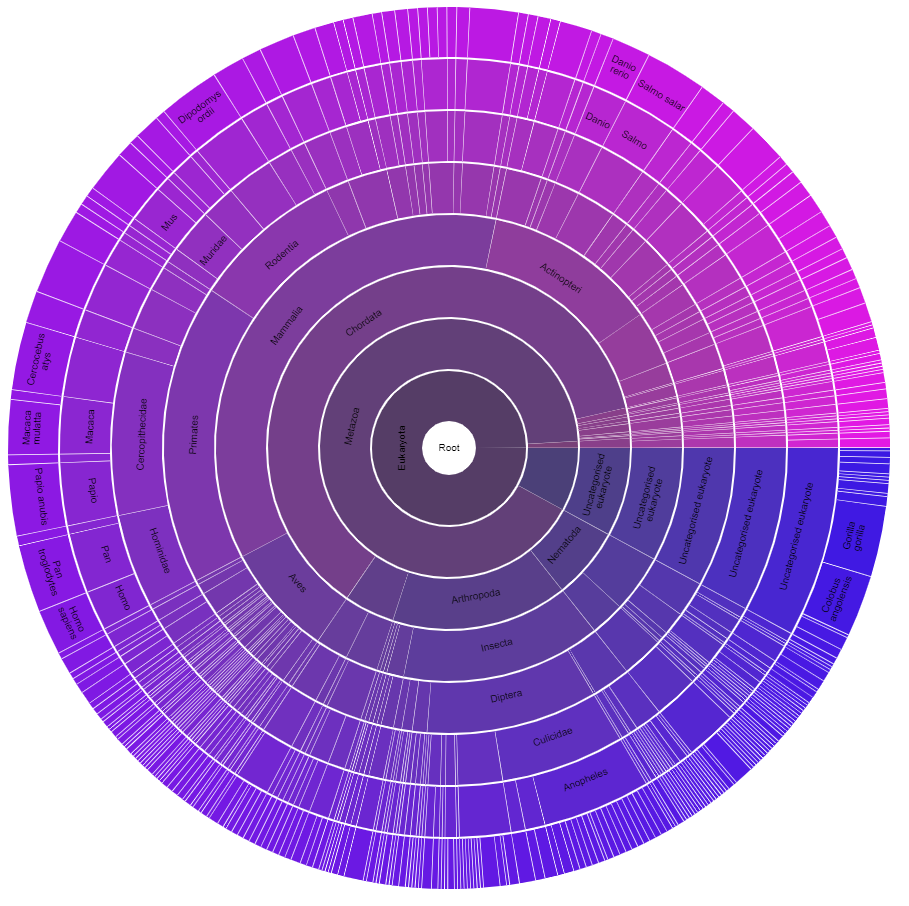
| Число белков с доменов по некоторым таксонам | Все (282) вида принадлежат к домену Eukaryota, 263 - царство Metazoa, остальные принадлежат к 2 неопознанным царствам (см. ниже). Думаю, что этого не достаточно, так что приведу некоторые типы: 109 - Chordata, Arthropoda - 81, Nematoda - 40. |
| Дата последнего изменения HMM профиля (файл) | Sat Aug 4 06:35:12 2018 |
| Число позиций в HMM профиле | 194 |
Комментарий к пункту с таксонами. Есть Uncategorised eukaryote (надпись повторяется в группе до вида) и просто no kingdom (далее no philum и no class). Но, что странно, в 1 группу отнесены некоторые гориллы, колобусы, мухи, хорёк, собака и тд. Скорее всего эту группу пока просто не отсортировали, но это странно. 2 группа, это часть клады Заднежгутиковые или Opisthokonta, но так как в современной систематике за этими таксонами не закреплён никакой ранг (?), то и в Pfam он отображается соответственно.
2. Анализ выравнивания из Pfam
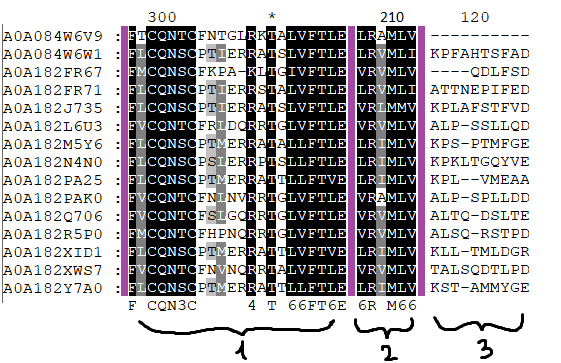
Для того, чтобы проанализировать выравнивание из базы данных Pfam, нам надо было взять небольшую выборку последовательностей белков с доменом, пользуясь Sunburst. Я взял всем известный род Anopheles (именно его представитель переносит возбудителя малярии). Мой выбор пал на него, так как это был единтсвенный род (предположительно внутри одного рода не будет очень сильных изменений белка), в котором было достаточное количество последовательностей и видов (38 последовательностей и 19 видов). Был скачан файл в формате fasta (тут). В программе JailView было проведено выравнивание с алгоритмом Muscle (проект jvp). После этого была проведена ревизия выравнивания: были удалены последовательности, подозрительно отличающиеся в консервативных участках, затем при помощи функции Remove Redundancy были удалены слишком похожие последовательности (результат) После я перевёл выравнивание в формат msf, чтобы открыть его в Genedoc (тут). При помощи программы Genedoc, информация была обработана и я нашёл консервативный, полуконсервативный и неконсервативный блоки (представлены на картинке снизу, 1-консервативный, 2-полуконсервативный, 3-неконсервативный. Слева представлены названия, для удобства я разделил блоки фиолетовыми полосками).
Поиск белков с данным доменом Pfam
При помощи UniProt были найдены белки содержащие данный домен (PF00870), таблицу можно получить тут. Число находок в SwissProt было равно 40, а в TrEMBL - 2477. Так как в 1 задании я рассматривал домен Эукариоты, то и здесь я провёл поиск по нему. Всего к ним отнесено 2515, а для 1 таксономия неизвестна (пуста). При помощи команды "=СЧЁТЕСЛИМН(I:I;"PF00870;PF07710;")" я посчитал количество белков с такой доменной структурой. Их оказалось 476 (в Pfam - 26). Я не смог найти аналог своего домена Pfam в базе данных Prosite (ссылка). Скорее всего это связано с тем, что размер базы данных у них сильно меньше.
Общий вывод
В заключение, основываясь на полученных данных, можно сказать, что количество белков в базе данных Pfam гораздо меньше чем в UniProt, хотя она и имеет свои преимущества (расположение доменов белков, например). Что логично, учитывая, что источников информации у UniProt в разы больше.