
1. Определение функции и таксономии нуклеотидной последовательности
В данном практикуме мы продолжаем работать с последовательностью, полученной при анализе секвенирования. Использовался blastn, так как изначально про эти последовательности мне ничего не известно и мне не очень критична степень идентичности. Также необходимо увеличить порог на E-value, ведь нам, опять же, нужно хоть что-то. Чтобы ускорить поиск, мы исключили из базы образцы неизвестного происхождения и метагеномы. Также я увеличил длину слова (10).
Как можно предположить из выдачи Blast, данная последовательность является последовательностью гистона H3, и, скорее всего, принадлежит ракообразному из рода Gammarus (можно было бы предположить и вид Gammarus italicus, но построенное отдельно филогенетическое дерево в JalView говорит о том, что: либо это не тот вид, либо я очень сильно обрезал последовательность при анализе).

Рис.2. Ракообразное из рода Gammarus
2. Поиск генов белков в неаннотированной нуклеотидной последовательности
В данной части практикума необходимо было найти в выбранной последовательности ген и предсказать его функцию. Для этого я взял контиг из генома Большой Синицы ("Great tit" или Parus major NCBI:txid9157). Чтобы не перегружать Blast, я выбрал контиг с AC JRXK01000008.1 и длиной 3870 (файл). Далее необходимо было загрузить нуклеотидную последовательность в blastx, так как необходимо найти белок. В параметрах поиска отметил exclude 'Great tit txid9157'. Судя по выдаче Blast (файл), в данной последовательности содержится ген ATOH1, кодирующий белок Protein atonal homolog 1 (ATOH1) или ato. Данный белок участвует в нейрогенезе, необходим для формирования хордотональных органов (органы чувств у некоторых членистоногих, относящиеся к периферической нервной системе) и фоторецепторов, для человека "генетическая делеция ATOH1 показала, что этот фактор транскрипции необходим для развития мозжечковых гранулярных нейронов, волосковых клеток внутреннего уха, промежуточных нейронов в спинном мозге, клеток Меркеля в коже, секреторных клеток кишечника" wiki.
3. Интепретация карты локального сходства гомологичных хромосом двух бактерий
В данной части практикума необходимо было построить карту локального сходства гомологичных хромосом 2-х бактерий. Необходимо было выбрать бактерии одного рода, но разных видов. Я выбрал род Bifidobacteriaceae. Чтобы получить хорошую карту, я пострался найти наиболее близких родственников (использовал филогенетическое дерево из википедии), так как в остальных случаях карта просто не несёт почти никакой информации. Также необходимо было учитывать наличие полного расшифрованного генома. Bifidobacterium longum strain CACC 517 и Bifidobacterium breve strain lw01 были выбраны для данной задачи. После перебора нескольких вариантов длины слова, лишний шум был убран и была получена приемлемая карта.
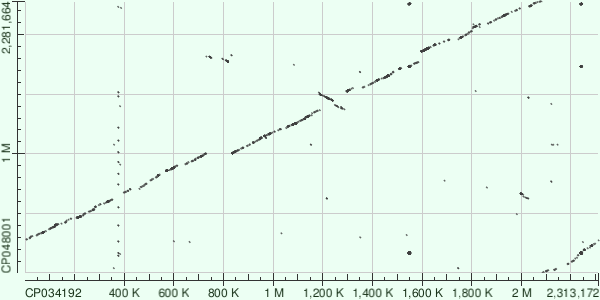
Рис.3. Карта локального сходства B. longum и B. breve
На данной карте хорошо видна 1 инверсия (на карте она отображается, как повёрнутый на 45 градусов участок) а также множество мелких инсерций и делеций.