
1. Составление списка гомологичных белков, включающих паралоги
Сначала все файлы с протеомами были слиты в один при помощи функции cat (ссылка). Затем файл был проиндексирован при помощи makeblastdb. Затем при помощи blastp были найдены гомологи для cpx_ecoli (ссылка). При помощи питона были выделены в отдельный (файл) ID полученных гомологов. Файл был передан JalView, полученные последовательности были выравняны muscle на kodomo (так как почему-то выдавалась ошибка сервера) (файл).
2. Реконструкция и визуализация
При помощи MEGA было построено дерево путём максимального правдоподобия. Его скобочная формула:
((((((CLPX_NEIMA,CLPX_AROAE),CLPX_SACD2),CLPX_HAEIN),(CLPX_ROSDO,(CLPX_ACICJ,(CLPX_BARHE,CLPX_BRUSU)))),(((HSLU_SACD2,HSLU_HAEIN),HSLU_AROAE),(A5FYD7_ACICJ,(HSLU_ROSDO,(HSLU_BARHE,HSLU_BRUSU))))),(Q16C81_ROSDO,(((RUVB_NEIMA,RUVB_BARHE),RUVB_ROSDO),((A0A0H3GCZ6_BRUSU,A0A0H3LXZ4_BARHE),(Q167Z2_ROSDO,(FTSH_HAEIN,A5FVF9_ACICJ))))),A0A0U1RJ22_NEIMA);
Два гомологичных белка будем называть ортологами, если они из разных организмов и разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. Два гомологичных белка из одного организма будем называть паралогами.
Ортологи:
🐾 CLPX_NEIMA и CLPX_AROAE
🐾 CLPX_SACD2 и CLPX_HAEIN
🐾 HSLU_SACD2 и HSLU_HAEIN
Паралоги:
🐾 CLPX_HAEIN и HSLU_HAEIN
🐾 CLPX_SACD2 и HSLU_SACD2
🐾 Q167Z2_ROSDO и Q16C81_ROSDO
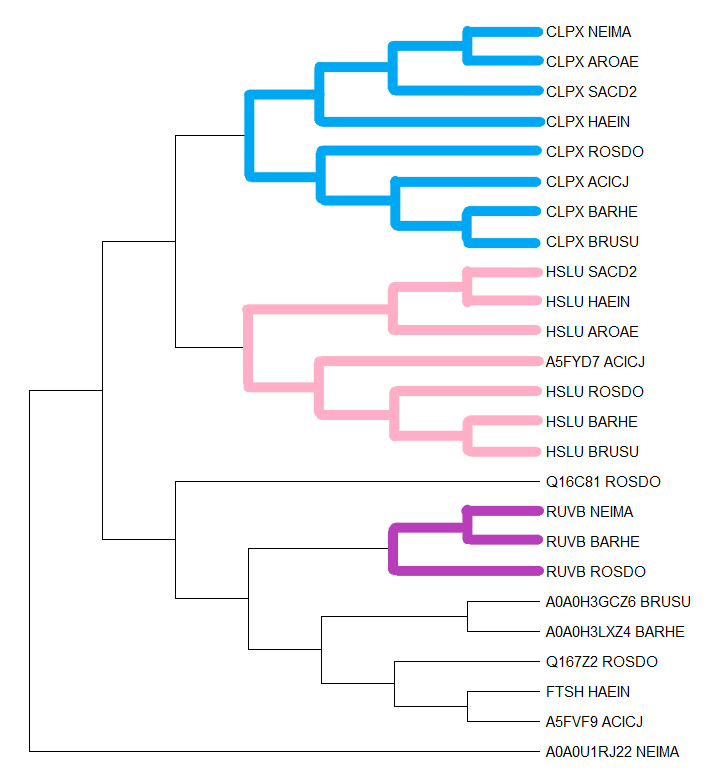
Рис. 1. Разными цветами показаны разные ортологичные группы.

Рис. 2. Ортологичные группы "схлопнуты".