
Вычисление информационного содержание (IC) последовательностей Козак и построение LOGO этого сигнала
В этом задании я вычислил информационное содержание (IC) последовательностей Козак и построил LOGO этого сигнала, основываясь на данных последовательностях (файл).
Поиск сайтов разрывной транскрипции sgmRNA в геноме коронавируса
В этом задании я, основываясь на выполнении задания 6.2 предыдущего практикумума, должен был проанализировать полученный мотив. Для этого я подготовил входной файл, используя веб-версию MEME и указав, что мне необходим только 1 мотив (файл). В качестве базы использовалась ДНК вируса, так как требуется посмотреть на то, где расположен "мой" мотив.
Для сравнения были выбраны 3 вируса, выбранных на чисто случайном основании привлекательности названия: Bulbul coronavirus HKU11; Pipistrellus bat coronavirus HKU5 и исходный Shrew Coronavirus.
Из них были подготовлены файлы в fasta-формате, которые вместе с файлом с 1 мотивом были переданы FIMO [1]. Получили 3 файла: Shrew-CoV, Bulbul-CoV, Pipistrellus-CoV.
Ниже представлены отредактированные таблицы выдачи. Я удалил колонку "Motiv ID", так как я ищу единственный мотив ("CAMGWAWWACMCACWSWGGMTSTWYKYKTGGTCYARMMC"), а также я удалил "Alt ID", так как для всех таблиц он имеет вид "Meme-1", что не несёт дополнительной информации, но ухудшает восприятие.
| Sequence Name | Strand | Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|---|---|
| NC_046955.1 | + | 24815 | 24853 | 4.47e-24 | 1.19e-19 | CAAGTAATACCCACACAGGATGTTTGCTTGGTCTAAACC |
| NC_046955.1 | + | 278 | 316 | 2.29e-16 | 3.04e-12 | CACGAATAACACACTGTGGCTCTACTTGTGGTCCAGCAC |
| NC_046955.1 | + | 7989 | 8027 | 9.02e-05 | 0.634 | CTCGATAACACCACAGTTGTTGAATGATTATGCCAATAT |
| NC_046955.1 | + | 16089 | 16127 | 9.51e-05 | 0.634 | GCACAATCACACACTTATGAAGCTCTTTCTGACAAATAC |
Таблица 1. Поиск мотива в геноме Shrew-Cov.
| Sequence Name | Strand | Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|---|---|
| NC_009020.1 | + | 26549 | 26587 | 3.04e-05 | 0.463 | TGTGATAAACCCACACATTGTATTCGCTTGGTTTTTGAT |
| NC_009020.1 | + | 16037 | 16075 | 3.28e-05 | 0.463 | AATGTCTTCATGTCAGAGGCTAAATGCTGGGTGGAAACC |
| NC_009020.1 | + | 21302 | 21340 | 6.15e-05 | 0.463 | GAAACAAATGAAGCAAAGGCGCTTTTCTTTGTGTATCTC |
| NC_009020.1 | + | 22077 | 22115 | 6.35e-05 | 0.463 | CAAGTGGAGTCCTTTGATGATGGCTTTGTGGTCCGTATT |
| NC_009020.1 | + | 16521 | 16559 | 7.64e-05 | 0.463 | TACGAAGACCCTTCCTTTGCTGTAAGTGTTGTTATGACC |
Таблица 2. Поиск мотива в геноме Pipistrellus bat coronavirus HKU5.
| Sequence Name | Strand | Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|---|---|
| NC_011547.1 | + | 14769 | 14807 | 5.35e-05 | 0.678 | TACAAATTACAAACTGGTGATGTACTTGTACTTATGGCA |
| NC_011547.1 | + | 15261 | 15299 | 6.57e-05 | 0.678 | TATGAGCTTTCTTCAGTAAATGCTCGTTTGGTTTACAAC |
| NC_011547.1 | + | 10378 | 10416 | 7.98e-05 | 0.678 | CAAATTTTGCCTATACATGCTGTAGTGGGTGTGTCAAAT |
Таблица 3. Поиск мотива в геноме Bulbul coronavirus HKU11-934.
Как видно из представленных таблиц, мотив встречается в исходном геноме 4 раза из 5, в геноме Bulbul-Cov - 3 раза из 9 (но если не считать "nonstructural protein", то всего 5), в геноме Pipistrellus bat-Cov - 3 из 9 (опять же, если не считать "hypothetical protein", то всего 6).
Далее при помощи Meme был построен logo последовательности Козак для исходного вируса. Для logo были взяты участки -6 7, чтобы провести сравнение с человеческой последовательностью Козак.
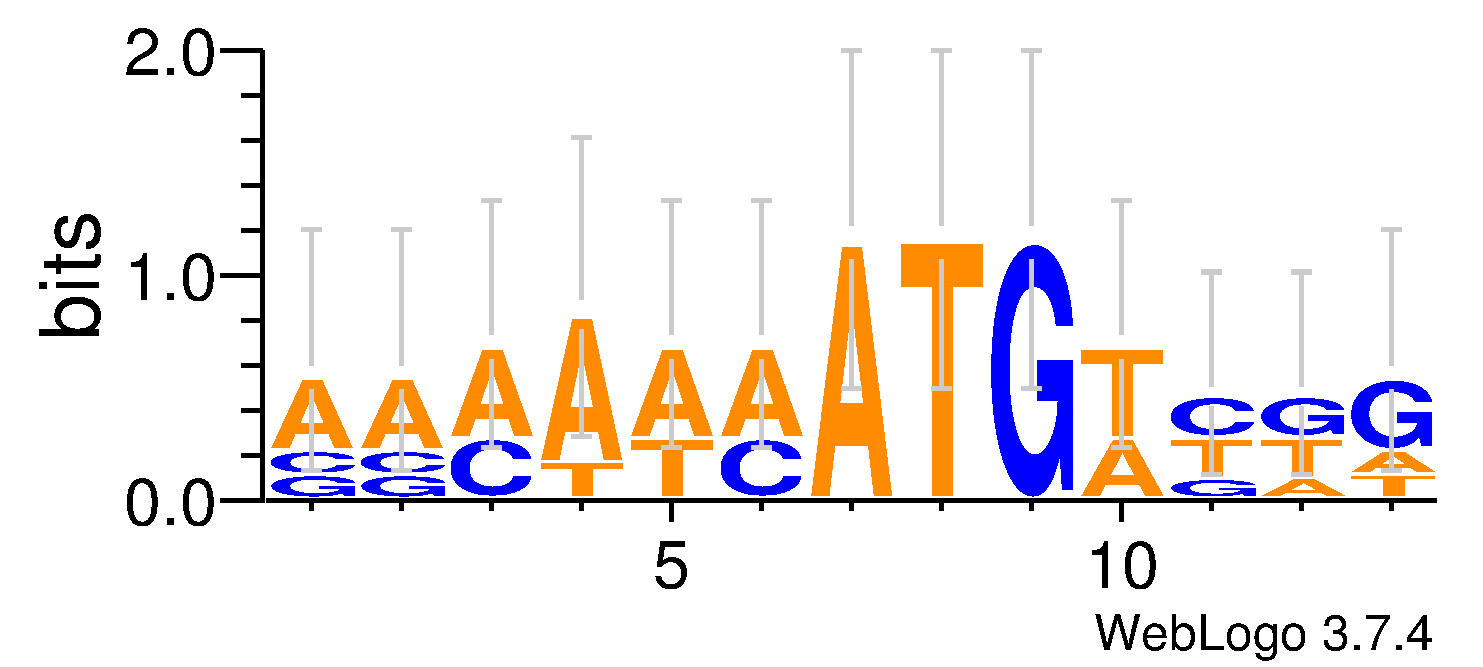
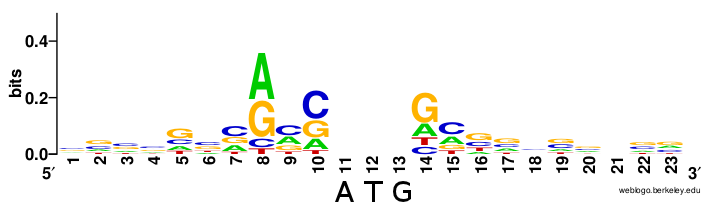
Рис. 1. Сравнение последовательностей Козак Shrew-Cov (слева) и Human [2] (справа).
Как видно, сходства почти нет. Только в позициях -3 (наиболее вероятен Аденин) и 5 (наиболее вероятен Цитозин).
То, что данный мотив находится в 4 из 5 генах данного вируса, скорее всего, подверждает правильность находки. Но вот то, что этот же мотив можно найти в других вирусах семейства Coronaviridae, вероятно, говорит о том, что он не специфичен для данного вида.
Update
После того, как я переделал 6 практикум и немного улучшил свой мотив, я решил переделать и FIMO: Shrew-Cov; Bulbul-Cov и Pipistrellus-CoV. Полученный мотив: TACCMGGTGTTA.
| Motif ID | Sequence Name | Strand | Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|---|---|---|
| 1 | NC_046955.1 | + | 24771 | 24782 | 6.75e-08 | 0.00159 | TACCAGGTGTTA |
| 1 | NC_046955.1 | + | 26 | 37 | 1.21e-07 | 0.00159 | TACCCGGTGTTA |
| 1 | NC_046955.1 | + | 2441 | 2452 | 2.01e-06 | 0.0176 | TACCAAGTGTTA |
| 1 | NC_046955.1 | + | 15334 | 15345 | 7.36e-06 | 0.0483 | TACCGGATGTTA |
| 1 | NC_046955.1 | + | 10889 | 10900 | 6.78e-05 | 0.301 | TAACCGTTGTTA |
| 1 | NC_046955.1 | + | 13459 | 13470 | 8.13e-05 | 0.301 | AACCTGGTGGTA |
| 1 | NC_046955.1 | + | 3707 | 3718 | 9.17e-05 | 0.301 | TAGCTGGTGTTG |
| 1 | NC_046955.1 | + | 8711 | 8722 | 9.17e-05 | 0.301 | TGCATGGTGTTA |
Таблица 4. Поиск укороченного мотива в геноме Shrew-Cov.
| Motif ID | Sequence Name | Strand | Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|---|---|---|
| 1 | NC_009020.1 | + | 23060 | 23071 | 7.36e-06 | 0.223 | TACCGGTTGTTA |
| 1 | NC_009020.1 | + | 28060 | 28071 | 1.89e-05 | 0.286 | TACCAGCTGTTC |
| 1 | NC_009020.1 | + | 14708 | 14719 | 3.4e-05 | 0.343 | TACAAGGAGTTA |
Таблица 5. Поиск укороченного мотива в геноме Pipistrellus-Cov.
| Motif ID | Sequence Name | Strand | Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|---|---|---|
| 1 | NC_011547.1 | + | 17420 | 17431 | 2.88e-05 | 0.299 | TACCAGATGTAA |
| 1 | NC_011547.1 | + | 23491 | 23502 | 4.14e-05 | 0.299 | TAGCAGGTATTA |
| 1 | NC_011547.1 | + | 18661 | 18672 | 6.93e-05 | 0.299 | TGCCTGGTGTTG |
| 1 | NC_011547.1 | + | 21555 | 21566 | 6.93e-05 | 0.299 | TGCCGGGTGTTG |
| 1 | NC_011547.1 | + | 7277 | 7288 | 7.59e-05 | 0.299 | AACCTGGTGTTT |
| 1 | NC_011547.1 | + | 6770 | 6781 | 8.74e-05 | 0.299 | TACCTGGTATTC |
| 1 | NC_011547.1 | + | 1391 | 1402 | 9.17e-05 | 0.299 | TAGCTGGTGTTG |
| 1 | NC_011547.1 | + | 3497 | 3508 | 9.17e-05 | 0.299 | TAACTGGTGTTG |
Таблица 6. Поиск укороченного мотива в геноме Bulbul-Cov.
Данный мотив встречается в вирусах того же семейства достаточно часто, что, скорее всего, говорит о его неспецифичности.
[1] Charles E. Grant, Timothy L. Bailey, and William Stafford Noble, "FIMO: Scanning for occurrences of a given motif", Bioinformatics, 27(7):1017-1018, 2011.
[2] Kozak consensus sequence (ссылка)