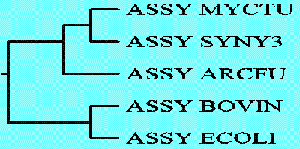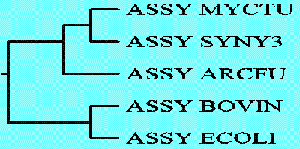Филогенетические деревья, реконструированные разными способами.
На главную страницу второго семестра
| Название белка (ID) |
Название домена |
Идентификатор Pfam |
Положение в последовательности |
| ASSY_MYCTU |
Arqinosuc synth |
PF00764 |
6-395 |
| ASSY_SYNY3 |
Arqinosuc synth |
PF00764 |
8-397 |
| ASSY_ECOLI |
Arqinosuc synth |
PF00764 |
14-417 |
| ASSY_ARCFU |
Arqinosuc synth |
PF00764 |
4-388 |
| ASSY_BOVIN |
Arqinosuc synth |
PF00764 |
8-405 |
Эталонное выравнивание доменов.
| |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
| |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
* |
|
|
|
|
|
|
|
|
2 |
0 |
|
|
|
|
|
|
|
|
|
* |
|
|
|
|
|
|
|
|
4 |
0 |
|
|
|
|
|
|
|
|
|
* |
|
|
|
|
|
|
|
|
|
|
|
|
| A |
S |
S |
Y |
_ |
M |
Y |
C |
T |
U |
|
: |
|
I |
L |
A |
Y |
S |
G |
G |
L |
D |
T |
S |
V |
A |
I |
S |
W |
I |
G |
K |
E |
T |
G |
- |
R |
E |
V |
V |
A |
V |
A |
I |
D |
L |
G |
Q |
G |
G |
- |
E |
H |
M |
D |
V |
I |
R |
Q |
R |
A |
L |
D |
C |
G |
A |
V |
E |
A |
V |
|
: |
|
5 |
5 |
| A |
S |
S |
Y |
_ |
S |
Y |
N |
Y |
3 |
|
: |
|
V |
L |
A |
Y |
S |
G |
G |
V |
D |
T |
S |
V |
C |
I |
P |
Y |
L |
M |
H |
E |
W |
G |
V |
E |
E |
V |
I |
T |
L |
A |
A |
D |
L |
G |
Q |
G |
- |
- |
D |
E |
L |
G |
P |
I |
Q |
E |
K |
A |
L |
R |
C |
G |
A |
V |
E |
S |
L |
|
: |
|
5 |
5 |
| A |
S |
S |
Y |
_ |
E |
C |
O |
L |
I |
|
: |
|
G |
I |
A |
F |
S |
G |
G |
L |
D |
T |
S |
A |
A |
L |
L |
W |
M |
R |
Q |
K |
G |
- |
- |
A |
V |
P |
Y |
A |
Y |
T |
A |
N |
L |
G |
Q |
P |
D |
E |
E |
D |
Y |
D |
A |
I |
P |
R |
R |
A |
M |
E |
Y |
G |
A |
E |
N |
A |
R |
|
: |
|
5 |
5 |
| A |
S |
S |
Y |
_ |
A |
R |
C |
F |
U |
|
: |
|
V |
L |
S |
Y |
S |
G |
G |
L |
D |
T |
T |
V |
C |
I |
P |
L |
L |
K |
E |
K |
Y |
G |
F |
D |
E |
V |
I |
T |
V |
T |
V |
D |
I |
G |
Q |
P |
E |
- |
A |
D |
I |
K |
Q |
A |
E |
E |
R |
G |
K |
K |
Y |
A |
D |
- |
K |
H |
Y |
|
: |
|
5 |
5 |
| A |
S |
S |
Y |
_ |
B |
O |
V |
I |
N |
|
: |
|
V |
L |
A |
Y |
S |
G |
G |
L |
D |
T |
S |
C |
I |
L |
V |
W |
L |
K |
E |
Q |
G |
- |
- |
Y |
D |
V |
I |
A |
Y |
L |
A |
N |
I |
G |
Q |
K |
- |
- |
E |
D |
F |
E |
E |
A |
R |
K |
K |
A |
L |
K |
L |
G |
A |
K |
K |
V |
F |
|
: |
|
5 |
3 |
| |
|
|
|
|
|
|
|
|
|
|
|
|
|
6 |
a |
5 |
S |
G |
G |
6 |
D |
T |
3 |
|
|
6 |
|
|
6 |
|
|
|
|
|
|
|
|
v |
|
|
|
|
|
1 |
6 |
G |
Q |
|
|
|
|
|
|
|
|
|
|
|
4 |
a |
|
|
|
g |
a |
|
|
|
|
|
|
|
|
|
| |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
| |
|
|
|
|
|
|
|
|
|
|
|
|
|
6 |
0 |
|
|
|
|
|
| A |
S |
S |
Y |
_ |
M |
Y |
C |
T |
U |
|
: |
|
V |
V |
D |
|
: |
|
5 |
8 |
| A |
S |
S |
Y |
_ |
S |
Y |
N |
Y |
3 |
|
: |
|
V |
I |
D |
|
: |
|
5 |
8 |
| A |
S |
S |
Y |
_ |
E |
C |
O |
L |
I |
|
: |
|
L |
I |
D |
|
: |
|
5 |
8 |
| A |
S |
S |
Y |
_ |
A |
R |
C |
F |
U |
|
: |
|
T |
I |
D |
|
: |
|
5 |
8 |
| A |
S |
S |
Y |
_ |
B |
O |
V |
I |
N |
|
: |
|
I |
E |
D |
|
: |
|
5 |
6 |
| |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
D |
|
|
|
|
|
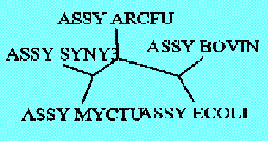
Построение дерева по алгоритму UPGMA.
Результаты в xl. книге. UPGMA.xls
Филогенетическое дерево представляет собой граф, состоящий из вершин и ребер (ветвей),
в котором любые две вершины соединяет единственное ребро, и между любыми двумя точками
существует единственный маршрут. Вершины обозначают таксономические группы, а ребра -
отношения родства. Длина ребра обычно отражает число эволюционных изменений произошедших
во времени между потомком и предком. Порядок ветвления ребер дерева называется топологией.
Наиболее часто деревья рисуются так, что длина ребра на рисунке отражает время дивергенции
и не пропорциональны собственно длинам ребер в числе эволюционных замен.
Получение и сравнение двух изображений построенного дерева.
(((ASSY_MYCTU:24,ASSY_SYNY3:24):8,ASSY_ARCFU:32),(ASSY_BOVIN:30.5,ASSY_ECOLI:30.5));
Наблюдения.
Было получено 2 графических файла. Две различных программы (drawtree и drawgram) выдали
совершенно разные изображения одного и того же дерева.
1.Программа drawtree выдала изображение, больше похожее на рисунок. В данном случае,
на этом рисунке можно увидеть дерево, которое не имеет выпаженного корня.
Но, конечно, имеются ребра (ветви), также мы можем наблюдать, что 4 ветви соединяются
в 2 узла (каждые 2 ветви соединяются в единственный узел). И мы можем на этом рисунке
также увидеть подписи.
2.Программа drawgram выдала более структурированное изображение. Тут мы можем увидеть
подписи, ребра и конечно же узлы.
Отличие этих программ друг от друга состоит в том, что программа drawgram в отличае
от программы drawtree выдает изображение дерева в виде структуры, а не в виде рисунка.
Лучше всего мое представление о данном дереве отражает программа drawgram, т.к. в этом
случае, еще раз повторюсь, изображение выдается структурированным. Но хочу отметить,
что программа drawtree выдает изображение очень наглядное. Так же хочу заметить, что
сценарий эволюции удобнее писать по изображению, которое выдала программа drawgram.
Предположительный сценарий эволюции.
Примечание.
В моем примере у дерева не оказалось корня, из-за этого при написании сценария возникли
небольшие трудности.
Сценарий.
В моем случае, нельзя сказать какие последовательности произошли раньше, а какие позже
от гипотетической общей предковой последовательности. Происхождение ,в данном случае ,
последовательностей можно считать равноправным, т.к. это дерево не укорененное . И корень
этого дерева может теоретически находиться в любом месте. Но, в данном случае, можно
просмотреть как происходило разветвление последовательностей. Разветвление идет
на последовательности ASSY_BOVIN и ASSY_ECOLI, и на последовательности ASSY_ARCFU, ASSY_MYCTU
и ASSY_SYNY3.
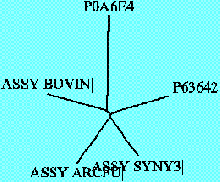
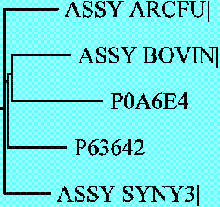
Получение и описание дерева по методу ближайших соседей.
(ASSY_ARCFU|:0.27108,((ASSY_BOVIN|:0.30267,P0A6E4:0.45947):0.01987,P63642:0.29504)
:0.01747,ASSY_SYNY3|:0.23149);
Наблюдения.
1.Было получено еще 2 графических файла с изображением одного и того же дерева,
но уже методом ближайших соседей. Как и в первом случае, 2 изображения изрядно
отличаются друг от друга способом подачи информации. В первом случае, мы видим
изображение, больше похожее на графический рисунок. Во втором случае, мы видим
структурированное изображение данного дерева.
2.NJ-дерево с UPGMA-деревом способом подачи информации друг от друга не отличаются,
но содержание отличается сильно. В результате мы видим сильные отличии в сценариях
эволюции, которые получились при реконструкции разными методами.
Предположительный сценарий эволюции.
Как было сказано выше, в моем случае, нельзя сказать какие последовательности
произошли раньше, а какие позже от гипотетической общей предковой последовательности.
Происхождение, в данном случае, последовательностей можно считать равноправным.
И корень этого дерева может теоретически находиться в любом месте. Но, в данном случае,
можно просмотреть, как происходило разветвление последовательностей.
Разветвление идет на последовательности ASSY_ARCFU и ASSY_SYNY3 , а затем на
ASSY_BOVIN и ASSY_ECOLI, и ASSY_MYCTU.
Вывод.
Мы предполагали, что наши белки эволюционировали с одинаковой скоростью, но после построения
деревьев мы видим, что белки эволюционировали с разной скоростью. Следовательно, в нашем
случае теория "Молекулярных часов" не работает. Так же можно сказать, что деревья построенные
по методу ближайших соседий не достоверные.
©Трембицкая Влада