Структура тРНК.
На главную страницу третьего семестра
Несколько слов о тРНК...
Молекулы тРНК самые маленькие (длиной около 80 нуклеотидов). Единичная цепь тРНК складывается,
формируя структуру, напоминающую клеверный лист с тремя петлями.
Подобно иРНК и рРНК, молекулы тРНК синтезируются в ядре путем спаривания оснований
с нуклеотидами ДНК в специфических тРНК-генах, а затем перемещаются в цитоплазму.
В синтезе белка тРНК способна связываться как со специфической аминокислотой, так и с
кодоном, кодирующим эту аминокислоту и расположенным на связанной с рибосомой иРНК.
Это позволяет ей быть связующим звеном между аминокислотой и кодоном иРНК для этой
аминокислоты.
Файл PDB - 1j1u. В этом файле описаны структуры тРНК и тирозил - тРНК синтетаза.
тРНК имеет идентификатор цепи В - 2.
Последовательность тРНК.

По данным PDB файла в состав тРНК входят 4 иона Mg. В цепи В - один с номером - 1501.
В цепи А - один 503. Два - не относятся ни к одной из цепей 502, 504.
тРНК имеет нумерацию: 501-574 (пропусков и/или вставок в нумерации нет), однако
три последних нуклеотида ("акцепторный хвост") ССА в структуре тРНК отсутствуют.
Анализ структуры, проведенный с помощью программы find_pair.
Для удобства работы с последовательностью был создан файл tRNA.pdb, который описывает
только структуру тРНК (без синтетазы).
По данным работы программы find_pair было построено
предсказание вторичной структуры тРНК.
В структуре имеются 4 спирали: акцепторная (501-507; 567-573) выделена красным,
D-спираль (510-513; 523-526) - желтым, антикодоновая (527-533, 539-545) - бирюзовым,
Т-спираль (550-555; 559-566, выпетливание - 560 и 561) - болотным.
Спирали в трехмерной структуре тРНК.
 Межмолекулярные взаимодействия.
Межмолекулярные взаимодействия.
- Стекинг. Было найдено два случая внеспирального стекинг-взаимодействия:
между G547 и A522, C507 и G550.
- Не Уотсон-Криковское спаривание. К случаям некомплементарного взаимодествия
относятся 4 пары нуклеотидов: U556 и G519, U534 и A537, A514 и U508, G515 и C549.
С помощью программы analyze был проведен анализ конформационно важных
торсионных углов тРНК.
Из анализа было определено, что спирали тРНК похожи на А-форму ДНК.
Поиск инвертированных последовательностей при помощи программы einverted.
Даже при значении "Minimum score threshold", которое равно нулю,
программа выдала единственный результат - акцепторный стебель.
: Score 21: 7/7 (100%) matches, 0 gaps
1 ccggcgg 7
|||||||
73 ggccgcc 67
Но зато правильно.
Причина, по которой другие спирали программой не распознаны, видимо индивидуальна
для каждой из них, но суть одна - недостаточная длина непрерывных комплементарных
пар. Так, D-стебель сам по себе состоит лишь из четырех пар, T-стебель имеет выпетливание,
а у антикодонового целых две пары некомплементарных оснований.
Анализ последовательности тРНК программой mfold.
Для выполнения этого задания потребовались следущие команды:
mfold SEQ=tRNA2.fasta
mfold SEQ=tRNA2.fasta P=10
mfold SEQ=tRNA2.fasta P=15
mfold SEQ=tRNA2.fasta P=20
mfold SEQ=tRNA2.fasta P=25
mfold SEQ=tRNA2.fasta P=30
mfold SEQ=tRNA2.fasta P=35
mfold SEQ=tRNA2.fasta P=40
mfold SEQ=tRNA2.fasta P=45
mfold SEQ=tRNA2.fasta P=50
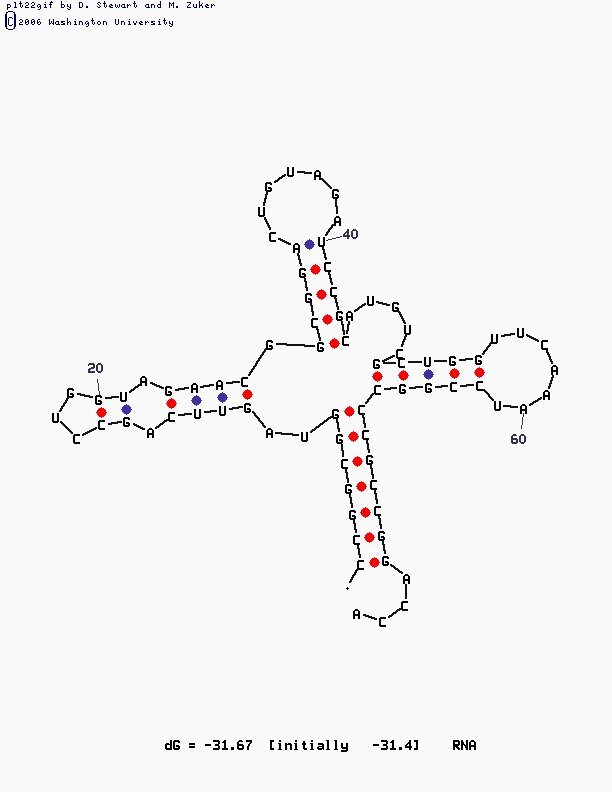
С помощью программы mfold была проведена попытка предсказать вторичную структуру
тРНК, максимально близкую к реальности.
Структуру, максимально близкую к реальности, программа mfold выдала на первом же
этапе, причем на этом этапе параметр "P" не указывался. Далее был задан параметр "P"
со значением равным 10 и программа выдала больше предполагаемых структур, что вполне естественно,
т.к. параметр "P" указывает, на сколько процентов выдаваемое предсказание структуры может
отличаться по своей вычисленной энергии от оптимального. Чем больше этот параметр, тем
больше вариантов вторичной стрктуры.
При значениях больше 15, программа выдавала совершенно бессмысленные варианты.
Эта программа хороша тем, что она сокращает время построения вторичной структуры.
Конечно, она будет отличаться от идеальной, но не на много.
©Трембицкая Влада

