 8 (926) 907 94 08
8 (926) 907 94 08
Всё на свете является чудом!
Классификация ферментов (Enzymes Classification - EC)
Выбрали соответствующий фермент (CLAT_HUMAN) из таблицы.
Далее, чтобы найти EC-код фермента, зная идентификатор Swiss-Prot, воспользуемся сервисом на биоинформатическом портале ExPASy. В окошко поиска вводим идентификатор, предварительно изменив базу данных для поиска на "ENZYMES". В результате получили страницу с информацией о ферменте.
Этот ресурс интересен и удобен тем, что он выдаёт помимо "своих" данных ещё и, так называемые, "Cross-references" (Перекрёстные ссылки), что даёт возможность найти всю интересующую нас информацию (подробнее о сборе информации...).
Prosite
Prosite состоит из документации записей описания белковых доменов, семейств и функциональных сайтов, а также связанных моделей и профилей для их идентификации.
Ссылка на запись о ферменте.
Brenda
Комплексная информационная система ферментов BRENDA является основной коллекцией функциональных данных о ферментах, доступных для научного сообщества.
Ссылка на запись о ферменте.
Реакция, катализируемой ферментом:
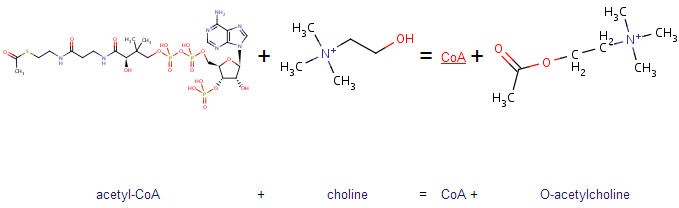
Тип реакции: перенос ацетильной группы
EC~PDB
База данных EC~PDB содержит известные структуры ферментов, которые были сданы на хранение в Protein Data Bank (PDB). Ферменты классифицируются по ЕС номеру.
Ссылка на запись о ферменте.
Название: Choline O-acetyltransferase
Другие названия: Choactase, Choline acetylase
Реакция: Acetyl-CoA + choline = CoA + O-acetylcholine
Кристаллическая структура фермента холинацетилтрансферазы у крысы (1q6x):
Другие изображения структуры из банка PDB.
EC-код:
ExploreEnz
ExplorEnz - это общедоступная, вручную курируемая и рецензируемая база данных ферментов из списка ферментов IUBMB (International Union of Biochemistry and Molecular Biology).
Ссылка на запись о ферменте.
Принятое название: choline O-acetyltransferase
Другие названия: choline acetylase; choline acetyltransferase
Систематическое название: acetyl-CoA:choline O-acetyltransferase
Комментарии: Пропионил-КоА может действовать медленнее, чем ацетил-КоА.
KEGG
KEGG - это Киотская энциклопедия генов и геномов, которая представляет собой базу данных ресурсов для понимания функций высокого уровня и частей биологической системы, таких как клетки, организм и экосистемы, особенно крупномасштабные молекулярные наборы данных, полученные секвенированием генома и другими экспериментальными технологиями.
Ссылка на запись о ферменте.
В разделе KEGG BRITE, представлен набор иерархических классификаций, представляющий наши знания о различных аспектах биологических систем. Иерархия BRITE для заданного фермента:
IntEnz
IntEnz (Integrated relational Enzyme database) - это свободно доступный ресурс, сосредоточенный на номенклатуре ферментов.
Ссылка на запись о ферменте.
Иерархическая классификация EC:
Расшифровка кода:
Класс: EC 2 - Transferases - трансферазы - отдельный класс ферментов, катализирующих перенос функциональных групп и молекулярных остатков от одного соединения (как правило, рассматривается в качестве донора) в другое соединение (как правило, рассматривается в качестве акцептора).
Подкласс: EC 2.3 - Acyltransferases - ацилтрансферазы - ферменты, переносящие ацильную группу на молекулу субстрата, образуя сложные эфиры или амиды. Донором в большинстве случаев является соответствующая ацил-коэнзим А производная.
Подподкласс: EC 2.3.1 - Transferring groups other than amino-acyl groups - ферменты, переносящие группы, отличные от амино-ацильных.
Наконец, объединим всю собранную информацию и создадим визитную карточку заданного фермента CLAT_HUMAN:

2. Поиск ферментов человека с близкими функциями (в Swiss-Prot)
Для поиска белков человека, являющихся ферментами и имеющих общие с заданным ферментом уровни классификации, воспользуемся сервисом SRS.
Поиск будем проводить по двум параметрам, меняя только EC-код (2.3.1.6):
- Species - "Human"
- ECNumber - 2.*; 2.3.*; 2.3.1.*; 2.3.1.6
Результаты поиска приведены в таблице ниже:
| EC-код | Число записей в Swiss-Prot | Параметры поиска |
| 2.*.*.* | 1600 | ([swissprot-ECNumber:2.*] & [swissprot-Species:Human]) |
| 2.3.*.* | 177 | ([swissprot-ECNumber:2.3.*] & [swissprot-Species:Human]) |
| 2.3.1.* | 153 | ([swissprot-ECNumber:2.3.1.*] & [swissprot-Species:Human]) |
| 2.3.1.6 | 1 | ([swissprot-ECNumber:2.3.1.6] & [swissprot-Species:Human]) |
3. Определение того, насколько сохраняется функция заданного фермента у белков со сходными последовательностями
I) Чтобы получить список всех белков мыши (Mus musculus) из Swiss-Prot, у которых класс EC 2 - Transferases, снова воспользуемся сервисом SRS.
На запрос ([swissprot-Species:Mouse] & [swissprot-ECNumber:2.*]) нашлось 1383 белка.
Полученные данные сохранили в fasta-формате и в виде текстовой таблицы.
Также нашли совпадения белков с порядковыми номерами EC:
- EC 2.3: 174 белка
- EC 2.3.1: 154 белка
- EC 2.3.1.6: 1 белок (CLAT_MOUSE)
II) Из базы данных UniProt взяли последовательность заданного фермента. В названиях последовательностей, полученных ранее, оставили только мнемоники, используя команду sed:
sed -e "s/sp|.*|//" -e "s/ .*//" < clat_mouse.fasta > mouse.fasta
Далее проиндексировали полученный файл и выполнили поиск гомологов CLAT_HUMAN программой blastp:
makeblastdb -in mouse.fasta -out prot -dbtype prot
blastp -query clat_human.fasta -db prot -evalue 1 -out prot_clat.out
blastp -query clat_human.fasta -db prot -evalue 10 -out prot_clat_2.out
blastp -query clat_human.fasta -db prot -evalue 0.001 -out prot_clat_3.out
Получили три файла, отличающиеся результатом только из-за порога на E-value.
Ниже приведена таблица, 11-ти белков, полученных при самом слабом пороге (-evalue 10):
| Белок | E-value | Identity | Перекрывание |
| CLAT_MOUSE | 0.0 | 87% | 92% |
| CACP_MOUSE | 4,00E-168 | 43% | 63% |
| OCTC_MOUSE | 5,00E-79 | 30% | 50% |
| CPT1A_MOUSE | 1,00E-73 | 31% | 48% |
| CPT2_MOUSE | 3,00E-72 | 29% | 48% |
| CPT1B_MOUSE | 7,00E-72 | 30% | 48% |
| CPT1C_MOUSE | 1,00E-60 | 29% | 47% |
| BRSK2_MOUSE | 0.34 | 40% | 49% |
| PKN1_MOUSE | 1.5 | 26% | 50% |
| C1GLT_MOUSE | 1.7 | 37% | 67% |
| TITIN_MOUSE | 7.9 | 52% | 61% |
Из этих белков, можно считать достоверными гомологами только первые 7 (которые получились при сильном пороге -evalue 0,001) с высоким процентом перекрывания (>45%) и низким E-value (< 10 в минус 63 степени).
III) Теперь добавим в таблицу информацию о EC-коде и сохранении функции каждого белка:
|
Белок |
E-value |
Identity |
Перекрывание |
EC-код |
Сохранение функции |
|
CLAT_MOUSE |
0.0
|
87% |
92% |
2.3.1.6 |
сохраняется полностью |
|
CACP_MOUSE |
4,00E-168 |
43% |
63% |
2.3.1.7 |
сохраняется до подподкласса |
|
OCTC_MOUSE |
5,00E-79 |
30% |
50% |
2.3.1.137 |
|
|
CPT1A_MOUSE |
1,00E-73 |
31% |
48% |
2.3.1.21 |
|
|
CPT2_MOUSE |
3,00E-72 |
29% |
48% |
2.3.1.21 |
|
|
CPT1B_MOUSE |
7,00E-72 |
30% |
48% |
2.3.1.21 |
|
|
CPT1C_MOUSE |
1,00E-60 |
29% |
47% |
2.3.1.21 |
|
|
BRSK2_MOUSE |
0.34 |
40% |
49% |
2.7.11.1 |
сохраняется только класс |
|
PKN1_MOUSE |
1.5 |
26% |
50% |
2.7.11.13 |
|
|
C1GLT_MOUSE |
1.7 |
37% |
67% |
2.4.1.122 |
|
|
TITIN_MOUSE |
7.9 |
52% |
61% |
2.7.11.1 |
Итак, сравнивая с EC-кодом CLAT_HUMAN (2.3.1.6), заметим, что у первого белка сходство вплоть до порядкового номера EC 2.3.1.6, у шести последующих белков функция сохраняется до подподкласса EC 2.3.1, у последних же 4-х сохраняется лишь класс EC 2.
Как мы видим, чем выше значение E-value, тем хуже сохраняется функция (например, у первой находки с полным сохранением функции E-value=0, плюс к этому очень большой процент перекрывания и соответственно идентичности).
| Главная | Об авторе | Учебные семестры | Проекты автора | Друзья | Ссылки партнеров | Extra | Контакты |
Mneff © 2011-2013