 8 (926) 907 94 08
8 (926) 907 94 08
Всё на свете является чудом!
Киотская энциклопедия генов и геномов
KEGG ID: Homo sapiens (human): 1103
Пути:
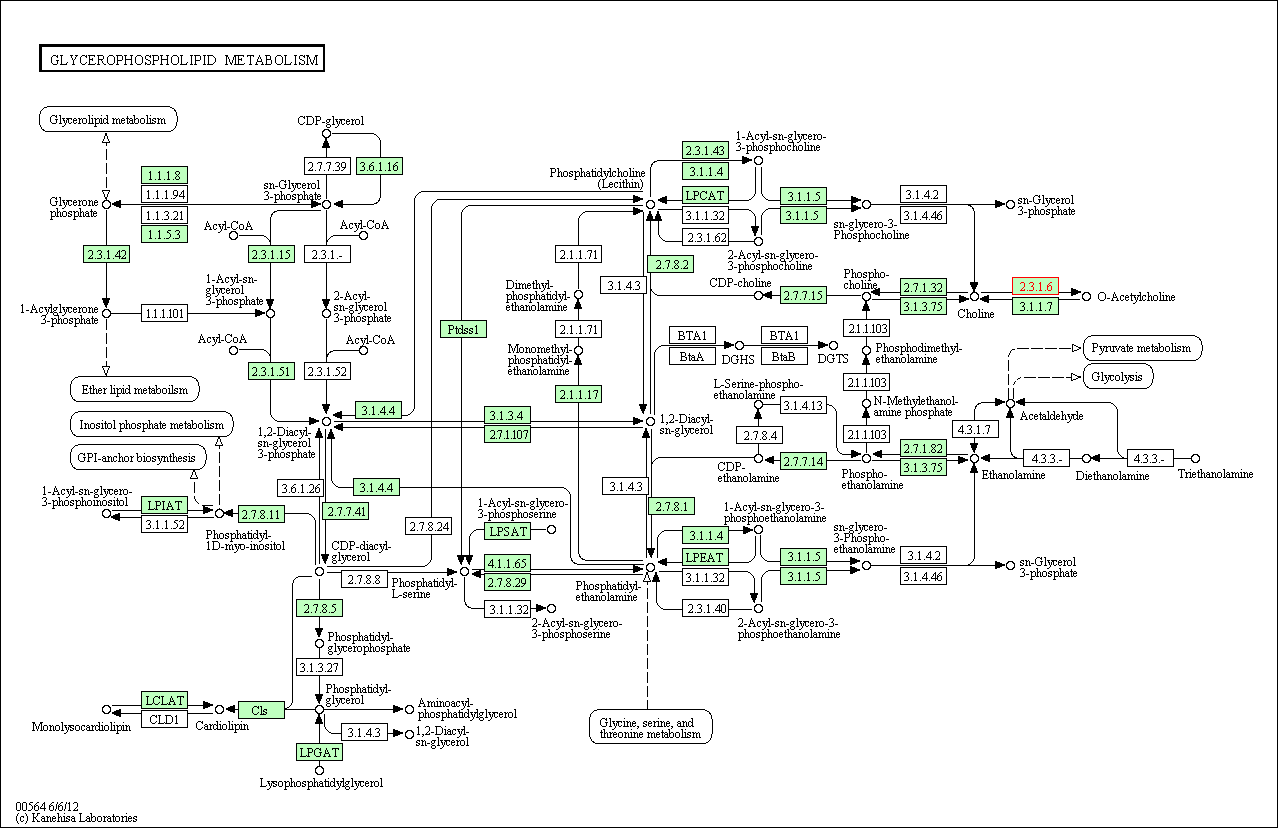
- hsa00564 Glycerophospholipid metabolism (глицерофосфолипидный метаболизм)
- hsa04725 Cholinergic synapse (холинергический синапс)
2. Поиск в KEGG структурных формул заданных соединений
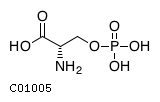
1) 0-фосфо-L-серин
O-Phospho-L-serine;
(L-O-Phosphoserine;
3-Phosphoserine;
Dexfosfoserine)
C01005
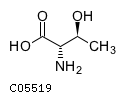
2) L-аллотреонин
L-Allothreonine
C05519
3. Поиск метаболического пути от одного заданного вещества к другому
Карта: метаболизм глицина ,серина и треонина
Путь: фосфосерин>L-аллотреонин
это единственный возможный путь на карте, он идет через серин и глицин.
Карта, с отмеченными промежуточными продуктами.
4. Сравнение метаболических путей у разных организмов
Возможность выбранной цепочки ферментативных реакций у разных организмов.
| Организм | Цепочка реакций возможна | Обоснование |
| (да/нет/неизвестно) | ||
| Bacillus subtilis | нет | из 3 нужных ферментов, есть только фермент, превращающий серин в глицин (2.1.2.1) |
| Archaeoglobus fulgidus | нет | не хватает фермента L-треонин альдолазы(4.1.2.5), катализирующего превращение глицина в аллотреонин |
| Arabidopsis thaliana | да, карта | есть все необходимые ферменты |
| Homo sapiens | нет | не хватает фермента L-треонин альдолазы(4.1.2.5), катализирующего превращение глицина в аллотреонин |
5. Мнение о KEGG
KEGG - база знаний по систематическому анализу функций генов, созданная институтом химических исследований (Kyoto University, Japan) в рамках японской программы по геному человека. Она содержит такие базы данных, как, например, метаболические пути (PATHWAY), гены (GENES) и лиганды (LIGAND), экспериментальные данные по экспрессии генов (EXPRESSION и BRITE), по белкам (SSDB) и обеспечивает обширные возможности для работы со всеми крупными мировыми информационными ресурсами. Базы данных KEGG представлены в виде графических диаграмм, включающих большинство метаболических путей и некоторые известные регуляторные пути. С одной стороны это приводит все во едино, систематизирует, но с другой, чтобы разобраться в сложной запутанной схеме требуется время. Кроме того, информация о путях представлена в виде таблиц ортологов, которые содержат как гены-ортологи, так и паралоги из различных организмов - это весьма полезно.
KEGG2 - фактически оглавление, путеводитель по ресурсу. Если сразу в
поле запрос, то в полученном списке находок можно получить необходимые
сведения о рассматриваемых белках, имена генов, ссылки на другие разделы
KEGG и вообще др. базы данных, аминокислотные и нуклеотидные
последовательности, ссылки на ортологи и паралоги, а также данные по
кластерам генов.
Можно узнать информацию о мотивах ( по данным SSDB ) конкретного белка,
а также наличие таких мотивов в других генах, посмотреть
последовательность мотива.
В поле "Position" - позиция гена в геноме; можно посмотреть карту
генома.
KEGG PATHWAY - раздел, содержащий информацию о метаболических путях (см.
полученные выше результаты ).
При поиске в этом разделе можно делать запрос и по ЕС фермента. Если
перевести карту в режим "Reference pathway (Reaction)", можно посмотреть
на уравнения реакций. И потом, чтобы определить наличие выбранной
цепочки реакций, провести "сортировку" по организму.
KEGG BRITE - документ, содержащий ссылку на BRITE. Здесь содержится описание биологических процессов в иерархическом порядке. Очень похоже на GO - фактически отражена связь между терминами для KEGG Orthology.
KEGG GENES - содержит информацию о генах и геномах, содержащихся в KEGG. Чтобы получить всю информацию, относящуюся к рассматриваемому ферменту, проводим поиск по организму и EC фермента.
KEGG LIGAND - содержит данные о ферментах. Проводим поиск поиск по
ENZYME и получаем названия фермента (поле Name), классификацию - со
ссылкой на BRITE (поле Class), систематическое название (номенклатурное)
- поле Sysname, ссылки на карты метаболическмх путей с участием фермента
(поле Pathway), уравнение реакции, катализируемой данным ферментом (поле
Reaction(IUBMB) и поле Reaction(KEGG); по ссылке на документ можно
получить больше информации о реакции) и информацию о субстратах
(исходных веществах) и продуктах реакции (для веществ приведены
структурные и брутто - формулы; список реакций, в которых они
фигурируют; список ферментов, связанных с данными веществами).
Еще есть список генов (поле Genes); в поле Structures - список 3D
структур и пр.
"организм-специфический" раздел поиска:
* - KEGG Organisms - тут можно узнать о том, информация по белкам каких
организмов приведена в БД (эукариоты + бактерии + археи); какие
трехбуквенные сокращения приняты для названий организмов.
"тематические" разделы (фактически, это "прикладные" к приведенным выше разделам):
- KEGG DRUG - часть KEGG LIGAND; содержит структурные формулы всех разрешенных (в Японии, США) лекарственных средств, подразделенных на категории.
- KEGG GLYCAN - часть KEGG LIGAND; содержит структуры (экспериментально определенные) гликанов (уникальные структуры CarbBank, структуры из последних публикаций и структуры, приведенные в KEGG pathways).
- KEGG REACTION - часть KEGG LIGAND, содержащая биохимические реакции из IUBMB Enzyme Nomenclature и из KEGG metabolic pathways.
- KAAS - KEGG - расшифровка: KEGG Automatic Annotation Server - обеспечивает функциональную аннотацию генов BLAST (в отличие от курируемой информации KEGG GENES).
Кроме всех перечисленных разделов, имеются в "Тематическом поиске"
пункты (без прямых ссылок):
KEGG DISEASE - о соединениях, связанных с болезнями человека (как
нарушения в метаболических путях связаны с различными заболеваниями
(всего 5 групп: нейродегенеративные растройства; инфекционные болезни;
болезни, связанные с нарушением метаболизма; различные виды рака));
KEGG ENVIRONMENT - раздел о метаболизме и деградации ксенобиотиков.
Приведен список доступных режимов просмотра и Java - приложений для
использования KEGG.
Таким образом, KEGG - весьма удобная, регулярно обновляющаяся БД (все очень логично связано (четкая цепь "ген - белок - функция"), обширная информация по метаболическим путям с возможностью получения информации о веществах, вовлеченных в процесс). Радуют полнота, разнообразие информации, интерфейс и скорость обработки запроса (последнее - немаловажно; надеемся, что после присоединения к SRS, KEGG не утратит этой прекрасной характеристики).
| Главная | Об авторе | Учебные семестры | Проекты автора | Друзья | Ссылки партнеров | Extra | Контакты |
Mneff © 2011-2013