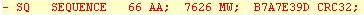8 (926) 907 94 08
8 (926) 907 94 08
Всё на свете является чудом!
Поисковые
системы
1. Поиск моего
белка в банке
SwissProt
с помощью
SRS
и описание «особенностей» в его аминокислотной
последовательности
- переходим на сайт
SRS http://srs.ebi.ac.uk/
- заходим на страницу «Library
page»,
нажав на вкладку вверху сайта
- среди доступных баз данных выбираем «UniProtKB/Swiss-Prot»,
затем слева в блоке «Search
Options»
выбираем «Standard
Query
Form»
- в одном из полей запроса выбираем «ID»
и вносим в окошко идентификатор моего белка –
THIS, после чего нажимаем на красную кнопку поиска «Search»
- из полученного списка белков выбираем мой белок –
THIS_BACSU
- переходим в раздел «Features»
вверху страницы, где выбираем произвольные три особенности (например,
альфа-спираль (она единственная в моём белке), второй бета-тяж и поворот
(он единственный в моём белке, как и альфа-спираль)).
Альфа-спираль
– образована 7 аминокислотными остатками (с 18 по 24): изолейцином (I),
глутамином (Q),
аспарагиновой кислотой (D),
двумя лейцинами (LL),
аланином (A) и
серином (S).
ID THIS_BACSU_6; parent: THIS_BACSU
FT HELIX 18 24
SQ Sequence 7 AA;
IQDLLAS
//
Второй бета-тяж
– образован 3 аминокислотными остатками (с 12 по 14): двумя лизинами (KK)
и аспарагиновой кислотой (D).
ID
THIS_BACSU_5; parent:
THIS_BACSU
FT STRAND
12
14
SQ
Sequence 3
AA;
KKD
//
Поворот
– образован 5 аминокислотными остатками (с 44 по 48): глутаминовой
кислотой (E),
аргинином (R),
тирозином (Y),
гистидином (H)
и глутаминовой кислотой (E).
ID
THIS_BACSU_9; parent:
THIS_BACSU
FT
TURN
44
48
SQ
Sequence 5
AA;
ERYHE
//
2. Получение и
сохранение описания всех записей банка трёхмерных структур
PDB, относящихся к моему белку
-
находясь на странице с информацией о белке, в левой части страницы в
блоке «Entry
Options»
нажимаем на красную кнопку «Link»,
после чего переходим на страницу находок
-
выбираем раздел «Protein
function,
structure
and
interaction
databases»,
в котором отмечаем галочкой базу данных «PDB».
Затем нажимаем на красную кнопку «Search» в верхней левой части страницы.
-
получив список документов
PDB (в моём
случае это всего один документ), нажимаем на красную кнопку «Save» в графе «Result
Options»
в верхней левой части страницы
-
перейдя на страницу параметров сохранения информации, отмечаем «File (text)»
в блоке «Output
To:»,
после чего нажимаем на красную кнопку «Save» в нижней правой части страницы.
Таким образом, сохранив полученную информацию, мы получаем файл
pdb_info_THIS_BACSU.txt,
в котором записана информация о всех записях, которые были в «находках»
(в моём случае всего одну запись
PDB:1TYG).
3. Поиск
полноразмерных белков из
Firmicutes, выполняющих функцию,
сходную с функцией моего белка
- в
одном из полей запроса выбираем «Taxonomy»
и вносим в окошко «Firmicutes»
- в
следующем поле выбираем «Comments:
Comment» и
вносим в окошко описание функций или отдельные слова из описания,
которые собираемся искать
- нажимаем на красную кнопку
поиска «Search»
- для поиска только
полноразмерных последовательностей выбираем в новом поле запроса «Description»
и записываем в окошко «*!fragment»
Таблица встречаемости
в
SwissProt
белков
из Firmicutes,
имеющих
функцию,
сходную
с
функцией
белка
THIS_BACSU
из
Bacillus subtilis
|
Формулировка функции
белка |
Строка запроса |
Количество найденных
документов |
|
Запрос всех
последовательностей |
||
|
Переносчик
серы к триазолу
(sulfur & transfer & to
& thiazole) |
([swissprot-Taxonomy:Firmicutes*]
& (((([swissprot-Comment:sulfur*]
& [swissprot-Comment:transfer*])
& [swissprot-Comment:to*])
& [swissprot-Comment:thiazole*])
> parent )) |
126 |
|
Донор серы в синтезе
триазола
(sulfur & donor & in &
the & synthesis & of &
the & thiazole) |
([swissprot-Taxonomy:Firmicutes*]
&
(((((((([swissprot-Comment:sulfur*]
&
[swissprot-Comment:donor*])
& [swissprot-Comment:in*])
&
[swissprot-Comment:the*])
&
[swissprot-Comment:synthesis*])
&
[swissprot-Comment:of*])
& [swissprot-Comment:the*])
&
[swissprot-Comment:thiazole*])
>
parent )) |
1 |
|
Упоминание в описании
функции «переносчика
серы»
(sulfur;
transfer) |
(([swissprot-Taxonomy:Firmicutes*]
&
([swissprot-Comment:sulfur*]
>
parent )) &
([swissprot-Comment:transfer*]
>
parent )) |
260 |
|
Запрос только
полноразмерных
последовательностей |
||
|
Переносчик серы к
триазолу
(sulfur & transfer & to
& thiazole) |
(([swissprot-Taxonomy:Firmicutes*]
&
(((([swissprot-Comment:sulfur*]
&
[swissprot-Comment:transfer*])
& [swissprot-Comment:to*])
&
[swissprot-Comment:thiazole*])
>
parent )) &
([swissprot-Description:*]
!
[swissprot-Description:fragment*])) |
126 |
|
Донор серы в синтезе
триазола
(sulfur & donor & in &
the & synthesis & of &
the & thiazole) |
(([swissprot-Taxonomy:Firmicutes*]
&
(((((((([swissprot-Comment:sulfur*]
&
[swissprot-Comment:donor*])
& [swissprot-Comment:in*])
&
[swissprot-Comment:the*])
&
[swissprot-Comment:synthesis*])
&
[swissprot-Comment:of*])
& [swissprot-Comment:the*])
&
[swissprot-Comment:thiazole*])
>
parent )) &
([swissprot-Description:*]
!
[swissprot-Description:fragment*])) |
1 |
|
Упоминание в описании
функции «переносчика
серы»
(sulfur; transfer) |
((([swissprot-Taxonomy:Firmicutes*]
&
([swissprot-Comment:sulfur*]
>
parent )) &
([swissprot-Comment:transfer*]
>
parent )) &
([swissprot-Description:*]
!
[swissprot-Description:fragment*])) |
260 |
4. Сохранение
полноразмерных последовательностей найденных белков в fasta формате
-
вернулись к первому запросу (из таблицы в задании 3)
- выбрали 10 находок из
имеющихся 126 и нажали на красную кнопку «Save»
из блока «Result
Options»
- на открывшейся странице
параметров сохранения в разделе «Output
To»
отмечаем «File
(text)», а в
разделе «Save
as»
вместо «UniprotView»
выбираем «FastaSeqs»
-
нажимаем на красную кнопку «Save»
в нижней правой части страницы
-
переименовываем сохранённый файл в файл
similar_fasta_files_THIS_BACSU.fasta
и получаем файл с последовательностями в
fasta-формате
-
вернулись к первому запросу (из таблицы в задании 3)
- в появившемся списке последовательностей отметили те, которые были сохранены в fasta-формате, после чего нажали на красную кнопку «Save» в блоке «Result Options» в левой части страницы
- на открывшейся странице с параметрами сохранения в разделе «Output To» отмечаем «File (text)», а в разделе «Save As» выбираем «SWISSPROT»
-
нажимаем на красную кнопку «Save»
в нижней правой части страницы
- переименовываем сохранённый файл в файл table_of_similar_fasta_files_THIS_BACSU.xls и получаем плоскую таблицу
6. Описание изменений, произошедших с записью моего белка от первого её появления в банке Uniprot до последней версии- Для выполнения задания, воспользуемся сервисом expasy.org
- В верхней части страницы в комбобоксе "query" выбираем БД Uniprot, а в поле запроса "query text" вписываем идентификатор моего белка (THIS_BACSU). Затем осуществляем поиск, нажав на кнопку "search".
- В результате поиска получаем 1 результат с полной записью, поэтому переходим по ссылке "History".
- Перейдя по ссылке "History", попадаем на страницу со списком документов всех версий о моём белке.
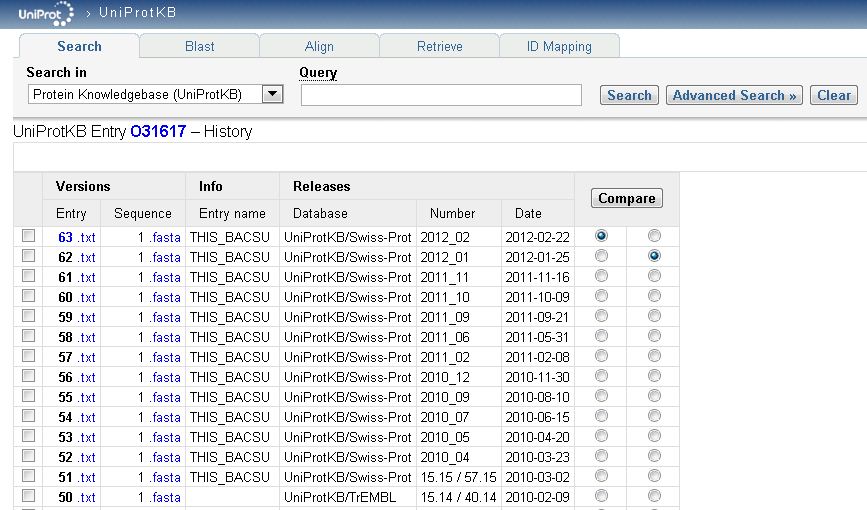
- Для сравнения выбираем 1 и 63 (последнюю) версии, поставив маркеры в правой части таблицы. После этого нажимаем на кнопку "Compare", чтобы сравнить эти записи.
Результат сравнения представлен в виде таблицы со скрин-шотами
|
|
Скрин-шоты |
||
|
Дата создания |
01-JAN-1998 |
22-FEB-2012 |
|
|
Идентификатор
ID |
THIS_BACSU |
THIS_BACSU |
|
|
Идентификатор
AC |
O31617 |
O31617 |
|
|
Изменения в
последовательности |
SEQUENCE 66 AA; 7626 MW;
B7A7E39D CRC32; |
SEQUENCE
66 AA;
7626 MW;
476D61DCC98BAADE
CRC64;
MLQLNGKDVK WKKDTGTIQD LLASYQLENK IVIVERNKEI IGKERYHEVE LCDRDVIEIV HFVGGG |
|
|
Поле
KW |
- |
3D-structure; Complete
proteome;
Nucleotide-binding;
Reference proteome;
Thiamine biosynthesis. |
|
|
Поле
FT |
- |
12 строчек |
|
|
Поле
DR |
EMBL; Z99110; E1183188;
-. |
EMBL; AL009126;
CAB13025.1; -;
Genomic_DNA.
+ 29 строчек |
|
От момента первого появления записи о моём белке прошло 14 лет. Со времени экономического кризиса 1998 года (год 1 версии) до выбора Президента В.В.Путина в 2012 году (год 63 версии) многое изменилось как в мире, так и в информации о моём белке. Про изменения в мире я думаю не стоит рассказывать, а вот про белок скажу пару слов.
Идентификаторы ID и AC моего белка остались неизменными; появились такие поля как "Keywords" и "Feature Table", которые, на мой взгляд, несут в себе немало полезной информации. За время 63-х версий количество строчек в записи изменилось с 55-ти до 133-х. Так что теперь мы знаем о моём белке как минимум в два раза больше информации.
7. Поиск и изучение страницы с описанием поля Taxonomy банка Swissprot
- перешли на сайт SRS http://srs.ebi.ac.uk/
- нажали на вкладку "Library Page" вверху страницы, затем щёлкнули по названию банка "UniProtKB/Swiss-
Prot". Открылась страница с описанием банка. В нижней части этой страницы имеется список полей, пройдём по
ссылке "Taxonomy" из этого списка.
- Пользуясь возможностями, предоставляемыми этой страницей, отвечаем на вопросы
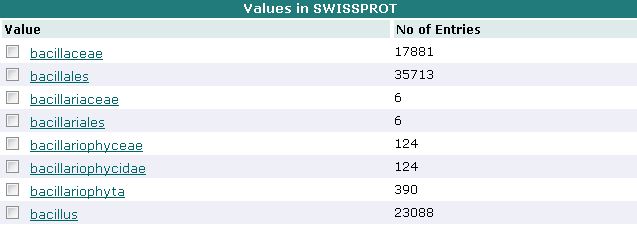
Вопрос 1. Названия каких таксонов начинаются на "bacil"?
Вносим в строку поиска bacil* и нажимаем на красную кнопку "List values". В результате получаем названия
8-ми таксонов:
Вопрос 2. Сколько в банке Swissprot записей, описывающих белки из рода Bacillus?
Вносим в поисковую строку bacillus и осуществляем поиск, нажав на красную кнопку "List values". Получаем
23088 записей:

Вопрос 3. Сколько в банке Swissprot записей, описывающих белки из отдела Firmicutes?
Вносим в поисковую строку firmicutes и нажимаем на красную кнопку "List values" для поиска. Получаем 68036
записей:

8. Описание особенностей сервиса MRS по сравнению с SRS
- открываем сайт mrs.cmbi.ru.nl
- в комбобоксе "Search" выбираем Swiss-prot, а в окошке поиска "for" записываем идетификатор моего белка
id:this_bacsu
- попав на страничку с описанием моего белка, нажимаем на кнопку "Find similar" вверху страницы
- нам представляется список 15-ти записей из 123577. Записи упорядочены по уменьшению актуальности, чем больше "Relevance" у белка, тем выше он будет по списку.
- при необходимости можно загрузить интересующие нас записи, отметив их галочкой. После чего появится кнопка "Download", нажав на которую мы можем выбрать Plain text или FastA формат для сохранения.
Правда в загрузке есть один нюанс, мы можем загрузить только те записи, которые находятся в данный момент на странице, так как, выделив их и перейдя на следующую страницу, кнопка "Download " исчезает. Это конечно минус, но, на мой взгляд, незначительный. В целом MRS пользоваться так же удобно, как и SRS.
9. Поиск записей SwissProt, связанных с научными интересами известного российского (советского) биолога А.А.Нейфаха
- перешли на сайт SRS http://srs.ebi.ac.uk/
- заходим на страницу "Library page", нажав на вкладку вверху сайта
- среди имеющихся баз данных выбираем "UniprotKB/Swiss-Prot", затем слева в блоке "Search Options" выбираем "Standard Query Form"
- в одном из полей запроса выбираем "References: Authors" и вносим в окошко Neyfakh,A.A.
- из списка возможных полей вывода выбираем: Description; Refefrences:Title; Refefrences:Authors; Refefrences:Year и нажимаем на красную кнопку "Search" вверху страницы
- получаем 12 записей, из которых отмечаем те, в которых Нейфах является одним из авторов самой первой публикации, тем самым сокращаем количество записей до 9-ти
- нажимаем на красную кнопку "Save" в блоке "Result Options" в левой части страницы, чтобы сохранить выбранные записи
- на открывшейся странице с параметрами сохранения в разделе "Output To" отмечаем "File (text)", а в разделе "Save As" выбираем "SWISSPROT", затем нажимаем на красную кнопку "Save" в нижней правой части страницы
- переименовываем сохранённый файл в файл neyfaxh.xls и получаем плоскую таблицу
Открыв и изучив файл с таблицей, приходим к выводу, что Нейфах А.А. изучал геном бактерии Bacillus
subtilis, а также был одним из авторов статей на тему генетического контроля синтеза белков.
10. Описание нескольких наиболее существенных отличий в формате записи моего белка в БД Proteins на сайте
NCBI от формата Uniprot
NCBI предоставляет информацию о базах данных белковых доменов, ДНК (GenBank) и РНК, базах данных статей научной литературы (PubMed) и таксономичной информации (TaxBrowser), обеспечивает поиск данных о конкретном биологическом виде (Taxonomy). Также содержит различные стандартные программы биоинформатики (BLAST). Базы данных доступны через поисковую систему Entrez.
- для сравнения NCBI и Uniprot перейдём на их соответствующие сайты, где осуществим поиск записи о моём белке. Как это делать в Uniprot описано в практикуме "Protein in Uniprot", а чтобы искать в NCBI выбираем в комбобоксе "Protein" и пишем в поисковой строке Uniprot идентификатор белка - THIS_BACSU
- теперь, можем сравнить формат записи в БД Protein на сайте NCBI с форматом Uniprot
В общем эти два формата похожи, но всё же есть некоторые различия в структуре и оформлении полей.
На NCBI появляется поле "LOCUS" с идентификатором и датой последнего изменения белка вместо поля ID формата Uniprot. Также на NCBI последовательность записывается в поле "Origin", а на Uniprot в поле "SQ". Нетрудно заметить, что названия полей также изменились, стали писаться полностью (Features вместо FT, например). На NCBI в отличие от Uniprot есть различные режимы работы (GenPept, Fasta и Graphics) и в некоторых полях доступно больше информации.
Но лично мне удобнее работать с Uniprot, где нет ничего лишнего и всё чётко структурировано.
| Главная | Об авторе | Учебные семестры | Проекты автора | Друзья | Ссылки партнеров | Extra | Контакты |
Mneff © 2011-2012