Применение BLAST для поиска сходства нуклеотидных последовательностей
Ранее мной была получена консенсусная последовательность для некоторых двух прочтений, полученных методом Сэнгера. Пришло время разобраться, какова функция участка ДНК, из которого эти риды были получены, и каково систематическое положение организма, из которого был взят образец
Выбор варианта BLAST
Очевидно, что нас интересуют нуклеотидные варианты, так как мы работаем с последовательностью ДНК. Варианты с трансляцией также не подходят, ведь функция участка ДНК неизвестна. MEGAblast ищет только очень близкие гомологи, поэтому мне кажется наиболее эффективным использовать классический blastN
Я использовал дефолтные настройки blastN, получив следующую выдачу. Значения e-value топовых 20 находок достаточно маленькие (менее 10-110), и практически во всех находках так или иначе фигурирует субъединица 1 митохондриальной цитохром-С-оксидазы (либо цитохром-С-оксидаза, либо митохондриальный геном). Отсюда можно сделать вывод, что риды были получены с участка гена первой субъединицы митохондриальной цитохром-С-оксидазы, и далее воспользоваться TblastX для поиска по транслированным базам для более точного поиска организма-хозяина
В TblastX я также использовал параметры по умолчанию, только генетический код для Invertebrate mitochondria; далее отобрал 21 находку с e-value порядка не выше 10-60 и рассмотрел их таксономию. Все находки попали в Lophotrochozoa, однако внутри распределились следующим образом:
-9, обладающих наилучшими score, попали в Entoprocta
-3 попали в Mollusca
-13 попали в Heteronemertea
(в сумме получается более 21, так как одна находка может выровняться на разных участках)
Отсюда можно сделать вывод, что риды получены при секвенировании митохондрий представителя Lophotrochozoa, вероятнее всего, из Entoprocta
Я выбрал 32-й контиг из сборки Psilocybe cubensis, описанной в предыдущем практикуме
В выбранном контиге находится 3 участка, помеченных как гены предполагаемых белков. Я решил взять ген с координатами 11563-14382, про который также написано, что он является трансмембранным
Так как нам необходимо найти сходные с продуктом гена белки, ген нужно транслировать и искать совпадения в базе данных белков, то есть воспользоваться blastX
В blastX я использовал базу данных SwissProt (чтобы ускорить поиск и найти аннотированные белки) и длину слова 3 (чтобы найти схожие белки в отдалённых организмах), получив при этом следующую выдачу
Все находки содержат α-субъединицы ГТФ-связывающих белков, что вкупе с информацией о том, что белок является трансмембранным, позволяет предположить, что это α-субъединица GPCR
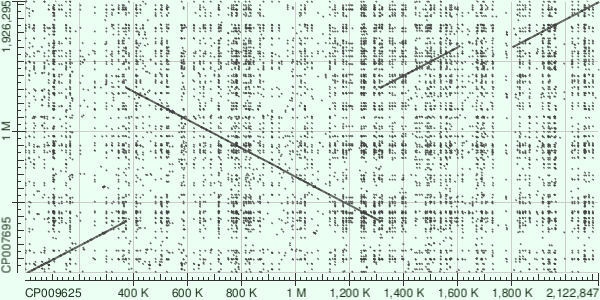
Я выбрал для сравнения первые хромосомы Brucella abortus str 104M (CP009625) и Brucella suis str Bs143CITA (CP007695)
В качестве варианта blast2seq, на мой взгляд, лучше всего подходит blastN с длиной слова 15, так как поиск выполняется по близкородственным видам, по всем участкам ДНК (а не только белок-кодирующим)
Эволюционные события (для Brucella suis относительно Brucella abortus):
-инверсия участка 350К-1300К
-делеция участка в позиции 1600К, который у Brucella abortus 1600К-1800К (или же вставка соответствующего участка в Brucella abortus)