| Элемент структуры | find_pair (3DNA) | einverted (EMBOSS) | Алгоритм Зукера |
|---|---|---|---|
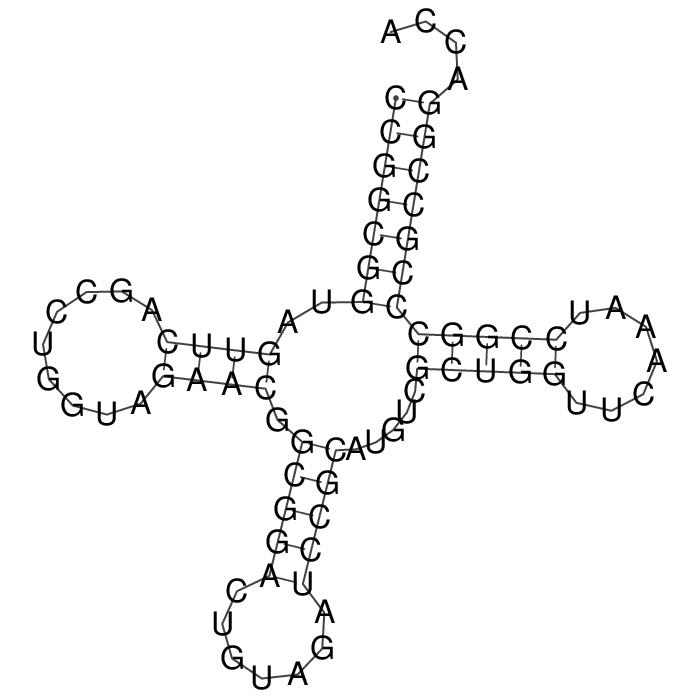
| Акцепторный стебель | 501-507,567-573 | 501-507,567-573 | 501-507,567-573 |
| D-стебель | 510-513,523-526 | - | 510-513,523-526 |
| Т-стебель | 550-554,562-566 | - | 550-554,562-566 |
| Антикодоновый стебель | 527-534,537-545 | - | 528-532,540-544 |
| Общее число канонических пар нуклеотидов | 21 | 7 | 21 |
Я написал скрипт для PyMOL, однако в нём проблема с исполнением команды time.sleep(); PyMOL, в отличие от Jupiter, исполняет все паузы в первую очередь. Вариант с использованием пустого input() выдаёт ошибку :(
Аналогично, для подсчёта контактов я использовал другой скрипт для PyMOL, он выдаёт подобный текстовый файл. Кривой способ подсчёта через длину списка я использовал, так как обычное выражение "count+=1" для числового счётчика в iterate PyMOL игнорирует
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 0 | 9 | 9 |
| остатками фосфорной кислоты | 9 | 10 | 19 |
| остатками азотистых оснований со стороны большой бороздки | 6 | 4 | 10 |
| остатками азотистых оснований со стороны малой бороздки | 6 | 10 | 16 |
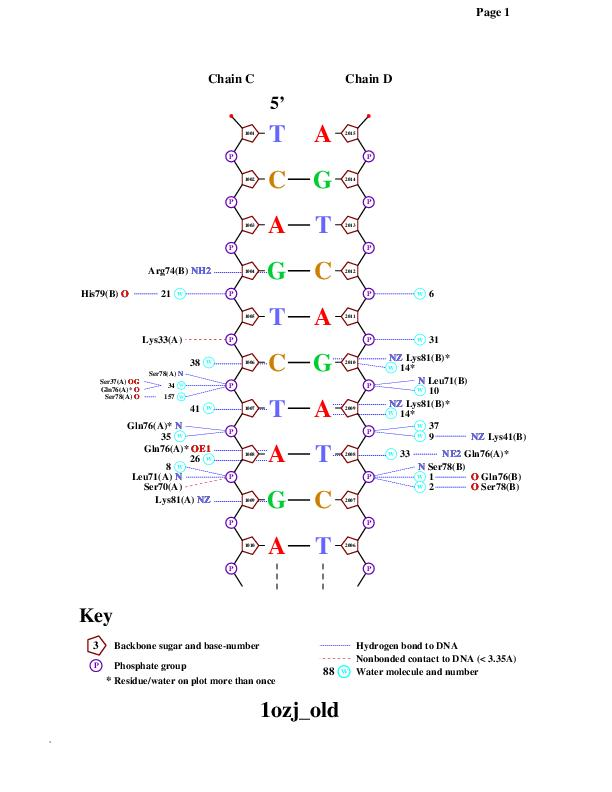
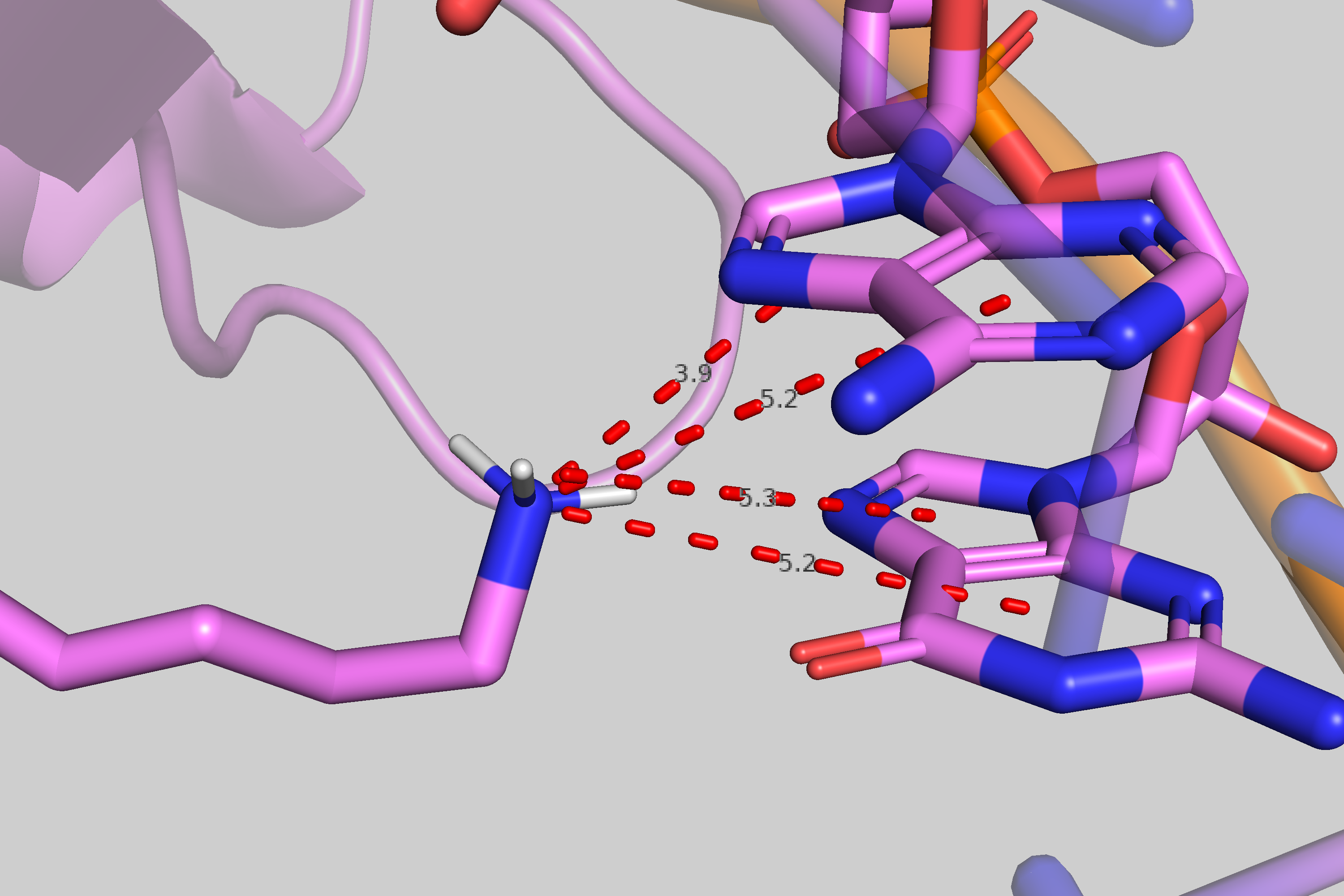
Исходя из полученной схемы, наибольшее число контактов с ДНК имеет K81 из цепи B, а именно его атом NZ. Какие именно взаимодействия он осуществляет, не вполне понятно: отрицательно заряженных групп в близлежащих азотистых основаниях нет, а для водородных связей, на мой взгляд, не подходит геометрия. Я предполагаю, что между пуриновыми кольцами A и G создаётся отрицательно заряженная область делокализованной π-орбитали, к которой и притягивается близко расположенная аминогруппа боковой цепи лизина
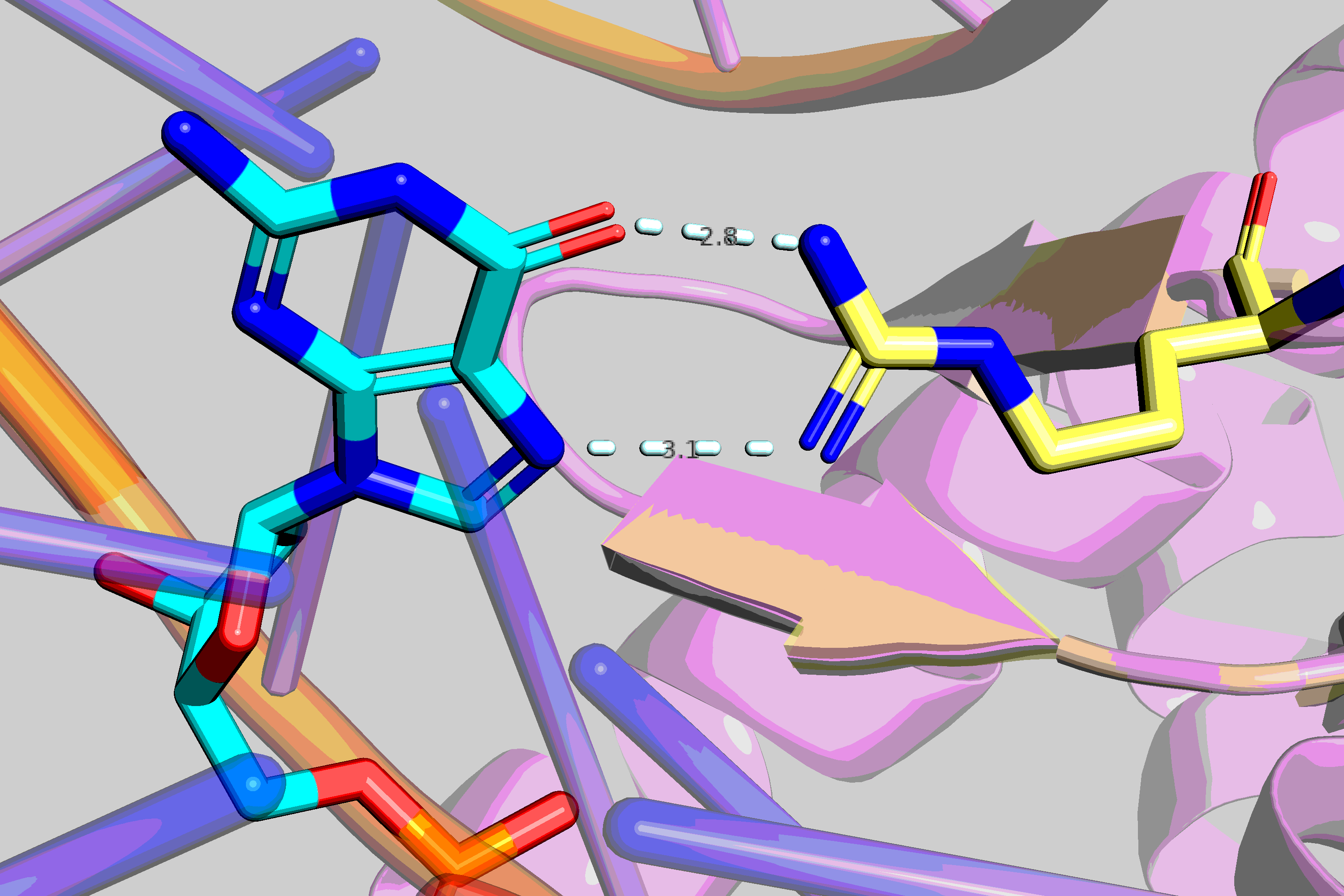
Самым важным для связывания мне показался R74 из цепи B. Его гуанидиновая группа расположена практически в плоскости кольца гуанина, тем самым он потенциально может образовать в протонированном состоянии две водородные связи (см. рисунок 2)