Построение дерева гомологов белка CLPX_ECOLI
Для запуска локального BlastP использовалась база данных белков предоставленных бактерий, порог на e-value 0,001. Выдача blastP включала в себя находки из SwissProt и TrEMBL (выдача без лишних деталей); чтобы выделить их последовательности из UniProt, я использовал скрипт на Python. Получился следующий список последовательностей
Скобочная формула дерева, полученного методом UPGMA в Mega 11
Ортологи:
CLPX MYCLE и CLPX MYCTU
CLPX COREF и CLPX CORDI
CLPX THEFY и CLPX STRAW
Паралоги:
RUVB BIFLO и CLPX BIFLO
FTSH MYCTU и CLPX MYCTU
FTSH MYCLE и CLPX MYCLE

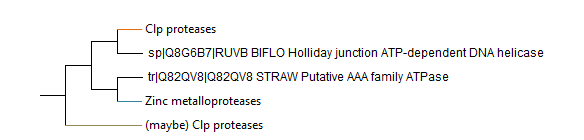
В полученном дереве можно выделить три большие группы ортологов. Первая - АТФ-связывающие субъединицы протеазы Clp (все, кроме одного, из SwissProt); вторая - АТФ-зависимые цинковые металлопротеазы; третья - разношёрстно аннотированные белки, в общем и целом похожие по описаниям на какие-то части Clp-протеаз, все из TrEMBL. Довольно странно, что они образуют отдельную от первой ветвь; возможно, это псевдогены (так как в TrEMBL зачастую попадают предсказанные по гомологии белки, а некоторые белки из этой ветви являются паралогами белков из SwissProt)
Филогения бактерий предсказывается неплохо, но только по находкам из SwissProt. При этом корректно сближенными оказались все пары организмов кроме BIFLO и LEIXX